遗传算法求解一元函数最大值
基于遗传算法求解一元函数最大值python
- 一、问题介绍
- 二、算法设计
-
- 2.1 类数据成员
- 2.2 初始化种群
- 2.3 转换种群编码
- 2.4 适应度计算
- 2.5 种群选择
- 2.6 种群交配
- 2.7 基因变异
- 2.8 种群进化
- 2.9 主函数
- 三、实验结果
-
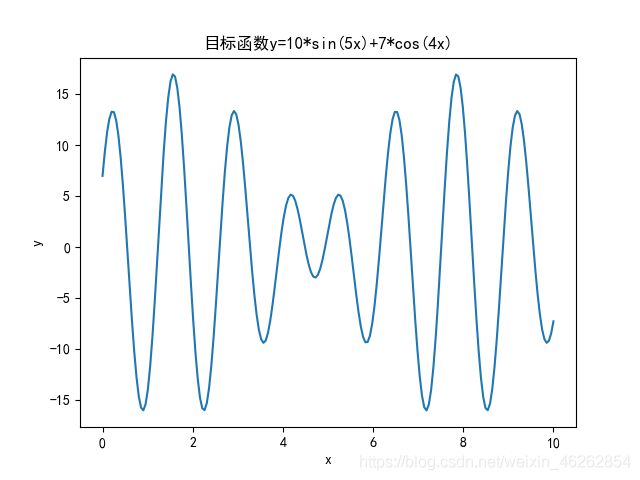
- 3.1 目标函数图像
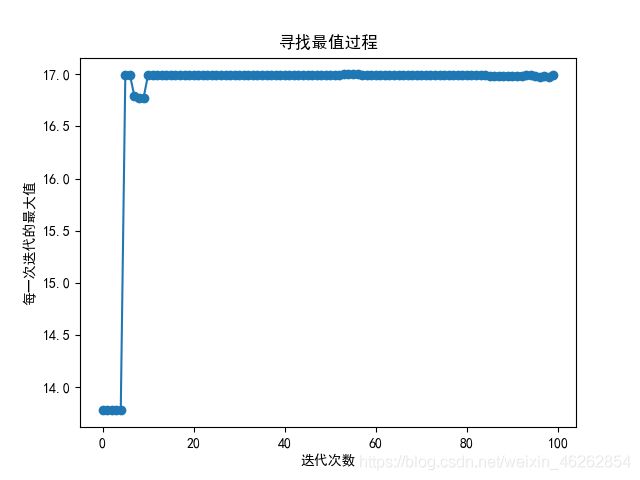
- 3.2 寻找最优值过程
- 四、总结
一、问题介绍
用遗传算法求解 f ( x ) = 10 ∗ s i n ( 5 x ) + 7 ∗ c o s ( 4 x ) , x ∈ [ 0 , 10 ] f(x)=10*sin(5x)+7*cos(4x),x\in[0,10] f(x)=10∗sin(5x)+7∗cos(4x),x∈[0,10]上的最大值。
二、算法设计
2.1 类数据成员
为了可以更好的使用遗传算法,因此定义了一个Solve类,将一些基本操作进行封装,提高代码的通用性和可读性,Solve类主要包括种群数量、迭代次数、每条染色体的基因、交配的概率、基因变异的概率、最大的函数值以及最大函数值所对应的自变量。
'''
Solve类说明
m_population: 种群数量
m_iterations: 迭代次数
DNA_size: 每条染色体的基因个数,越大,则精度越好
m_copulation: 交配的概率
m_mutation: 变异的概率
best_value: 最大的函数值
max_x: 最大的函数值所对应的自变量
'''
class Solve:
def __init__(self, m_population, m_iterations, DNA_size, m_copulation, m_mutation):
self.m_population = m_population
self.m_iteration = m_iterations
self.DNA_size = DNA_size
self.m_copulation = m_copulation
self.m_mutation = m_mutation
self.best_value = np.zeros(self.m_iteration)
self.max_x = np.zeros(self.m_iteration)
2.2 初始化种群
种群初始化主要是随机生成二进制染色体,输入形参有种群的数量以及每条染色体上基因的个数,作为第一代种群。例如基因数为5时,就随机生成10010类似的二进制数据,最后生成一个二维数组,行代表不同种群,列代表每一个种群染色体上面的基因。
def init_population(self): # 初始化种群
population = np.random.randint(low=0, high=2, size=(self.m_population, self.DNA_size))
return population
2.3 转换种群编码
种群的编码方式有很多,但是在计算函数值时,需要将不同进制的编码转换为十进制,进行编码的格式化,这样才可以进行函数值的计算。具体是将每一行作为一个二进制数,然后将其转化为十进制,比如说10010,转化为十进制就是18,然后再将该数进行自变量区间的归一化,在本题中自变量的区间范围是[0, 10],转化公式为: n u m = m i n + ( m a x − m i n ) ∗ m 2 n − 1 num=min+\frac{(max-min)*m}{2^n-1} num=min+2n−1(max−min)∗m,转化之后的结果约为5.806。
def transformDNA(self, population, min_value, max_value): # 把基因二进制变为十进制
a = np.zeros(self.m_population) # 输入形参为种群,转化为十进制的最小值和最大值,对应自变量的范围
for i in range(self.m_population):
for j in range(self.DNA_size):
a[i] += population[i][j]*math.pow(2, j)
# print(a[i])
a[i] = min_value+(max_value-min_value)*a[i]/math.pow(2, self.DNA_size)
# print(a[i])
return a
2.4 适应度计算
适应度函数是遗传算法寻优的关键要素,是连接算法与测试用例生成问题的桥梁,通过适应度函数可以评估相应启发式搜索算法输入参数的性能优劣。本算法中以目的函数为基础,稍加修改作为适应度函数。目的是为了保证计算得到的适应度值都是非负的,因为在进行种群选择时利用的是轮盘赌法,要求每一个值都是非负的。
def fitness(self, a): # 适应度函数,第一个形参是输入的数组,每个元素是十进制
population_value = a
fitness_value = 10 * np.sin(5 * population_value) + 7 * np.cos(4 * population_value)
return fitness_value + 20 # 为了轮盘选择时,每个概率都是正的,因此要把适应度值做适当修改
2.5 种群选择
为了使得后代更加优良,采用达尔文进化原理“适者生存,不适者淘汰”,以轮盘赌法选取优良的种群,适应度值越大,基因越好,被选择的概率越大,并且每次在选择时可以选择相同的种群,即每一次选择时,各个种群都可能被选中,选中的概率是该种群的适应度值除以全体种群适应度值的总和。
def select(self, population, fitness_value): # 适应度选择,选择适应度最大的函数值 形参是种群和适每个种群的适应度
bingo = np.random.choice(np.arange(self.m_population), size=self.m_population, replace=True, p=fitness_value/fitness_value.sum())
# print(bingo) # 从第一个形参中取出数据(必须是一维数组) 组成size的数组;replace为True时,可以选择重复的数据 为假时,不可以选择重复的数据;p为选择数组中每个元素的概率,默认为一样
return population[bingo]
2.6 种群交配
两个种群进行交配,产生新的种群,本算法中是先随机对种群进行两两配对,然后对每一对以交配概率m_copulation进行交配,即生成一个(0,1)之内的随机数,判断该随机数是否小于交配概率,若小于交配概率,则进行交配;否则不交配,判断下一对染色体。在交配时选取的位置也是随机的,即生成一个(0,DNA_SIZE)的随机数m,交换两个染色体中[0,m]的片段。每一次交配的点都是不同的,这样可以更好的模拟自然界的规律,也更容易寻找各个解。
def copulation(self, last_generation, new_generation): # 交配 两个染色体交配生成一个新的染色体,第一个形参和第二个形参是父代染色体
match = np.random.choice(np.arange(self.m_population), size=self.m_population, replace=False) # 将染色体排序,随机选择两个染色体进行交配
for i in range(self.m_population//2): # 在python3中要用// 否则是浮点数
if np.random.rand() < self.m_copulation:
loc = np.random.randint(low=0, high=self.DNA_size) # 选出两个染色体交换的点的位置
temp = last_generation[match[i]][:loc]
last_generation[match[i]][:loc] = new_generation[match[self.DNA_size-i-1]][:loc]
new_generation[match[self.DNA_size-i-1]][:loc] = temp
return new_generation
2.7 基因变异
自然界中普遍存在着基因变异,基因变异有好有坏,有可能会产生一个崭新的种群,也有可能毁掉一个种群,在遗传算法中为了模拟自然界中的变异,因此有变异概率这一因子,在本算法中是对每一个基因以变异概率m_mutation,进行变异,希望跳出局部最优解,找到全局最优解,即对每一个种群染色体的每一个基因进行遍历判断,生成一个(0,1)之内的随机数,与变异概率进行比较,若该数小于变异概率,则该基因值变为:1-原来的值(仅适用于二进制,例如原来是0,1-0即为1;原来是1,即1-1为0);若不小于变异概率,则进行下一个基因的判断。
def mutation(self, new_generation): # 变异 输入的形参种群,以变异的概率对每一个基因进行变异操作
for i in range(self.m_population):
for j in range(self.DNA_size):
if np.random.rand() < self.m_mutation:
new_generation[i][j] = 1-new_generation[i][j]
return new_generation
2.8 种群进化
经过种群间交配和变异之后,就形成了种群的进化,此函数是模拟种群整个进化过程,首先是进行种群的初始化,然后将种群的基因编码方式格式化,统一成十进制,这样就可以计算种群的适应度,以轮盘赌的方式来选择适应度最高的种群,然后将选择之后的种群进行交配和变异,依次为循环,不断选择适应度最高的种群,然后进行交配和变异,最后选择最优种群,即适应度值最大的种群。
def evolution(self): # 进化 即将种群进行初始化,然后进行适应度计算,选择最好的适应度种群,之后进行交配和变异
max_population = self.init_population() # 种群的初始化
transform = self.transformDNA(max_population, min_value=0, max_value=10) # 将二进制基因变为十进制自变量进行计算
fitness_value = self.fitness(transform) # 计算适应度函数 输入形参是转换之后的十进制数
# max_value = np.zeros(self.m_iteration)
# max_x = np.argmax(fitness_value)
# max_population = fitness(population[max_x])
for i in range(self.m_iteration): # 迭代次数
# max_x = population[np.argmax(fitness_value)]
# print(fitness(population[max_x]))
population_selects = self.select(max_population, fitness_value) # 选择
population_selects_copy = population_selects.copy() # 将父代进行复制
population_copulation = self.copulation(population_selects, population_selects_copy) # 父代交配
population_mutation = self.mutation(population_copulation) # 变异
transform = self.transformDNA(population_mutation, min_value=0, max_value=10) # 二进制变为十进制
fitness_value = self.fitness(transform) # fitness的形参必须先转化为10进制
max_xx = np.argmax(fitness_value) # 返回函数值最大的元素的索引
max_x = transform[max_xx] # 最大的十进制数,即自变量
max_population = population_mutation # 最好的种群
best_population = fitness_value.max() # 取每一次迭代的最好的种群所计算的适应度
self.best_value[i] = best_population-20 # 之前+20,在这里要减去20 每一次迭代的值都放到一个数组里面
self.max_x[i] = max_x # 把每一次的值都放到一个数组里面
print(best_population)
2.9 主函数
def main():
ga = Solve(m_population=50, m_iterations=100, DNA_size=40, m_copulation=0.5, m_mutation=0.01) # 初始化种群参数
ga.evolution() # 进化
# #########绘图#############
x = np.linspace(0, 10, num=200)
# 解决中文显示问题
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
plt.figure(1)
plt.plot(x, ga.fitness(x)-20) # 原函数图像
plt.title('目标函数y=10*sin(5x)+7*cos(4x)')
plt.xlabel('x')
plt.ylabel('y')
plt.figure(2)
ga_x = np.arange(ga.m_iteration)
plt.plot(ga_x, ga.best_value, marker='o') # 每一次迭代计算的函数值
plt.title('寻找最值过程')
plt.xlabel('迭代次数')
plt.ylabel('每一次迭代的最大值')
plt.show()
print('------------------------------------------- ga------------------------------------------------')
print('经过', ga.m_iteration, '次迭代之后,目标函数 y=10*sin(5x)+7*cos(4x)的最大值为:', ga.best_value.max())
print('--------------------------------------------ga-------------------------------------------------')
三、实验结果
3.1 目标函数图像
3.2 寻找最优值过程
四、总结
本算法主要设计了一个Solve类,将各个函数进行封装,可以将各个遗传过程通用化,提高代码的利用率,还可以为了适用不同的场景,修改类中相应的函数,文献[3]针对分区编码方法的不足,设计了一种改进的编码方式。该算法的编码方法基于组件的处理顺序和组件的组装关系。而且还设计了一种基于邻接矩阵的方法来修复不可行染色体,通过与分区编码的对比,最后证明了该算法的优越性。通过修改遗传算法不同的部分,比如说适应度函数、选择种群的方式、操作算子、交配的方法以及融合其他优化算法等等,从而改进遗传算法,提高算法的精度和收敛速度。