常用激活函数activation function(Softmax、Sigmoid、Tanh、ReLU和Leaky ReLU) 附激活函数图像绘制python代码
激活函数是确定神经网络输出的数学方程式。
激活函数的作用:给神经元引入了非线性因素,使得神经网络可以任意逼近任何非线性函数。
1、附加到网络中的每个神经元,并根据每个神经元的输入来确定是否应激活。
2、有助于将每个神经元的输出标准化到1到0或-1到1的范围内。
常用非线性激活函数对比
激活函数
公式
函数图像
适合场景
Softmax
多分类任务输出层
Sigmoid
二分类任务输出层,模型隐藏层
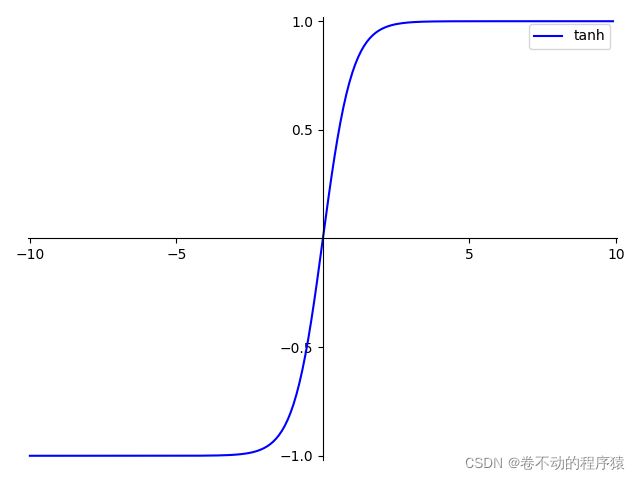
Tanh
ReLU
![]()
回归任务,卷积神经网络隐藏层
Leaky ReLU
激活函数必须满足:
- 可微,优化方法是基于梯度。
- 单调,保证单层网络是凸函数。
- 输出值范围,有限则梯度优化更稳定,无限则训练更高效(学习率需要更小)。
1、Softmax(也可视作激活函数)
常用且重要的一种归一化函数,其将输入值映射为0-1之间的概率实数,常用于多分类。
2、Sigmoid
使用范围最广的一种激活函数,具有指数形状。
优点:
在物理意义上最为接近神经元,输出是(0,1),可以被表示做概率或者用于输入的归一化,平滑的渐变,防止输出值“跳跃”。
缺点:
饱和性,从图中也不难看出其两侧导数逐渐趋近于0,可能导致梯度消失问题。
偏移现象,输出值均大于0,使得输出不是0的均值,这会导致后一层的神经元将得到上一层非0均值的信号作为输入。
梯度消失:导数值变得接近于0,导致反向传播的梯度也变得非常小,此时网络参数可能不更新。
3、Tanh(双曲正切)
优点:输出均值为0,使其收敛速度比较快,减少了迭代更新的次数。
缺点:饱和性,容易导致梯度消失。
4、ReLU(Rectified Linear Units)
公式:![]()
优点:缓解sigmoid和tanh的饱和性,当x大于0时不存在饱和性问题,计算效率高,允许网络快速收敛。
缺点:神经元死亡和偏移现象影响网络收敛性**。**
神经元死亡:随着训练,部分输入会落入硬饱和区(小于0的区域),导致权重无法更新。
5、Leaky ReLU
优点:通过在小于0部分添加参数α,解决硬饱和问题。
缺点:不稳定,结果不一致,无法为正负输入值提供一致的关系预测(不同区间函数不同)。
图像绘制代码(Python):
import math
from matplotlib import pyplot as plt
import numpy as np
def softmax(x):
return np.exp(x)/np.sum(np.exp(x), axis=0)
def sigmoid(x):
return 1. / (1 + np.exp(-x))
def tanh(x):
return (np.exp(x) - np.exp(-x)) / (np.exp(x) + np.exp(-x))
def relu(x):
return np.where(x < 0, 0, x)
def prelu(x):
return np.where(x < 0, 0.1 * x, x)
'''
def sigmoid(x):
result = 1 / (1 + math.e ** (-x))
return result
'''
def plot_softmax():
x = np.linspace(-10, 10, 200)
y = softmax(x)
plt.plot(x, y, label="softmax", linestyle='-', color='blue')
plt.legend()
plt.savefig("softmax.png")
#plt.show()
def plot_sigmoid():
fig = plt.figure()
ax = fig.add_subplot(111)
x = np.linspace(-10, 10)
y = sigmoid(x)
ax.spines['top'].set_color('none')
ax.spines['right'].set_color('none')
ax.xaxis.set_ticks_position('bottom')
ax.spines['bottom'].set_position(('data', 0))
ax.set_xticks([-10, -5, 0, 5, 10])
ax.yaxis.set_ticks_position('left')
ax.spines['left'].set_position(('data', 0))
ax.set_yticks([-1, -0.5, 0.5, 1])
plt.plot(x, y, label="Sigmoid", linestyle='-', color='blue')
plt.legend()
plt.savefig("sigmoid.png")
#plt.show()
def plot_tanh():
x = np.arange(-10, 10, 0.1)
y = tanh(x)
fig = plt.figure()
ax = fig.add_subplot(111)
ax.spines['top'].set_color('none')
ax.spines['right'].set_color('none')
ax.spines['left'].set_position(('data', 0))
ax.spines['bottom'].set_position(('data', 0))
ax.plot(x, y, label="tanh", linestyle='-', color='blue')
plt.legend()
plt.xlim([-10.05, 10.05])
plt.ylim([-1.02, 1.02])
ax.set_yticks([-1.0, -0.5, 0.5, 1.0])
ax.set_xticks([-10, -5, 5, 10])
plt.tight_layout()
plt.savefig("tanh.png")
#plt.show()
def plot_relu():
x = np.arange(-10, 10, 0.1)
y = relu(x)
fig = plt.figure()
ax = fig.add_subplot(111)
ax.spines['top'].set_color('none')
ax.spines['right'].set_color('none')
ax.spines['left'].set_position(('data', 0))
ax.plot(x, y, label="relu", linestyle='-', color='blue')
plt.legend()
plt.xlim([-10.05, 10.05])
plt.ylim([0, 10.02])
ax.set_yticks([2, 4, 6, 8, 10])
plt.tight_layout()
plt.savefig("relu.png")
#plt.show()
def plot_prelu():
x = np.arange(-10, 10, 0.1)
y = prelu(x)
fig = plt.figure()
ax = fig.add_subplot(111)
ax.spines['top'].set_color('none')
ax.spines['right'].set_color('none')
ax.spines['left'].set_position(('data', 0))
ax.spines['bottom'].set_position(('data', 0))
ax.plot(x, y, label="leaky-relu", linestyle='-', color='blue')
plt.legend()
plt.xticks([])
plt.yticks([])
plt.tight_layout()
plt.savefig("leaky-relu.png")
#plt.show()
if __name__ == "__main__":
plot_softmax()
plot_sigmoid()
plot_tanh()
plot_relu()
plot_prelu()