【Kaggle:UW-Madison GI Tract Image Segmentation】肠胃分割比赛:赛后复盘+数据再理解
目录
- 前言
- 一、任务介绍
- 三、我们的探索
-
- 3.1、baseline
-
- 3.1.1、K折交叉验证
- 3.2、数据层面
-
- 3.2.1、删除错误数据
- 3.2.2、2.5D方案探索
- 3.2.3、数据增强
- 3.3、Loss
- 3.4、模型融合
- 3.5、TTA
- 3.6、训练技巧:梯度累加
- 3.7、不work
-
- 3.7.1、多尺度训练
- 3.7.2、伪标签
- 四、其他的优秀方案
-
- 4.1、两阶段方法
- 4.2、2.5D+3D融合
- 五、经验、教训
- 六、最后看看牌子
- Reference
前言
花了一个多月的时间,打了一个kaggle比赛,这是我第一次做分割任务,还是很有意思的。
比赛地址 Kaggle肠胃分割比赛: UW-Madison GI Tract Image Segmentation
给我最大的感受就是,打比赛和发论文最大的不同在于,发论文首先会从model的角度去看待问题,会先想的是怎么让这个model更强,怎么让这个trick听起来更高大上,然后去编故事发论文。但是打比赛一定是先考虑数据的事,一定是根据对数据的理解然后去想可能有用的trick去尝试提分。
比赛最后是拿了铜牌(本来A榜是银尾的,后来B榜shake到了铜尾,别人都是shake up的,我是shake down,唉,还是对数据理解的不深入啊),然后我是组队参加的,建议大家多组队啊,可以从队友身上学到很多东西。
这篇博客主要是记录下自己打这个比赛自己探索的过程,以及比赛后看了一些top方案重新对数据的反思。
一、任务介绍
这是一个多标签、3类别、语义分割问题。
训练集共85个病例,每个病例有1-5次MRI扫描,每次扫描会截取 144张 单通道 的图片。比赛方给的数据如下,train下面放的是所有的扫描图像,按 train/casexxx/dayx 每个病例的每次扫描的图片分开存储。train.csv存放训练的mask标签,train.csv总共有三列,分别是:
- id:每个切片图片的唯一标识符,如case123_day20_slice_0001,标识第123号病例第20天的第0001张MRI切片图片,id列可以展开成三列信息:case、day和slice,通过这三个信息一样可以标记到唯一的图片;
- class,一共三类:大肠large bowel, 小肠small bowel or 胃stomach;
- segmentation:RLE segmentation mask,一种非常高效的mask编码存放方式,后面需要用RLE解码得到mask图片;
经过简单的数据探索,我们得到数据集的一些基本特点,这里总结下:
- 多标签、3类、语义分割问题;
- 训练集有85例病例,每个病例有1-5天的扫描,每次扫描144张slice切片;
- 训练集有38496张图片,有mask的16590张,没有mask的21906张;
- 大肠出现图片:小肠出现图片:胃出现图片 = 14085 : 11201 : 8627
- 图片有四种尺寸,266 x 266、360 x 310、276 x 276 、234 x 234,前两种尺寸占90%
- 测试集完全不可见,有部分是训练集出现的病例,但是有50例是训练集完全没出现过的病例,A榜和B榜各占50%的测试集;
- 每个病例每天会扫描很多的切面(1-144),slice1是最底部的图像,slice144是最上面的图像;
- 测试集虽然不可见,但是测试目录中的图像分布和格式将与训练目录中的相同;
可视化图片和mask(单通道原图)
病例18,day0,切片0060-0069的图片和mask标签可视化长这样:
三、我们的探索
3.1、baseline
我是在比赛结束前一个半月开始打的,为了快速的上手这个项目,我的baseline直接用的就是 Discussion 中的项目:UWMGI: Unet [Train] [PyTorch] 和 UWMGI: Unet [Infer] [PyTorch]
amp配置(这里做了几组消融实验才确定这个amp配置):
unet-effb3
320x320
epoch12
lr=1e-3
train_bs=64
fold=5
# 数据增强 直接用的肾小球比赛的数据增强参数
dict(type='RandomFlip', direction='horizental', p=0.5)
dict(type='RandomFlip', direction='vertical', p=0.5)
dict(type='RandomRotate',prob=0.5, degree=90, pad_val=0, seg_pad_val=255),
transforms=[dict(type='ElasticTransform', alpha=1, sigma=50, alpha_affine=50, p=0.2)
dict(type='GridDistortion', num_steps=5, distort_limit=0.05, p=0.2)
dict(type='OpticalDistortion', distort_limit=2, shift_limit=0.5, p=0.2)]
,p=0.5),
0.5bce+0.5dice
lb:0.855
3.1.1、K折交叉验证
这个trick应该是一个kaggle比赛必涨点的trick了,所以我这个比赛也直接用了这个trick。关于原理不理解的可以看下这篇博文: 【Kaggle比赛常用trick】K折交叉验证、TTA。
不过这个比赛比较特殊,我是这样用的:
# 划分为CFG.n_fold折,打乱数据集顺序进行训练
skf = StratifiedGroupKFold(n_splits=CFG.n_fold, shuffle=True, random_state=CFG.seed)
# 先按病例分group, 保证每一个病例的所有图片只在一折上,防止出现同一病例的图片既在训练集又在验证集上
# 再在group的基础上按照empty进行分层采样,保证每一折有mask和无mask的样本尽量均衡
for fold, (train_idx, val_idx) in enumerate(skf.split(df, df['empty'], groups = df["case"])):
3.2、数据层面
3.2.1、删除错误数据
这部分我们主要是参考这个 kaggle: discussion 和 kaggle: discussion,看看大佬的发现,说实话自己不太可能一张张的检测数据,而且这个任务的数据还是 1 channel 的数据,不是很好观察,所以我就直接去 Discussion找,没想到还真有人探索这个问题,Discussion牛皮!
我把大佬们的发现总结了一下,下面这些数据都是有问题的:
mask是错的:CASE 7 –– DAY 0
mask是错的:CASE 43 –– DAY 18 –– Day26
mask是错的:CASE 81 –– DAY 30
缺少mask:Case85 –– Day23 slices 119-124
缺少mask:Case90 –– Day29 slices 115-119
缺少mask:case133 –– Day25 slices 111-113
mask是错的:CASE 138 –– DAY 0
代码:
# 删除脏数据
from tqdm.notebook import tqdm
Case138_Day0 = [i for i in range(76,145)]
Case85_Day23 = [119,120,121,122,123,124]
Case90_Day29 = [115,116,117,118,119]
Case133_Day25 = [111,112,113]
df3 = df1.copy()
Case7 = []
Case43 = []
Case81 = []
Case85 = []
Case90 = []
Case133 = []
Case138 = []
for i,row in tqdm(df1.iterrows(), total=len(df1)):
if row.id.rsplit("_",2)[0]=='case7_day0':
Case7.append(i)
elif row.id.rsplit("_",2)[0]=='case43_day18' or row.id.rsplit("_",2)[0]=='case43_day26':
Case43.append(i)
elif row.id.rsplit("_",2)[0]=='case81_day30':
Case81.append(i)
elif row.id.rsplit("_",2)[0]=='case138_day0':
if int(row.id.rsplit("_",1)[-1]) in Case138_Day0:
Case138.append(i)
print(len(Case7),len(Case43),len(Case138))
df1.drop(index=Case7+Case43+Case81+Case138 ,inplace=True)
df3 = df1.reset_index(drop=True)
删除前后数据对比:
| - | 删除前数据 | 删除后数据 |
|---|---|---|
| 总图片数量 | 38496 | 37851 |
| 空mask图片数量 | 21906 | 21553 |
| 有mask图片数量 | 16590 | 16298 |
| 病例数量 | 85 | 85 |
总共删除645张图片,有mask的292张,没mask的353张。
性能提升:lb=0.856
3.2.2、2.5D方案探索
原先的数据是单通道的数据,为了让这种单通道的数据能在2D的网络上跑起来,baseline是将每个图片复制3份,然后直接concat拼接,送入网络进行训练的。
我自己探索数据一星期左右,又看到大佬开源了对2.5D数据的探讨: UWMGI: 2.5D [Train] [PyTorch] 和 UWMGI: 2.5D [Infer] [PyTorch]
2.5D的思路:对于slice3这张训练图片而言,我不再是把slice3复制3份再concat送入网络了,而是将slice1、slice3、slice5这三种图片concat起来,再送入网络中训练,mask还是实验slice3的mask。依次增加模型的深度信息/空间上的信息。
代码:
channels=3
stride=2
for i in range(channels):
df[f'image_path_{i:02}'] = df.groupby(['case','day'])['image_path'].shift(-i*stride).fillna(method="ffill")
df['image_paths'] = df[[f'image_path_{i:02d}' for i in range(channels)]].values.tolist()
几个细节:
- 测试集的格式,也是按照跟训练集一样的形式组织的,这就为我们造2.5D格式的依据创造了条件;测试集我们看不到,但是测试集也要进行构造2.5D再推理的。构造方法和train类似,直接修改csv中每条mask_path对应的img_paths的路径,把s-2和s+2的图片路径加入进来,推理的时候直接concat拼接即可。
- stride=2的想法就是说,如果太相近的时间序列的图片,它的信息太相近了,不能带来太多的深度信息;太远了也不行,太远了差异性太大,会造成那次重叠信息反而效果更差(因为mask用的是中间slice的mask)。
- 对于所有病人的每一天(1-5)天图片数据进行2.5D化,如果s-2没有那么就复制s+2的图片,比如slice3+slice1+slice3+mask1、slice4+slice2+slice4+mask2;如果s+2没有就复制s-2的图片,比如slice142+slice144+slice142+mask144、slice141+slice143+slice141+mask143。
2.5D改造后的图片和mask:
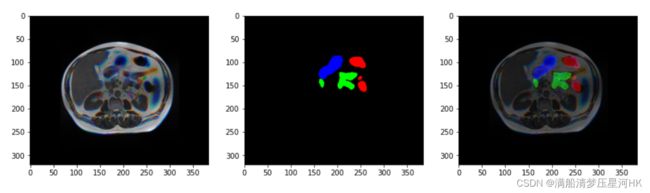
效果提升最大,lb=0.869
对比实验:s=1 3channel、s=2 3channel、s=1 5channel
3.2.3、数据增强
看了看大佬关于数据的理解分析: Data augmentations that make sense
这个比赛的胸腔位置相对固定,肠胃分布也相对固定,所以初步分析数据增强:
- 水平翻转是可行的,竖着翻转是不可行的,因为数据集中整个胸腔的位置不会颠倒的;
- 小范围的旋转是非常正常的,但是大范围的旋转是没有意义的;
- 缩放是完全有意义的,因为人的胃、大肠、小肠大小是不一样的;
- 位移是没有意义的,几乎所有的胸腔都是位于图片的中心区域,位移了反而打乱数据的分布;
- 颜色变换和亮度增强也没有意义,图像就是黑白色的,亮度也差不多是一样的;
最终的实验方案:
CenterCrop # 上下左右各裁剪5%
dict(type='RandomFlip', direction='horizental', p=0.5)
# 三种非刚性变换 描述的是对几何物体大小的改变 形变比较小的
transforms=[dict(type='ElasticTransform', alpha=1, sigma=50, alpha_affine=50, p=0.5) # 弹性变换
dict(type='GridDistortion', num_steps=5, distort_limit=0.05, p=0.5) # 网格失真
dict(type='OpticalDistortion', distort_limit=2, shift_limit=0.5, p=0.5)] # 光学畸变
,p=1)
def CenterCrop(image, crop_ratio=0.9):
height, width, channel = image.shape
xmin, ymin, xmax, ymax = int(width * (1 - crop_ratio) / 2), \
int(height * (1 - crop_ratio) / 2), \
int(width * (1 + crop_ratio) / 2), \
int(height * (1 + crop_ratio) / 2)
crop_image = image[ymin:ymax, xmin:xmax, ...]
extra_info = [height, width, xmin, ymin, xmax, ymax]
return crop_image, extra_info
实验结果:
- 中心裁剪是一个非常常用的一个trick,在很多比赛上都能看到它的身影。这个比赛由于胸腔几乎都是位于图片的中心区域,所以我第一个想到的trick就是中心裁剪;
- 水平翻转ok,竖着翻转直接掉点;
- 小范围旋转试了好久,10度、15度、30度都没用,可能是医学数据集它数据集真的就全是正正方方的吧,没有一点歪;
- 位移有可能会使胸腔区域不完整;
- 颜色全是黑白的没用,亮度也没用,猜测可能数据集是在同一个房间拍的,那它的亮度都是相同的,没有差异;
性能效果,lb=0.873
3.3、Loss
因为数据集当中有mask的16590张,没有mask的21906张,没有mask的图片比有mask的图片还多,而且有mask的图片当中也有一些背景区域的,所以整个数据集我认为是有正负样本不平衡问题的。所以这里尝试使用focal loss来代替bce。
最后的方案,0.5focal loss+0.5dice loss
focal loss: gamma=1.0,alpha=0.5
lb = 0.875
到此为止,单模总共最高是打到了0.875,从baseline的0.854到现在的单模0.875还是很爽的,到此,比赛也快结束了,下面是尝试模型融合部分。
3.4、模型融合
对齐单模的所有超参,我又跑了effb4、b5、b6、b7,因为我单卡3090,后面4个模型没法bs=64跑了,我就用了梯度累加的方法,把bs降到了32,每两个mini-batch更新一次梯度,具体原理下面会讲。
跑完之后发现可能是因为b6、b7的模型太大太复杂了,而我们本次的数据集真的算小了,所以可能出现过拟合了,b6、b7反而降分了。
| b2 | b3 | b4 | b5 | b6 | b7 |
|---|---|---|---|---|---|
| 0.873 | 0.875 | 0.877 | 0.876 | 0.873 | 0.873 |
最终用(b3、b4、b5三个模型)* 5折进行模型融合,代码如下:
for _, (images, ids, h, w) in pbar:
images = images.to(CFG.device, dtype=torch.float) # [b, c, w, h]
size = images.size()
masks = torch.zeros((size[0], 3, size[2], size[3]), device=CFG.device, dtype=torch.float32) # [b, c, w, h]
# cross validation infer
for sub_ckpt_path in ckpt_paths:
model = build_model(CFG, test_flag=True)
model.load_state_dict(torch.load(sub_ckpt_path))
model.eval()
y_preds = model(images) # [b, c, w, h]
y_preds = torch.nn.Sigmoid()(y_preds)
# 每个channel对应位置相加
masks += y_preds/len(ckpt_paths)
# 最后大于0.5的保留
masks = (masks.permute((0, 2, 3, 1))>CFG.thr).to(torch.uint8).cpu().detach().numpy() # [n, h, w, c]
说白了就是讲这15个模型全部进行前向推理,得到15个mask,然后让这15个mask进行element-wise add,再除以15,然后再进行阈值判断,如果>0.5说明属于这个类别。
lb=0.880
3.5、TTA
使用horizental进行测试增强,代码如下:
for _, (images, ids, h, w) in pbar:
images = images.to(CFG.device, dtype=torch.float) # [b, c, w, h]
size = images.size()
masks = torch.zeros((size[0], 3, size[2], size[3]), device=CFG.device, dtype=torch.float32) # [b, c, w, h]
# cross validation and TTA
total_ckpt_paths = len(ckpt_paths_dict) * CFG.n_fold
for backbone_name, ckpt_paths in ckpt_paths_dict.items():
CFG.backbone = backbone_name
for sub_ckpt_path in ckpt_paths:
model = build_model(CFG, test_flag=True)
model.load_state_dict(torch.load(sub_ckpt_path))
model.eval()
y_preds = model(images) # [b, c, w, h]
y_preds = torch.nn.Sigmoid()(y_preds)
masks += y_preds
if CFG.tta:
# flips = [[-1],[-2],[-2,-1]] # x,y,xy flips as TTA
flips = [[-1]] # x flip as TTA
for f in flips:
images_f = torch.flip(images, f)
y_preds = model(images_f) # [b, c, w, h]
y_preds = torch.flip(y_preds, f)
y_preds = torch.nn.Sigmoid()(y_preds)
masks += y_preds
if CFG.tta:
total_ckpt_paths = len(ckpt_paths_dict) * CFG.n_fold * 2
else:
total_ckpt_paths = len(ckpt_paths_dict) * CFG.n_fold
masks /= total_ckpt_paths
masks = (masks.permute((0, 2, 3, 1))>CFG.thr).to(torch.uint8).cpu().detach().numpy() # [n, h, w, c]
可以看出TTA的原理就是先对原图进行水平翻转,再送进模型,推理得到mask,再对mask进行水平翻转,然后和其他的模型一起做模型融合。最后呢我们的整个程序相当于有30个权重文件进行进行融合。
涨点了0.001,lb=0.881
3.6、训练技巧:梯度累加
原来的bs我们一直设置的64,但是effb4之后就无法使用64继续进行训练了(单张3090太伤了,只能找这种曲线救国的方法~),为了继续控制变量进行对比实验,这里我们使用梯度累加的方法。将bs变为32,但是2个batch再更新一次梯度,相当于变相的增大bs。不清楚的可以看看这篇文章:深度学习节省显存的trick之梯度累积。
具体使用方法如下:
计算损失
loss = loss / 2 # 计算当前mini-batch的loss 注意要/2 否则可能发生梯度爆炸
loss.backward() # 反向传播计算梯度 并累加梯度
if step % 2 == 0
optimizer.step() # 每2个mini-batch更新一次参数
optimizer.zero_grad() # 梯度清零
step+=1
- 注意这里是梯度进行累加,并不是累加loss。
- 梯度累加可能会让BN失效,但是我这里bs是从64->32,个人认为差别还是不大的,但是如果你的bs是从8->4/4->2这种,感觉还是慎用梯度累加,可能会让BN失效,效果反而变差。
3.7、不work
3.7.1、多尺度训练
因为这个数据集给了四种图片尺寸,266 x 266、360 x 310、276 x 276 、234 x 234,加上每个人的器官的大小都是不一样的,所以我认为多尺度训练可以有利于我们检测不同个体的不同大小的器官。所以探索了很久,设置224x224、256x256、288x288、320x320四种尺度随机进行多尺度训练,但是发现怎么也不涨点,反而还掉0.001-0.002,气死。
但是比赛完了之后,看到一个大佬的多尺度渐进式训练方案:使用 [224, 256], [256, 288], [288, 320],[320, 384]这四个尺度,每隔5个epoch涨一次分辨率,直到涨到[320, 384]。测试使用最大分辨率进行测试。然后他居然涨点了…
我的随机多尺度训练为什么不涨点?
3.7.2、伪标签
伪标签的思想来自于俄罗斯的第一位kaggle master。
- 我把我训练最好的单模去预测我删除的哪些脏数据,然后预测完再将所有的数据重新训练,相当于增大了数据量。但是发现几乎没有涨点;
- 还有一种伪标签,不过因为时间原因比赛到这就差不多结束了,我就没试了。这里我说下思想吧:在训练好单模去预测测试集的时候,把每个测试打出预测的label,然后存放在测试集的目录下,再对测试集和训练集一起训练一个epoch(kaggle有9个小时限制),训练完之后再对测试集进行test,得到csv,提交。这种方式,相当于就是让我们训练好的模型偷看了一下测试集。
四、其他的优秀方案
4.1、两阶段方法
因为这个比赛是有没有mask的数据,而且训练集当中没有mask的数据比有mask的数据还要多,测试集的比例并不知道,但是肯定也是有没用标注的mask的。
我的解决方案是使用focal loss代替BCE去解决它的正负样本不平衡问题。但是我看了大佬的方案后发现,有很多人都是使用两阶段的算法来更精确的解决这个问题的:
- 第一阶段使用yolov5对图片中的胸腔进行目标检测,如果有目标就将胸腔区域裁剪出来,然后继续进行第二阶段的分割。如果没用目标直接过下一张。训练时打了200张胸腔目标数据进行目标检测网络yolov5的训练,使用所有裁剪出来的胸腔图片进行分割训练。
- 第一阶段先使用一个分类网络对图片进行分类,如果有胸腔,就对这张图片进行分割,如果没有胸腔就直接过下一张。训练时使用所有数据进行分类训练,使用所有有mask的数据进行分割训练。
4.2、2.5D+3D融合
3D模型的思路后面有机会再讲吧,现在还不是很了解3D UNet模型。
3D图像分x、y、z轴,增加的z轴信息 / 空间信息 / 深度信息。
五、经验、教训
其实我们A榜是在银牌尾部的,但是最后B榜就变成铜牌区末尾了,我感觉的主要原因:
- 测试数据中肯定也有很多的没用mask的图片,虽然我用了focal loss,但是可以发现其实A榜其实只涨了0.002分,其实还是有很大问题的。相反几乎前10名全是使用了两阶段网络的,而且全是shape up进前10名的,所以两阶段能更彻底的解决这个数据集很多图片没用mask的问题;
- 模型融合,我没有使用3D的模型,后来发现几乎所有前面的大佬都融合了3D的模型,最后都涨了分,而且第一名的大佬根本就没有自己训3D模型,他直接拿了 这个Discussion 开源的3D模型权重和推理文件直接融合的,涨了0.004+,然后我当时觉得我的电脑跑个unet都费劲,我就不费劲去训练模型了,然后就后悔了…,真的是第一次参加没经验,鬼知道直接拿别人的权重就涨了,唉,应该试试的,直接拿权重和推理文件融合一下又不难。
经验:
- 多用模型融合,多使用不同类型的模型进行融合,也许可以起到一个补充的作用;
- 多思考数据,少思考模型
六、最后看看牌子
Reference
比赛地址: UW-Madison GI Tract Image Segmentation
比赛快速理解: Collection of My Resources/Thoughts For This Competition
数据集理解1: EDA & In Depth Mask Exploration
2.5D数据: UWMGI: 2.5D stride=2 Data
关于这个比赛数据增强的理解:Data augmentations that make sense
3D模型解决方案 :[LB 0.877] A 3D solution with MONAI