机器学习(4)--breastcancer随机森林网格搜索
目录
一、GridSearchcv概述
二、重要参数
三、Breastcancer数据集网格搜索
1、导入库
2、通过多次交叉验证求最优的n_estimators参数
编辑
3、网格搜索调参
4、调整max_features参数
一、GridSearchcv概述
GridSearchcv的存在意义是自动调参,通过输入多个不同的参数,给出最优化的acc结果和参数值,但这个方法适合于小型数据集。数据量大的时候需要用到坐标下降的快速调参方式,其实是一种贪心算法,每次放入一个影响最大的参数进行调优,并求出最优的参数,基于最优参数的情况下,继续对影响度其次的参数进行调优,以此类推。但是这个方法会存在缺点,可能会调到局部最优解的弊端。
二、重要参数
sklearn.model_selection._search.GridSearchCV def __init__(self,
estimator: Any,
param_grid: Union[dict, list],
*,
scoring: Any = None,
n_jobs: Any = None,
refit: Any = True,
cv: Any = None,
verbose: int = 0,
pre_dispatch: Any = "2*n_jobs",
error_score: Any = np.nan,
return_train_score: Any = False) -> None
| estimator | 选择的分类器,并且传入除需要确定最佳的参数之外的其他参数 |
| param_grid | 需要最优化得参数的取值,值为字典或者列表。字典的key值为参数名,value为参数调整范围 |
| n_jobs | CPU并行数,默认为1,值为-1时代表全部CPU核运行 |
| cv | 交叉验证参数,默认为None |
三、Breastcancer数据集网格搜索
1、导入库
from sklearn.datasets import load_breast_cancer #导入breastcancer数据库
from sklearn.ensemble import RandomForestClassifier #导入随机森林库
from sklearn.model_selection import cross_val_score #交叉验证
from sklearn.model_selection import GridSearchCV #网格搜索
import matplotlib.pyplot as plt
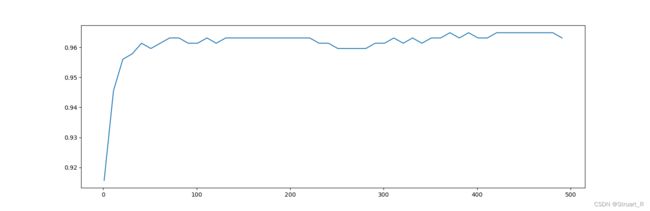
import pandas as pd2、通过多次交叉验证求最优的n_estimators参数
score_l=[]
for i in range(0,500,10):
rfc=RandomForestClassifier(n_estimators=i+1
,n_jobs=-1
,random_state=90)
score=cross_val_score(rfc,cancer.data,cancer.target,cv=10).mean()
score_l.append(score)
print(max(score_l),score_l.index(max(score_l))*10+1)
plt.figure(figsize=[20,5])
plt.plot(range(1,501,10),score_l)
plt.show()测试时最优的n_estimators参数为371,acc值约为0.96488,接下来基于改最优参数按照调参顺序进行下一步调参。
3、网格搜索调参
cancer=load_breast_cancer()
param_grid={'max_depth':np.arange(1,20,1)}
rfc=RandomForestClassifier(n_estimators=371
,random_state=90
,n_jobs=-1)
GS=GridSearchCV(rfc,param_grid,cv=10)
GS.fit(cancer.data,cancer.target)
print(GS.best_params_) #输出该参数最优值
print(GS.best_score_) #输出该参数最优时,acc值逐一调参优化max_depth,min_samples_split,min_sample_leaf,发现acc值并没有改变显然按着这个方法调下去,acc值不会再改变了。所以判断在最佳模型复杂度左侧,只能通过调整max_features值(唯一一个可以向左或向右调整,来使acc值更高的参数)。
4、调整max_features参数
param_grid={'max_features':np.arange(1,20,1)}
rfc=RandomForestClassifier(n_estimators=371
,max_depth=7
,random_state=90
,n_jobs=-1)
GS=GridSearchCV(rfc,param_grid,cv=10)
GS.fit(cancer.data,cancer.target)
print(GS.best_params_)
print(GS.best_score_)测试后得到max_features=7,acc值为0.96667,获得更优的参数。