SCS【3】单细胞转录组数据 GEO 下载及读取
点击关注,桓峰基因
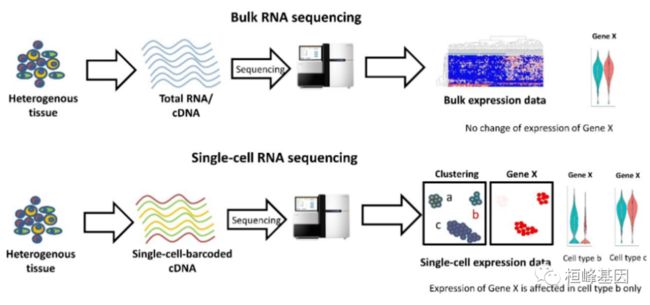
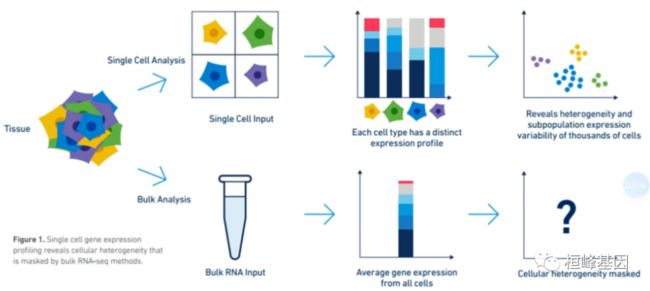
今天来介绍一下GEO单细胞转录组下载数据以及整理,单细胞测序的原理以及数据结果都与bulk测序的方式有一定的差距,所以我们单独说一下。
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.
桓峰基因公众号推出单细胞系列教程,有需要生信的老师可以联系我们!首选看下转录分析教程整理如下:
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
前言
单细胞近几年也是越来越火热,因此数据也都上传到GEO,这样我们有些分析就可以不用测序,而是通过单细胞现有的数据进行分析了,目前GEO提供数据大致包括三类:
-
原始数据:fastq格式原始测序数据
-
经过cellranger标准化后的10X数据
-
Counts数据信息
当然我们都希望获得第二种数据,毕竟从原始下机数据fastq处理起来,自己的小电脑也受不起,并且不是每个人都很擅长 Linux 和 shell ,所以有了第二种,对于我们来说算是省去了很多繁琐的步骤。
数据下载
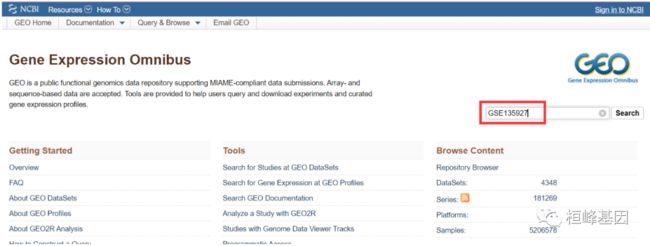
确定我们需要下载的GEO编号,我们这里使用 GSE135927, 登录网站
https://www.ncbi.nlm.nih.gov/geo/
数据集介绍
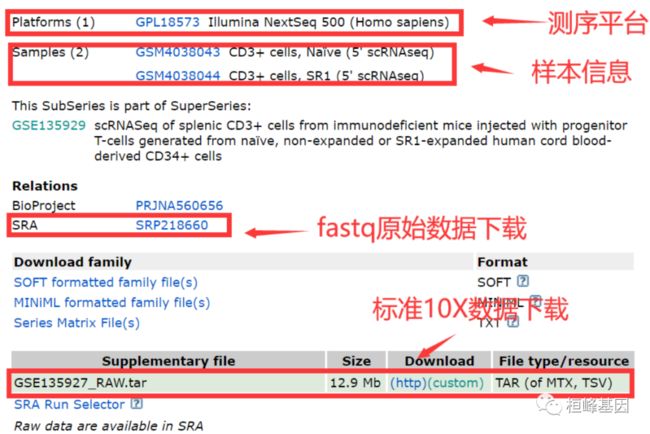
我们这里选择Cell Ranger生成的raw count。
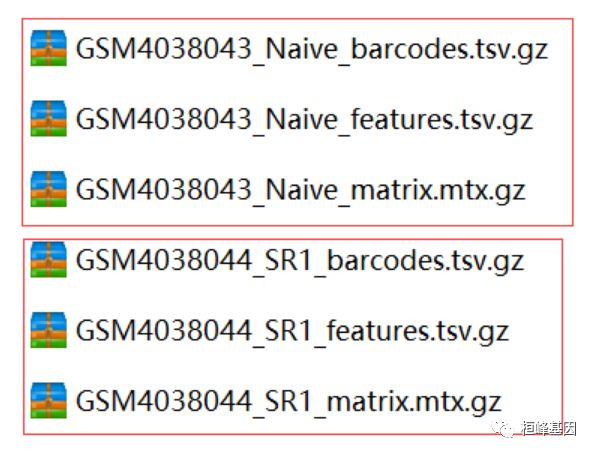
下载GSE135927_RAW.tar后解压,我们可以看到每个样本有三个文件,包括:
- barcodes.tsv
Barcodes通俗来讲就是每个细胞的代码,组成就是ATCG四个碱基排列组合成的不同的14个碱基组合;
AAACCTGAGGGCTCTC-1
AAACCTGAGTGAACGC-1
AAACCTGGTATGAATG-1
AAACCTGTCTGCGACG-1
AAACGGGAGGTGCAAC-1
- features.tsv
features.tsv一般是基因的ensembl ID 和symbol
ENSG00000243485 MIR1302-2HG Gene Expression
ENSG00000237613 FAM138A Gene Expression
ENSG00000186092 OR4F5 Gene Expression
ENSG00000238009 AL627309.1 Gene Expression
ENSG00000239945 AL627309.3 Gene Expression
ENSG00000239906 AL627309.2 Gene Expression
ENSG00000241599 AL627309.4 Gene Expression
- matrix.mtx
matrix.mtx就是每个细胞不同基因的表达矩阵:
%%MatrixMarket matrix coordinate integer general
%metadata_json: {"format_version": 2, "software_version": "3.0.1"}
3353813593017342
33509124
3350812
3350716
3350616
33505126
33504119
33503119
33502124
33501113
软件安装
我们使用Seurat软件包里的函数Read10X()进行数据读取。
if(!require(Seurat))
BiocManager::install("Seurat")
数据读取
我们发现下载后的每个样本包含三个文件,准备好每个样本一个文件夹。我们先看下函数说明给出来的例子:
1. CellRanger < 3.0
如果是CellRanger < 3.0 单样本读取的时候必须是每个文件格式为 barcodes.tsv, genes.tsv, and matrix.mtx
## Not run:
# For output from CellRanger < 3.0
data_dir <- 'path/to/data/directory'
list.files(data_dir) # Should show barcodes.tsv, genes.tsv, and matrix.mtx
expression_matrix <- Read10X(data.dir = data_dir)
seurat_object = CreateSeuratObject(counts = expression_matrix)
2. CellRanger >= 3.0
如果是CellRanger >= 3.0 单样本读取的时候必须是每个文件格式为barcodes.tsv.gz, features.tsv.gz, and matrix.mtx.gz
# For output from CellRanger >= 3.0 with multiple data types
data_dir <- 'path/to/data/directory'
list.files(data_dir) # Should show barcodes.tsv.gz, features.tsv.gz, and matrix.mtx.gz
data <- Read10X(data.dir = data_dir)
seurat_object = CreateSeuratObject(counts = data$`Gene Expression`)
seurat_object[['Protein']] = CreateAssayObject(counts = data$`Antibody Capture`)
3. 实际例子
我们先看下单个样本处理方法和多个样本合并。
单个样本读取
library(Seurat)
data_dir <- "./GSE135927"
dirs = list.dirs(data_dir)
dirs_sample = dirs[2:3]
dirs_sample
## [1] "./GSE135927/GSM4038043" "./GSE135927/GSM4038044"
GSM4038043 = Read10X(data.dir = dirs_sample[1])
GSM4038043[1:10, 1:3]
## 10 x 3 sparse Matrix of class "dgCMatrix"
## AAACCTGAGGGCTCTC-1 AAACCTGAGTGAACGC-1 AAACCTGGTATGAATG-1
## MIR1302-2HG . . .
## FAM138A . . .
## OR4F5 . . .
## AL627309.1 . . .
## AL627309.3 . . .
## AL627309.2 . . .
## AL627309.4 . . .
## AL732372.1 . . .
## OR4F29 . . .
## AC114498.1 . . .
s1 = CreateSeuratObject(counts = GSM4038043, min.cells = 3, min.features = 200)
head([email protected])
## orig.ident nCount_RNA nFeature_RNA
## AAACCTGAGGGCTCTC-1 SeuratProject 8506 2450
## AAACCTGAGTGAACGC-1 SeuratProject 5634 1743
## AAACCTGGTATGAATG-1 SeuratProject 8841 2309
## AAACCTGTCTGCGACG-1 SeuratProject 4484 1779
## AAACGGGAGGTGCAAC-1 SeuratProject 8221 2332
## AAACGGGTCGCTAGCG-1 SeuratProject 6471 2153
多个样本读取
GSM4038044 = Read10X(data.dir = dirs_sample[2])
s2 = CreateSeuratObject(counts = GSM4038044, min.cells = 3, min.features = 200)
m = merge(x = s1, y = s2, add.cell.ids = c("GSM4038043", "GSM4038044"), merge.data = TRUE)
as.data.frame(m@assays$RNA@counts[1:10, 1:2])
## GSM4038043_AAACCTGAGGGCTCTC-1 GSM4038043_AAACCTGAGTGAACGC-1
## AL669831.5 0 0
## LINC00115 0 0
## FAM41C 0 0
## NOC2L 0 0
## KLHL17 0 0
## PLEKHN1 0 0
## HES4 0 0
## ISG15 2 0
## AL645608.2 0 0
## AGRN 0 0
head([email protected])
## orig.ident nCount_RNA nFeature_RNA
## GSM4038043_AAACCTGAGGGCTCTC-1 SeuratProject 8506 2450
## GSM4038043_AAACCTGAGTGAACGC-1 SeuratProject 5634 1743
## GSM4038043_AAACCTGGTATGAATG-1 SeuratProject 8841 2309
## GSM4038043_AAACCTGTCTGCGACG-1 SeuratProject 4484 1779
## GSM4038043_AAACGGGAGGTGCAAC-1 SeuratProject 8221 2332
## GSM4038043_AAACGGGTCGCTAGCG-1 SeuratProject 6471 2153
总结
另外还有一种,只有一个表格,下载之后可以自己整理结果,例如 GSE135928 下载之后我们看下文件格式:
barcode is_cell contig_id high_confidence length chain v_gene d_gene j_gene c_gene full_length productive cdr3 cdr3_nt reads umis raw_clonotype_id raw_consensus_id
AAACGGGTCGTCGTTC-1TRUE AAACGGGTCGTCGTTC-1_contig_1 TRUE582 TRA TRAV5 None TRAJ8 TRAC TRUETRUE CAESSGTGFQKLVF TGTGCAGAGAGTTCCGGCACAGGCTTTCAGAAACTTGTATTT 19453 clonotype17 clonotype17_consensus_2
AAACGGGTCGTCGTTC-1TRUE AAACGGGTCGTCGTTC-1_contig_2 TRUE710 TRB TRBV4-3 TRBD2 TRBJ2-7 TRBC2 TRUETRUE CASSQDSGPSYEQYF TGCGCCAGCAGCCAAGATAGTGGGCCATCCTACGAGCAGTACTTC 468810 clonotype17 clonotype17_consensus_1
AAACGGGTCTCGAGTA-1TRUE AAACGGGTCTCGAGTA-1_contig_1 TRUE689 TRB TRBV5-1 TRBD1 TRBJ2-2 TRBC2 TRUETRUE CASSYRGVNTGELFF TGCGCCAGCAGCTACAGGGGCGTGAACACCGGGGAGCTGTTTTTT 619013 clonotype18 clonotype18_consensus_2
AAACGGGTCTCGAGTA-1TRUE AAACGGGTCTCGAGTA-1_contig_3 TRUE343 Multi None None TRBJ2-3 TRBC2 FALSE None None None 7374 clonotype18 None
AAACGGGTCTCGAGTA-1TRUE AAACGGGTCTCGAGTA-1_contig_4 TRUE694 TRA TRAV8-6 None TRAJ52 TRAC TRUETRUE CAVIKANAGGTSYGKLTF TGTGCTGTGATCAAAGCTAATGCTGGTGGTACTAGCTATGGAAAGCTGACATTT 54838 clonotype18 clonotype18_consensus_1
AAAGATGAGGTAAACT-1TRUE AAAGATGAGGTAAACT-1_contig_1 TRUE605 TRA TRAV12-3 None TRAJ22 TRAC TRUETRUE CARGSARQLTF TGTGCACGGGGTTCTGCAAGGCAACTGACCTTT 77239 clonotype19 clonotype19_consensus_1
AAAGATGAGGTAAACT-1TRUE AAAGATGAGGTAAACT-1_contig_3 TRUE543 TRB TRBV6-3 TRBD1 TRBJ2-5 TRBC2 TRUETRUE CASSYSFSGQGGETQYF TGTGCCAGCAGTTACTCATTTTCGGGACAAGGCGGAGAGACCCAGTACTTC 1098420 clonotype19 clonotype19_consensus_2
AAAGATGCACGACGAA-1TRUE AAAGATGCACGACGAA-1_contig_1 TRUE517 TRB TRBV4-1 TRBD2 TRBJ1-1 TRBC1 TRUETRUE CASSQDGLMNTEAFF TGCGCCAGCAGCCAAGATGGACTTATGAACACTGAAGCTTTCTTT 34097 clonotype20 clonotype20_consensus_2
AAAGATGCACGACGAA-1TRUE AAAGATGCACGACGAA-1_contig_2 TRUE543 TRA TRAV23DV6 None TRAJ37 TRAC TRUETRUE CAASRVNTGKLIF TGTGCAGCAAGCAGAGTCAACACAGGCAAACTAATCTTT 20283 clonotype20 clonotype20_consensus_1
这种格式起始也非常好用,利用TPM计算原理,有length,reads,barcode,raw_consensus_id 等信息计算出来获得一个表达矩阵即可。
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
References:
- Singh J, Chen ELY, Xing Y, Stefanski HE et al. Generation and function of progenitor T cells from StemRegenin-1-expanded CD34+ human hematopoietic progenitor cells. Blood Adv 2019 Oct 22;3(20):2934-2948. PMID: 31648315