python乳腺癌细胞挖掘
随着人们生活水平提高,大家不仅关注如何生活,而且关注如何生活得更好。在这个背景下,精准治疗和预测诊断成为当今热门话题。
据权威医学资料统计,全球大约每13分钟就有一人死于乳腺癌,乳腺癌已成为威胁当代人健康的主要疾病之一,并且随着发病率的增加,死亡率也逐渐增加,作为女性实在不能不重视。
其中前十位死因;女性乳腺癌为首因,其余顺序与全人群死因一致。其中,乳腺癌可能没有任何先兆,是一个隐形杀手。
有不少人的乳腺癌是没有任何征兆的,有可能只是发现肿块但没有任何不舒服的地方,但一检查就确诊乳腺癌的情况不在少数,更甚至于有些人已经发展到乳腺癌晚期,只能采取切除患病乳房的方式来挽救生命。因此一定要密切注意乳房的变化,每年体检一次,排除患癌因素最保险。有权威数据统计,中国将进入乳腺癌高峰期,到2021年中国将有250万人患乳腺癌!保养胸部将不再是“喜不喜欢、需不需要”可选问题,在未来的日子里乳腺癌预防将是每个不同年龄女人的必须选择。
乳腺癌的主要症状包括:
1、肿块
为95%乳腺癌病人的乎发症状。大多单发,少见多发,形态偏于圆形、椭圆形或不规则形。质地一般较硬、境界不清。个别如髓样癌质地较软,境界较清。多发于外上象限,肿块增大较快,早期可有活动度。
2、皮肤改变
常见为浅表静脉怒张,酒窝征和桔皮样皮肤。炎性乳癌病人胸部皮肤可大片颜色变暗,呈硬结、增厚,杂以癌性斑块和溃疡呈铠甲状胸壁。晚期乳癌可向浅表溃破,形成溃疡或菜花状新生物。
3、乳头乳晕改变
乳房中央区乳腺癌,大导管受侵犯可致乳头扁平、凹陷、回缩,甚至乳头陷入晕下,导致乳晕变形。Paget氏病可出现乳头、乳晕皮肤湿疹样改变。
4、乳头溢液
乳腺癌伴溢液占乳癌总数的1.3-7%,且多见于管内癌、乳头状癌。血性溢液多见,次为浆液性、浆血性、水样等也有。以溢液为唯一症状乳癌,极少见,且大多为早期管内癌、乳头状癌,溢液乳腺癌多数先发现肿块后伴有溢液。
5、疼痛
早期出现的为无痛性肿块。乳癌合并囊性增生病时,可有胀痛、钝痛。晚期乳癌疼痛常提示肿瘤直接侵犯神经。
6、腋淋巴结肿大
作为乳腺癌首发症状少见(除非隐匿型乳腺癌)。大多提示乳腺癌病程进展,需排除上肢、肩背、胸部其他恶性肿瘤转移所致。
精准医疗和诊断预测离不开计算机编程,临床数据和机器学习算法。
乳腺癌是世界各地女性常见的癌症,通过尽早对患者进行临床治疗,尽早发现BC可大大改善预后和生存机会。因此,仅通过使用数据,python和机器学习就能帮助挽救生命真是太神奇了!通过下述代码,您已经完成创建乳房检测程序来预测患者是否患有癌症!同样,如果您愿意,您可以报名听我讲解课程的所有代码。
欢迎各位同学学习《python机器学习-乳腺癌细胞挖掘》课程,教会大家建立诊断预测乳腺癌细胞模型
作者介绍
Toby,持牌照金融公司担任模型验证专家,国内最大医药数据中心数据挖掘部门负责人!和重庆儿科医院,中科院教授,赛柏蓝保持慢病数据挖掘项目合作!管理过欧美日中印巴西等国外药典数据库,马丁代尔数据库,FDA溶解度数据库,临床试验数据库,WHO药物预警等数据库。
课程概述
此课程讲述如何运用python的sklearn快速建立机器学习模型。课程结合美国威斯康辛乳腺癌细胞临床数据,实操演练,建立癌细胞预测分类器。
课程讲述十大经典机器学习算法:逻辑回归,支持向量,KNN,神经网络,随机森林,xgboost,lightGBM,catboost。这些算法模型可以应用于各个领域数据。
本视频系列通俗易懂,课程针对学生和科研机构,python爱好者。
本视频教程系列有完整python代码,观众看后可以下载实际操作。
了解癌症肿瘤基本常识,建立健康生活方式,预防癌症,减轻癌症治疗成本。
适用人群
研究生,博士生毕业论文,NCBI/SCI/Nature论文发布,python爱好者,机器学习,生物信息学,乳腺癌医学科研机构
课程背景
警钟长鸣!癌症离我们远吗?《我不是药神》催人泪下,笔者在此揭露真相,癌症不是小概率疾病,癌症就在身边。癌症早期发现和控制可极大延长寿命和减少治疗费用。笔者下载美国威斯康辛临床数据,运用python sklearn机器学习十大经典算法建立乳腺癌分类器模型,可预测正常细胞和癌细胞。我国医院重视治疗,但忽略疾病预防教育。通过我多年机器学习数据挖掘,我发现疾病可防可控,通过自身努力,我们可以提前发现疾病早期症状或扼杀疾病于摇篮。希望此课程让广大医疗科研工作者认识疾病预防教育重要性。课程还介绍RDKIT概述-开源化学信息工具包,以及如何运用python语言构建Rdkit化学分子溶解度模型,此乃化学,生物信息学爱好者又一个福音。
课程目录
章节1我的主页和课前咨询答疑
课时1课程概述(必看)
课时2python机器学习生物信息学概述(必看)
课时3欢迎项目合作
课时4如何下载脚本和原始数据
课时5所有数据和脚本在此课参考资料下载(电脑端登录)
章节2乳腺癌介绍和癌症常识
课时6乳腺癌-越来越年轻化癌症
课时7警钟长鸣!癌症就在你身边
课时8癌症科普介绍
课时9病毒细菌诱发的癌症
课时10祸从口入-致癌食物大揭秘
课时11Python机器学习挖掘癌细胞概述
章节3python编程环境搭建
课时12Anaconda快速入门指南
课时13Anaconda下载安装
课时14Canopy下载和安装
课时15Anaconda Navigator导航器
课时16Anaconda安装不同版本python
章节4python安装包
课时17python第三方包安装(pip和conda install)
课时18Python非官方扩展包下载地址
课时19清华大学开源镜像下载python第三方包
课时20pip install --user --upgrade package升级包
课时21pip install失败报错五种解决方案
章节5Jupiter Notebook概述
课时22jupyter1_为什么使用jupyter notebook
课时23jupyter2_jupyter基本文本编辑操作
课时24如何用jupyter notebook打开指定文件夹内容?
课时25jupyter4_jupyter转换PPT实操
课时26jupyter notebook用matplotlib不显示图片解决方案
章节6python基础知识
课时27为什么学习编程?大多数学校不会告诉你的秘密
课时28python官网02:12
课时29如何运用公开资料学习python(GitHub,kaggle,StackOverflow)
课时30Python文件基本操作
课时31变量_表达式_运算符_值
课时32字符串string
课时33程序基本构架(条件,循环)
课时34数据类型_函数_面向对象编程
课时35python2和3的区别
课时36编程技巧和学习方法
章节7python oop面向对象编程(选修)
课时37python面向对象编程1_类和实例
课时38python面向对象2_实例属性可以与众不同
课时39python面向对象3_self代表实例本身
课时40python面向对象编程4_init()初始化方法
课时41python面向对象5_属性和方法区别
课时42python面向对象6_函数和方法区别
课时43python面向对象7_类变量
课时44python面向对象8_父类和子类
课时45python面向对象9_私有变量
课时46python面向对象编程10_魔法函数__str__和__repr__
课时47python面向对象11运算符重载
章节8sklearn机器学习基础知识
课时48机器学习数据库介绍
课时49机器学习书籍推荐
课时50Python数据科学常用的包
课时51如何选择算法
课时52sklearn算法速查表
课时53sklearn建模基础代码
课时54python数据科学入门介绍(选修
章节9获取乳腺癌临床数据
课时55数据获取-乳腺癌细胞临床数据
章节10模型开发基础知识python脚本讲解
课时56数据读取read_excel和read_csv
课时57数据划分train_test_split
课时58LogisticRegression()模型构建和训练fit()
课时59模型预测predict和predict_proba区别
课时60模型验证的python脚本讲解
课时61pickle保存模型包
章节11数据预处理
课时62pandasl数据处理基础知识
课时63哑变量处理-hotcode热编码
课时64imputer-缺失数据处理
课时65scale-数据标准化处理
章节12变量筛选和描述性统计
课时66变量筛选1-模型法
课时67变量筛选2-比例法percentile
课时68变量筛选3-方差法
课时69变量筛选4-KBest
课时70好变量比算法更重要
课时71统计学随机抽样VS大数据
章节13十大经典机器学习算法-建立乳腺癌细胞分类器
课时72逻辑回归logistic regression
课时73支持向量SVM
课时74KNN最近邻算法
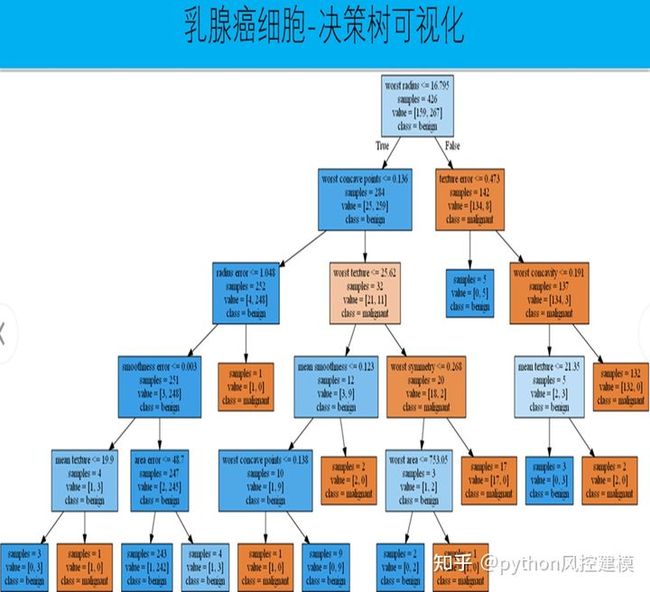
课时75决策树-decision tree
课时76随机森林-random forest
课时77神经网络neural network
课时78xgboost
课时79lightgbm基础讲解
课时80lightGBM脚本实现
课时81catboost基础讲解
课时82catboost脚本实现
课时83常见算法优劣对比
课时84bagging VS boosting
章节14变量(特征)重要性
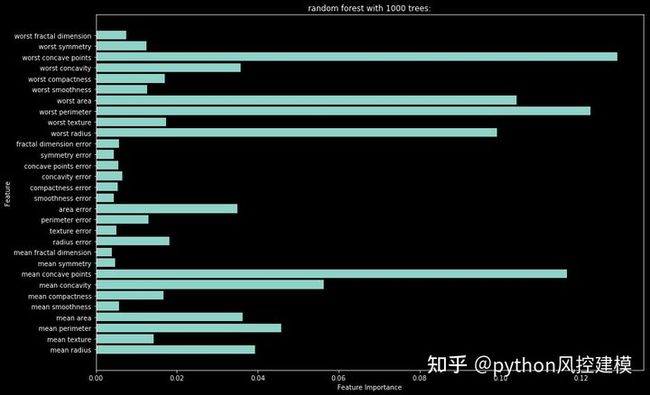
课时85逻辑回归和集成树算法变量(特征)重要性概述
课时86随机逻辑回归randomized logistic regression
课时87xgboost特征重要性
课时88catboost特征重要性
课时89lightgbm特征重要性
章节15降维1_主成分分析PCA
课时90边际效应基本概念
课时91模型维度与边际效应,变量越多越好吗?
课时92kaggle模型一定最好吗?降维在企业建模实际意义
课时93降维方法好不好,测试为准
课时94python主成分分析关键代码解读
课时95python主成分分析实战乳腺癌数据集
课时96PCA主成分降维在人脸识别应用-附代码(选修)
章节16降维2_因子分析factor analysis
课时97因子分析基本思想和使用限制条件
课时98python sklearn因子分析实战乳项目
课时99python因子分析第三方包训练模型,结果让人吃惊
课时100因子分析-KMO和巴特利球形度检验
课时101因子分析-python绘制碎石图
课时102因子分析旋转方法rotation
课时103因子分析-累计方差贡献率
课时104spss因子分析-解释癌细胞特征
章节17模型调参
课时105遍历调参法
课时106单个参数网格调参
课时107多参数网格调参
课时108随机网格调参_random size search cv
章节18模型验证
课时109模型验证必要性-市场80%模型存在问题
课时110交叉验证cross validation
课时111混淆矩阵
课时112ROC曲线
课时113PSI(population stability index)模型稳定性
课时114基尼系数GINI index-模型区分能力指标
课时115KS(kolmogorov-smirnoff)-模型区分能力指标
章节19非平衡样本数据imbalanced data
课时116非平衡数据是什么?对模型有什么坏处?
课时117解决非平衡数据方法(欠采样,过采样,SMOTE)
课时118SMOTE非平衡数据python脚本演示
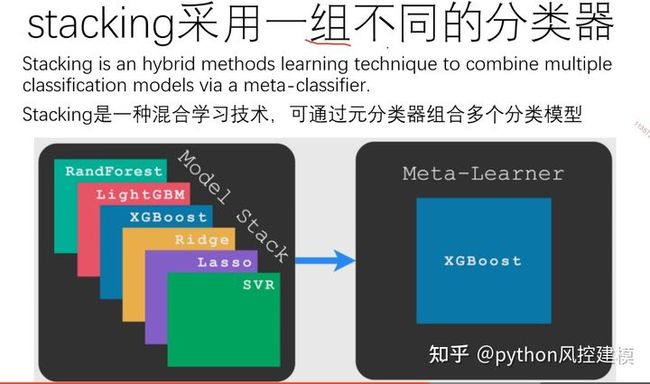
章节20stacking融合模型-kaggle夺冠大杀器
课时119stacking融合模型原理介绍
课时120stacking融合模型accuracy有显著提升
课时121stacking融合模型的AUC是否有提升?
课时122stacking融合模型f1分数显著提升
课时123stacking融合模型绘制决策域decision region
课时124使用概率值作为元-特征训练-模型性能得到第五次提升
课时125stacking融合模型和gridsearch网格调参
课时126stacking融合模型和机器学习管道pipeline综合应用
章节21Rdkit化学分子溶解度模型
课时127RDKIT概述-开源化学信息工具包
课时128Rdkit如何构建化学分子的溶解度预测模型
课时129conda-forge安装rdkit
课时130读取dat格式的化学分子式数据
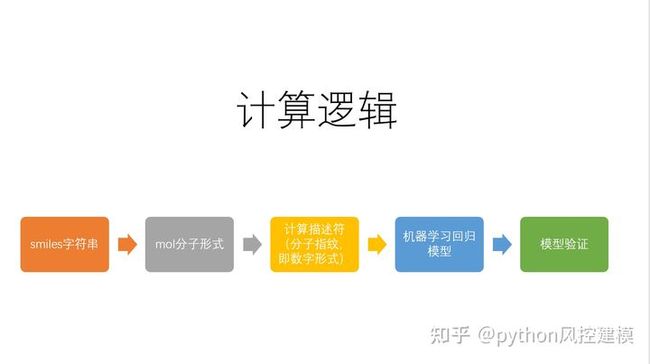
课时131smiles字符串形式转换为MOL分子式
课时132MOL分子结构转换为指纹数字形式
课时133随机森林和高斯算法建模
课时134rdkit本章节脚本和数据下载
章节22医药信息检索工具
课时135英语是获取专业信息钥匙
课时136WHO世界卫生组织
课时137FDA美国食药监局
课时138维基百科-开源检索工具
课时139TED-分享前言科技的平台
课时140医学美图
课时141中国疾控中心
课时142日本药典和马丁代尔药典介绍
课时143专业药品数据库查询网址
章节23附录
课时144致癌物分级列表(第四版)
课时145显微镜下癌细胞
课时146SIR模型预测新冠状病毒2019-nCoV
乳腺癌建模数据
课程中十大经典机器学习算法震撼登场:逻辑回归,支持向量,KNN,神经网络,随机森林,xgboost,lightGBM,catboost。课程提供视频里讲解脚本,这些模型脚本可以应用于各个领域数据,包括金融反欺诈模型,信用评分模型,收入预测模型等等,为中小企业提供现成解决方案。
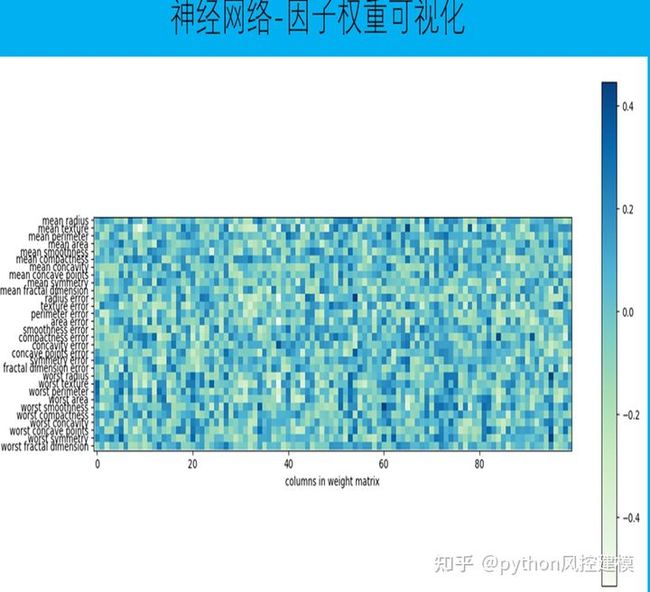
随机森林变量权重可视化
课程耗费三年时间,360度无死角的讲述整个模型开发周期,非市场上快餐教学。教程包括数据获取,数据预处理,变量筛选,模型筛选,模型评估,模型调参。
本视频系列通俗易懂,课程针对学生和科研机构,python爱好者。本视频教程系列有完整python代码,观众看后可以下载实际操作。这些模型代码可为中小型企业提供解决方案。
课程包含因子分析和主成分分析方法,对乳腺癌数据30个变量降维后建模,结果模型性能不仅没有下降,反而略有提升,可谓一石二鸟。
课程包含stacking融合模型方法,模型性能多次提升,是喜欢kaggle等模型竞赛学生一大福利。
下面是部分课程内容文字介绍
python机器学习编程环境搭建
python机器学习-乳腺癌细胞挖掘课程讲授初学者如何搭建python的Anaconda编程环境,Anaconda是一个集成数据科学编程框架,嵌入了sklearn,matplotlib,seaborn等常用机器学习和统计学包。
(1)下载anaconda
首先下载anaconda,这款框架比Python官网的编辑器更好用,下载网址为https://www.anaconda.com/download/
anaconda支持windows,linux,苹果操作系统
支持32位和64位操作系统
(2)导入sklearn第三方包
anaconda下载安装好后打开,自带sklearn第三方包
命令行输入import sklearn,无报错就表示运行正常
(3)pip install安装其他第三方包
机器学习中,有时候需要导入其他包,而sklearn没有,这时就需要用pip install安装其他第三方包
(4)非官方扩展包下载地址
有时候pip install安装失败,我们需要去欧文大学下载Python非官方扩展包
Python有大量非官方扩展包,应用于各行各业,主要是数据科学,人工智能,爬虫等等,下载地址为
https://www.lfd.uci.edu/~gohlke/pythonlibs/
乳腺癌细胞分类器建模
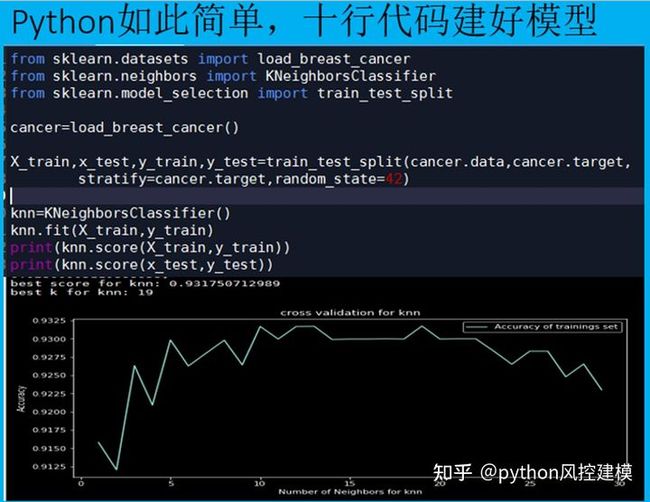
现在我们要用机器学习算法建立分类器,区分细胞为良性细胞或癌细胞。分类器就是解决二分类或多分类问题。
建立分类器算法很多,包括逻辑回归,xgboost,svm,神经网络等等。
开始编程:
在编写一行代码之前,我想做的第一件事是在代码的注释中加入描述。这样,我可以回顾我的代码并确切地知道它的作用
#Description: This program detects breast cancer, based off of data.现在导入包/库,以使其更容易编写程序。
#import libraries
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
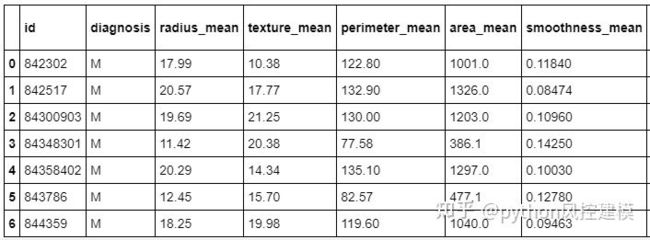
import seaborn as sns接下来,我将加载数据,并打印数据的前7行。
注意:每行数据代表可能患有或未患有癌症的患者。
#Load数据
#from google.colab 导入文件#用来加载数据在谷歌Colab #uploaded = files.upload() #使用在谷歌Colab负荷数据DF = pd.read_csv( 'data.csv')
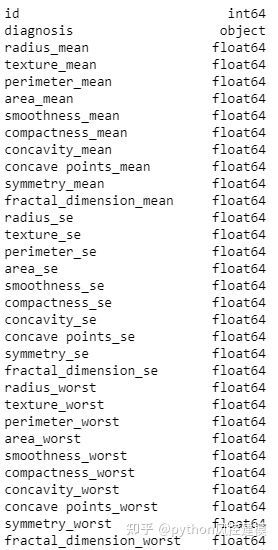
df.head (7)探索数据并计算数据集中的行数和列数。它们是569行数据,这意味着他们是该数据集中的569位患者,而33列则是每位患者的33个特征或数据点。
#计算数据集
df.shape中的行数和列数继续探索数据并获得包含空值(NaN,NAN,na)的所有列的计数。请注意,除了名为“ Unnamed:32 ”的列(其中包含569个空值)(数据集中的行数相同,这告诉我该列完全没有用)之外,所有列均未包含任何空值。
#计算每列
df.isna()。sum()中的空值(NaN,NAN,na)从原始数据集中删除“未命名:32 ”列,因为它没有任何值。
#Drop the column with all missing values (na, NAN, NaN)
df = df.dropna(axis=1)获取新的行和列数计数。
#Get the new count of the number of rows and cols
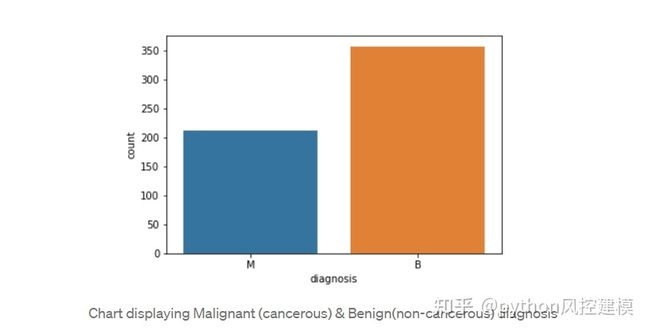
df.shape获取具有恶性(M)癌细胞和良性(B)非癌细胞的患者数。
#Get a count of the number of 'M' & 'B' cells
df['diagnosis'].value_counts()通过创建计数图可视化计数。
#Visualize this count
sns.countplot(df['diagnosis'],label="Count")查看数据类型以查看哪些列需要转换/编码。从数据类型中我可以看到,除“诊断”列外,所有列/功能都是数字,“诊断”列是在python中表示为对象的分类数据。
#Look at the data types
df.dtypes对分类数据进行编码。将“诊断”列中的值分别从M和B更改为1和0,然后打印结果。
#Encoding categorical data values (
from sklearn.preprocessing import LabelEncoder
labelencoder_Y = LabelEncoder()
df.iloc[:,1]= labelencoder_Y.fit_transform(df.iloc[:,1].values)
print(labelencoder_Y.fit_transform(df.iloc[:,1].values))创建一个对图。“对图”也称为散点图,其中同一数据行中的一个变量与另一变量的值匹配
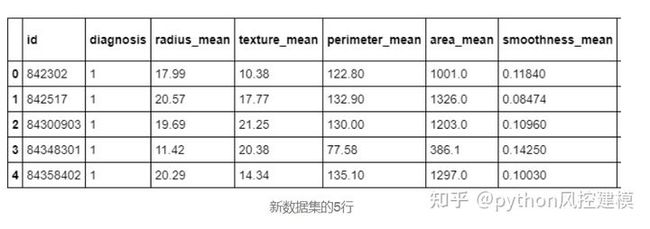
sns.pairplot(df,hue =“ diagnosis”)打印现在只有32列的新数据集。仅打印前5行。
df.head(5)获取列的相关性。
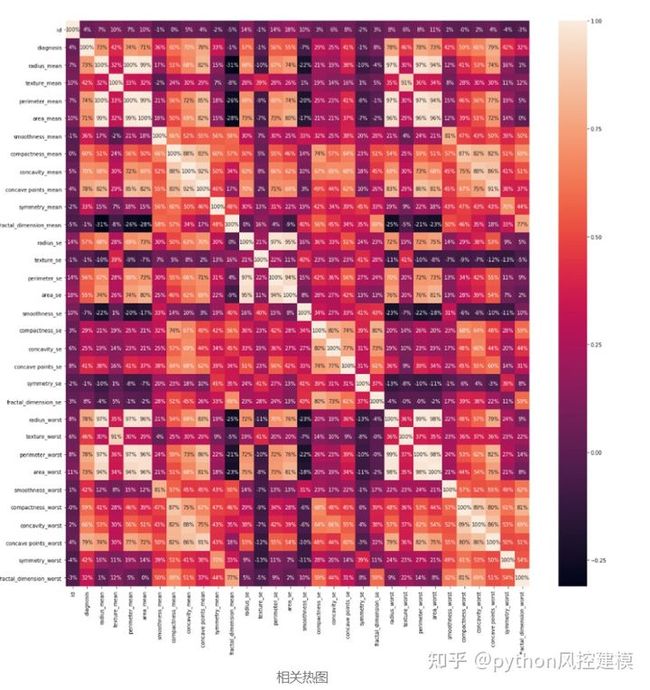
#Get the correlation of the columns
df.corr()列相关样本
通过创建热图可视化相关性。
plt.figure(figsize =(20,20))
sns.heatmap(df.corr(),annot = True,fmt ='。0%')现在,我完成了探索和清理数据的工作。我将通过首先将数据集分为特征数据集(也称为独立数据集(X))和目标数据集(也称为从属数据集(Y))来设置模型的数据。
X = df.iloc [:, 2:31] .values
Y = df.iloc [:, 1] .values再次拆分数据,但这一次分为75%的训练和25%的测试数据集。
from sklearn.model_selection import train_test_split
X_train,X_test,Y_train,Y_test = train_test_split(X,Y,test_size = 0.25,random_state = 0)缩放数据以使所有要素达到相同的大小级别,这意味着要素/独立数据将处于特定范围内,例如0-100或0-1。
#
from sklearn.preprocessing import StandardScaler
sc = StandardScaler()
X_train = sc.fit_transform(X_train)
X_test = sc.transform(X_test)创建一个函数以容纳许多不同的模型(例如,逻辑回归,决策树分类器,随机森林分类器)进行分类。这些模型将检测患者是否患有癌症。在此功能内,我还将在训练数据上打印每个模型的准确性。
def models(X_train,Y_train):
#Using Logistic Regression
from sklearn.linear_model import LogisticRegression
log = LogisticRegression(random_state = 0)
log.fit(X_train, Y_train)
#Using KNeighborsClassifier
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors = 5, metric = 'minkowski', p = 2)
knn.fit(X_train, Y_train)
#Using SVC linear
from sklearn.svm import SVC
svc_lin = SVC(kernel = 'linear', random_state = 0)
svc_lin.fit(X_train, Y_train)
#Using SVC rbf
from sklearn.svm import SVC
svc_rbf = SVC(kernel = 'rbf', random_state = 0)
svc_rbf.fit(X_train, Y_train)
#Using GaussianNB
from sklearn.naive_bayes import GaussianNB
gauss = GaussianNB()
gauss.fit(X_train, Y_train)
#Using DecisionTreeClassifier
from sklearn.tree import DecisionTreeClassifier
tree = DecisionTreeClassifier(criterion = 'entropy', random_state = 0)
tree.fit(X_train, Y_train)
#Using RandomForestClassifier method of ensemble class to use Random Forest Classification algorithm
from sklearn.ensemble import RandomForestClassifier
forest = RandomForestClassifier(n_estimators = 10, criterion = 'entropy', random_state = 0)
forest.fit(X_train, Y_train)
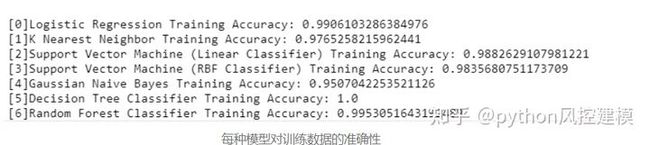
#print model accuracy on the training data.
print('[0]Logistic Regression Training Accuracy:', log.score(X_train, Y_train))
print('[1]K Nearest Neighbor Training Accuracy:', knn.score(X_train, Y_train))
print('[2]Support Vector Machine (Linear Classifier) Training Accuracy:', svc_lin.score(X_train, Y_train))
print('[3]Support Vector Machine (RBF Classifier) Training Accuracy:', svc_rbf.score(X_train, Y_train))
print('[4]Gaussian Naive Bayes Training Accuracy:', gauss.score(X_train, Y_train))
print('[5]Decision Tree Classifier Training Accuracy:', tree.score(X_train, Y_train))
print('[6]Random Forest Classifier Training Accuracy:', forest.score(X_train, Y_train))
return log, knn, svc_lin, svc_rbf, gauss, tree, forest创建包含所有模型的模型,并查看每个模型的训练数据上的准确性得分,以对患者是否患有癌症进行分类。
model = models(X_train,Y_train)catboost建模
今天我要介绍目前开源领域里最新的算法catboost。
catboost起源于俄罗斯搜索巨头yandex,准确率高,速度快,调参少,性价比高于xgboost
今天的CatBoost版本是第一个版本,以后将持续更新迭代。catboost三个特点:(1)“减少过度拟合”:这可以帮助你在训练计划中取得更好的成果。它基于一种构建模型的专有算法,这种算法与标准的梯度提升方案不同。(2)“类别特征支持”:这将改善你的训练结果,同时允许你使用非数字因素,“而不必预先处理数据,或花费时间和精力将其转化为数字。”(3)支持Python或R的API接口来使用CatBoost,包括公式分析和训练可视化工具。(4)有很多机器学习库的代码质量比较差,需要做大量的调优工作,”他说,“而CatBoost只需少量调试,就可以实现良好的性能。这是一个关键性的区别
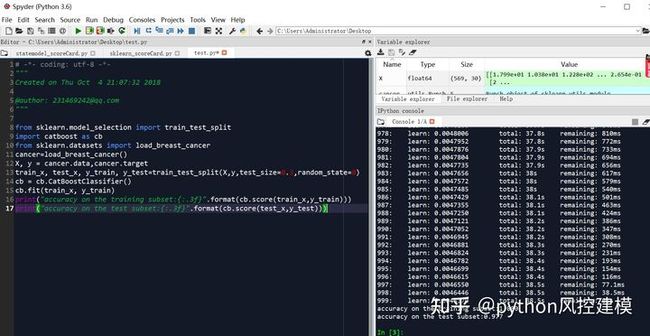
catboost建立乳腺癌分类器代码
#学习地址:https://ke.qq.com/course/3064292?tuin=dcbf0ba
from sklearn.model_selection import train_test_split
import catboost as cb
from sklearn.datasets import load_breast_cancer
cancer=load_breast_cancer()
X, y = cancer.data,cancer.target
train_x, test_x, y_train, y_test=train_test_split(X,y,test_size=0.3,random_state=0)
cb = cb.CatBoostClassifier()
cb.fit(train_x, y_train)
print("accuracy on the training subset:{:.3f}".format(cb.score(train_x,y_train)))
print("accuracy on the test subset:{:.3f}".format(cb.score(test_x,y_test)))大家可以看到catboost预测准确率非常高,训练集100%,测试集97.7%
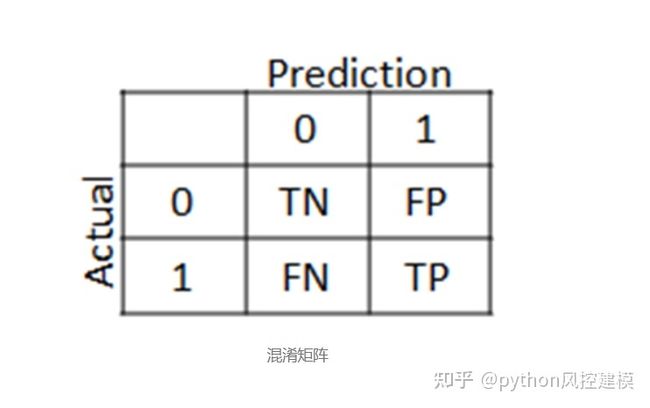
混淆矩阵
在测试数据上显示混淆矩阵和模型的准确性。该混淆矩阵告诉我们,每个模型有多少病人误诊(许多癌症患者是被误诊为不具有癌症又名假阴性,而谁没有癌症患者被误诊为患有癌症又名这个数字假阳性)和正确诊断的数量,真阳性和真阴性。
误报(FP) =测试结果错误地指示存在特定条件或属性。
真实阳性(TP) =灵敏度(在某些领域中也称为 真实阳性率或检测概率)衡量正确鉴定出的真实阳性的比例。
真实阴性(TN) = 特异性(也称为 真实阴性率)衡量正确鉴定出的实际 阴性的比例。
假阴性(FN) =测试结果,表明某个条件不成立,而实际上却成立。例如,测试结果表明某人实际患有癌症时没有罹患癌症
from sklearn.metrics import confusion_matrix
for i in range(len(model)):
cm = confusion_matrix(Y_test, model[i].predict(X_test))
TN = cm[0][0]
TP = cm[1][1]
FN = cm[1][0]
FP = cm[0][1]
print(cm)
print('Model[{}] Testing Accuracy = "{}!"'.format(i, (TP + TN) / (TP + TN + FN + FP)))
print()# Print a new line其他获取模型指标的方法,以查看每个模型的性能如何。
#Show other ways to get the classification accuracy & other metrics
from sklearn.metrics import classification_report
from sklearn.metrics import accuracy_score
for i in range(len(model)):
print('Model ',i)
#Check precision, recall, f1-score
print( classification_report(Y_test, model[i].predict(X_test)) )
#Another way to get the models accuracy on the test data
print( accuracy_score(Y_test, model[i].predict(X_test)))
print()#Print a new line模型预测
测试数据中1–6个性能指标的模型样本
从以上的准确性和指标来看,在测试数据上表现最佳的模型是随机森林分类器,其准确性得分约为96.5%。因此,我将选择该模型来检测患者的癌细胞。对测试数据进行预测/分类,并显示“随机森林分类器”模型分类/预测以及显示或不显示他们患有癌症的患者的实际值。
我注意到了该模型,该模型将一些患者误诊为没有癌症而误诊为癌症,并且将确诊为癌症的患者误诊为未患癌症。尽管此模型很好,但在处理他人的生活时,我希望该模型更好,并使其准确性尽可能接近100%,或者至少好于医生。因此,有必要对每个模型进行一些调整。
#Print随机森林分类器模型的预测
pred = model [6] .predict(X_test)
print(pred)
#
打印空间print()
#
打印实际值print(Y_test)Anaconda+KNN+网格调参+交叉验证
模型调参
python机器学习-乳腺癌细胞挖掘详细讲解模型调参技巧。调参是一门黑箱技术,需要经验丰富的机器学习工程师才能做到。幸运的是sklearn有调参的包,入门级学者也可尝试调参。
如果参数不多,可以手动写函数调参,如果参数太多可以用GridSearchCV调参,如果参数多的占用时间太长,可以用randomSizeCV调参,节约调参时间
GridSearchCV
如果参数太多可以用GridSearchCV调参
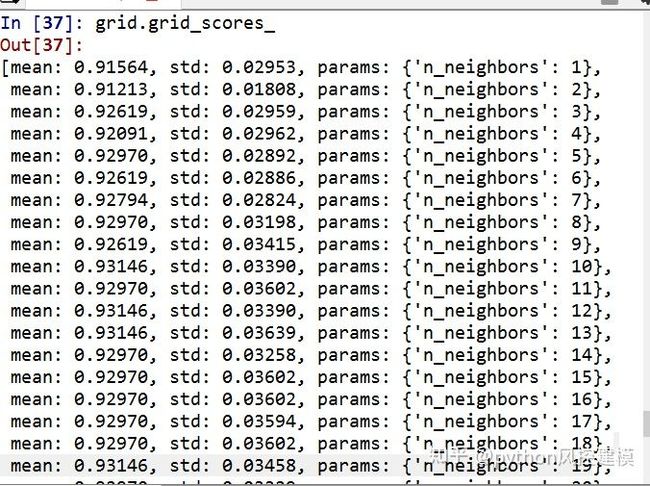
(1)单参数调参
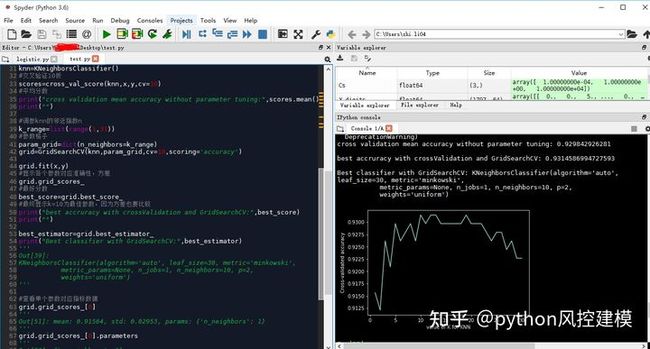
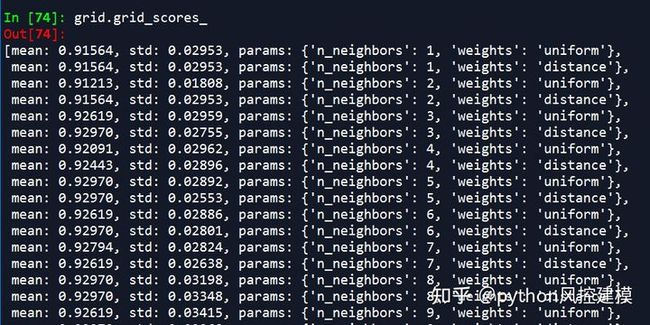
(2)多参数调参
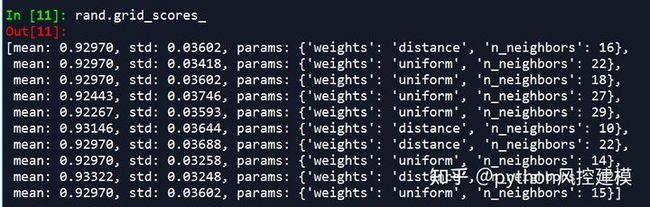
因为有n_neighbors和weights两个参数,因此诞生了60个结果
因为有两个参数,所以得到最佳模型:weight=distance,n_neighbor=12
RandomSizeSearchCV
randomSizeCV调参类似于GridSearchCV的抽样
如果参数多的占用时间太长,可以用randomSizeCV调参,节约调参时间。
randomSizeCV调参准确率会略低于GridSearchCV,但可以节约大量时间。
randomSizeCV调参代码
from sklearn.grid_search import RandomizedSearchCV
import matplotlib.pyplot as plt
#交叉验证
from sklearn.cross_validation import cross_val_score
from sklearn.datasets import load_breast_cancer
from sklearn.neighbors import KNeighborsClassifier
#导入数据
cancer=load_breast_cancer()
x=cancer.data
y=cancer.target
#调参knn的邻近指数n
k_range=list(range(1,31))
weight_options=['uniform','distance']
param_dist=dict(n_neighbors=k_range,weights=weight_options)
knn=KNeighborsClassifier()
#n_iter为随机生成个数
rand=RandomizedSearchCV(knn,param_dist,cv=10,scoring='accuracy',
n_iter=10,random_state=5)
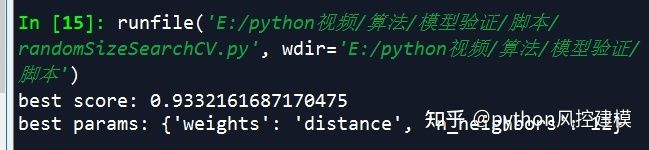
rand.fit(x,y)
rand.grid_scores_
print('best score:',rand.best_score_)
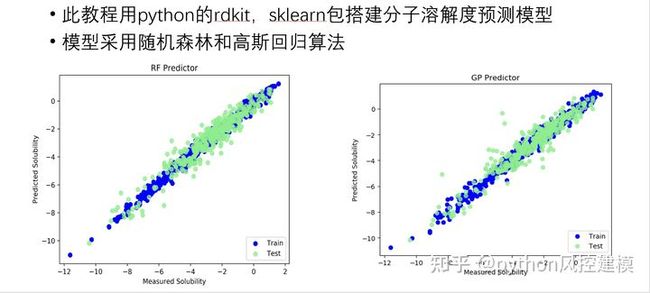
print('best params:',rand.best_params_)本课程还讲解了python的Rdkit包构建化学分子的溶解度预测模型
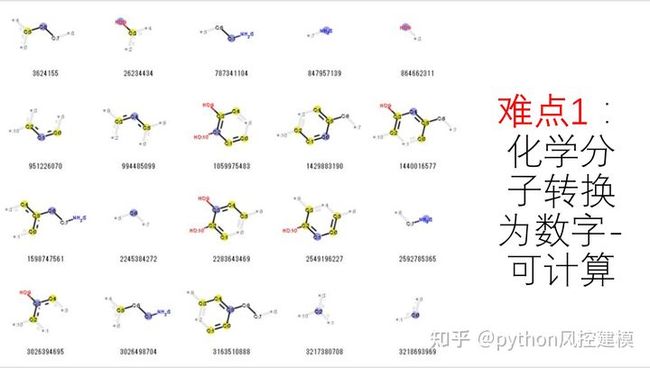
讲解RDKIT概述-RDKIT是一个开源化学信息工具包
强大的Rdikit,大家看看RDKIT对化学分子式完美操作。
关注公众号「python风控模型」,回复关键字:学习资料