- 【软件架构】DSA和ABSDM的区别及应用场景
小马哥编程
iSAQB软件架构架构系统架构软件工程开发语言设计模式
理解领域特定架构(DSA)和基于架构的软件开发方法(ABSDM)的区别及其应用场景,关键在于认清它们本质上是不同层次的概念:领域特定架构(Domain-SpecificArchitecture-DSA)是什么:DSA指的是一种针对特定应用领域(如电信、金融交易、图形处理、深度学习、汽车电子等)高度优化和定制化的软件架构设计。核心思想:领域聚焦:深刻理解特定领域的核心需求、约束(性能、功耗、实时性、
- 计算机毕业设计——springboot的准妈妈孕期交流平台
**欢迎来到琛哥的技术世界!**博主小档案:琛哥,一名来自世界500强的资深程序猿,毕业于国内知名985高校。技术专长:琛哥在深度学习任务中展现出卓越的能力,包括但不限于java、python等技术。近年来,琛哥更是将触角延伸至AI领域,对于机器学习、自然语言处理、智能推荐等前沿技术都有独到的见解和实践经验。博客亮点:琛哥坚信“授人以渔胜于授人以鱼”,因此我的博客中,你不仅可以找到关于技术的深入解
- TVP:用于高效二维时序视频定位的文本-视觉提示方法
AI专题精讲
强化学习强化学习文本视觉人工智能
温馨提示:本篇文章已同步至"AI专题精讲"TVP:用于高效二维时序视频定位的文本-视觉提示方法摘要本文研究的是时序视频定位(TemporalVideoGrounding,TVG)问题,其目标是在一段未经剪辑的长视频中,根据一条文本描述预测对应事件片段的起始和结束时间点。近年来,得益于精细的三维视觉特征,TVG技术取得了显著进展。然而,三维卷积神经网络(3DCNN)计算复杂度高,使得密集的3D视觉特
- 信而泰×DeepSeek:AI推理引擎驱动网络智能诊断迈向 “自愈”时代
DeepSeek-R1:强大的AI推理引擎底座DeepSeek是由杭州深度求索人工智能基础技术研究有限公司开发的新一代AI大模型。其核心优势在于强大的推理引擎能力,融合了自然语言处理(NLP)、深度学习、大规模数据分析等前沿技术。DeepSeek-R1具备卓越的逻辑推理、多模态分析(文本/图像/语音)和实时交互能力,能够高效处理代码生成、复杂问题求解、跨模态学习等高阶任务。凭借其开源、高效、多模态
- NLP论文速读|chameleon:一个即插即用的组合推理模块Plug-and-Play Compositional Reasoning with Large Language Models
Power2024666
NLP论文速读自然语言处理人工智能机器学习深度学习nlp语言模型
论文速读|Chameleon:Plug-and-PlayCompositionalReasoningwithLargeLanguageModels论文信息:简介:该论文介绍了一个名为Chameleon的人工智能系统,旨在解决大型语言模型(LLMs)在处理复杂推理任务时存在的固有限制,例如无法访问最新信息、使用外部工具以及执行精确的数学和逻辑推理。Chameleon通过插入即用模块增强LLMs,使其
- AI深度噪音抑制技术
这两年人工智能快速发展,AI已经渗透到了各行各业。在噪音抑制技术领域,AI也同样发挥了巨大的作用。AI深度噪音抑制技术是一种利用人工智能和深度学习算法来动态处理和减少音频信号中的噪声,从而提升音频的清晰度和质量。与传统的噪音抑制技术相比,AI深度噪音抑制能够更智能、更精准地分辨出背景噪音与有用的语音或音乐信号,尤其在复杂、多样的环境下表现尤为出色。1.工作原理AI深度噪音抑制技术基于深度神经网络(
- 【深度学习】softmax 回归的从零开始实现与简洁实现
Douglassssssss
深度学习深度学习回归人工智能softmax回归交叉熵损失函数
前言小时候听过一个小孩练琴的故事,老师让他先弹最简单的第一小节,小孩练了两天后弹不出。接着,老师让他直接去练更难的第二小节,小孩练习了几天后还是弹不出,开始感觉到挫败和烦躁了。小孩以为老师之后会让他从简单的开始练,谁知老师直接让他开始练最难的一小节。小孩不干了,问老师是不是故意刁难他。老师笑笑,让他现在弹弹第一小节试试。神奇的是,小孩竟然发现自己已经能完整弹出来了。这有点像我现在的学习状况,前些天
- 数据集标准化:软件2.0的基石工程
AI大模型应用之禅
javapythonjavascriptkotlingolang架构人工智能
数据集标准化,软件工程,数据质量,机器学习,人工智能,数据治理,数据可信度1.背景介绍在当今数据爆炸的时代,数据已成为企业和组织的核心资产。然而,海量的原始数据往往杂乱无章,格式不统一,质量参差不齐,这严重阻碍了数据价值的挖掘和应用。数据标准化作为解决这一问题的关键技术,已成为软件2.0时代不可或缺的基石工程。软件2.0时代,人工智能、机器学习等技术蓬勃发展,对数据质量提出了更高的要求。传统的软件
- 李开复:AI 2.0 时代的意义
AI大模型应用之禅
javapythonjavascriptkotlingolang架构人工智能
人工智能,深度学习,Transformer,大模型,通用人工智能,AI2.0,伦理问题,未来趋势1.背景介绍人工智能(AI)技术近年来发展迅速,从最初的局限于特定领域的应用,逐渐发展到能够处理更复杂的任务,甚至展现出一些类似人类智能的能力。2010年以来,深度学习技术的兴起,特别是Transformer模型的出现,为AI发展带来了新的突破。这些模型能够处理海量数据,学习复杂的模式,并在自然语言处理
- Python 现代时间序列预测第二版(五)
绝不原创的飞龙
默认分类默认分类
原文:annas-archive.org/md5/22eab741fce9c15dfad894ecf37bdd51译者:飞龙协议:CCBY-NC-SA4.0第十七章:概率预测及更多在整本书中,我们学习了生成预测的不同技术,包括一些经典方法,使用机器学习以及一些深度学习架构。但我们一直在关注一种典型的预测问题——为连续时间序列生成点预测,并且没有层级关系且历史数据足够丰富。我们之所以这样做,是因为这
- 人的价值就是能让别人生活得更有价值——读荆志强老师的书有感
花婆婆
最近读了荆志强老师的专著《快乐地做教育》,前言就提到:教育的本真,就是通过激励为学生赋能,帮助学生产生足够的“自我效能感”,让学生主动获取知识技能、培育情感态度;帮助学生建立自主深度学习的能力。荆志强老师倡导的“赋能教育”主要从两个方面建构:激励型课堂、自主化管理。其中,他还谈到生本教育的学习观:学习是生命成长的过程,它是人自身的一种需要,而不是外在压力的结果。教育的一切行为都应该是为了满足儿童的
- ChatGPT 之后的下一步是什么?四个迫在眉睫的进步
iCloudEnd
OpenAI的文本生成器ChatGPT进入公共领域已经两个半月了,该机器人令人印象深刻且深思熟虑的答案已经引起了1300万日常用户的注意,他们已经将其用于一般问题、开发想法和写作全长文章。尽管ChatGPT存在重大缺陷(例如一些事实不准确),但许多人担心这可能对劳动力、学校、新闻业等产生影响。然而,我们可能正处于一场巨大的人工智能革命的开端。OpenAI总裁兼联合创始人GregBrockman于1
- 喜爱购有什么新消息?如何打造百城万店
氧惠好物
自2020年10月起,西安喜爱购商贸商贸股份有限公司全力打造的“百城万店”新零售商业模式应运而生。在探索新零售的道路上,通过互联网、大数据、云计算、人工智能等新技术,重构“人、货、场”商业元素,秉持“舍利差赚服务”经营理念,在全国至少一百个城市的“一千户以上的中高端社区”,打造至少两万家“一区一店”社区生活超市。大家好!我是氧惠最大团队&联合创始人氧惠达人导师。氧惠佣金更高,模式更好,终端用户不流
- AI 驱动自动化运维平台架构与实现
大富大贵7
程序员知识储备1程序员知识储备2程序员知识储备3算法机器学习人工智能决策树大数据
摘要:随着云计算、容器化和大规模分布式系统的普及,传统人工运维方法已难以满足现代IT环境中海量指标、日志和拓扑关系的实时分析与故障响应需求。AI驱动的自动化运维(AIOps)平台通过融合机器学习、深度学习、图分析以及强化学习等多学科技术,实现对海量运维数据的智能感知、预测、诊断和自动化修复。本文深入探讨AI驱动自动化运维平台的整体架构设计与核心技术实现,涵盖数据采集与预处理、AI引擎设计、自动化执
- 冒充顺华文庭内部群胜天半子毛顺华就是骗子,中粮仓智慧农业虚拟盘及早远离切勿被套!
昌龙律法
人到老年,就怕手头没钱。一些不法分子利用老年人信息闭塞、认知较弱等特点瞄准了老年人的“钱袋子”花样百出实施诈骗老年人损失财产的同时还饱受精神打击不能忍!这些套路,应该让爸妈知道智慧农业,低碳环保双探交易市场,数字体育,人工智能十选五就是骗局我们曾曝光了无数种金融骗局,不知道能有多少人看到,能帮一个是一个,再次曝光一种炒股诱导做慈善参加数字经济的骗局,相信作为股民,大家都会经常接到一下分析个股,或者
- 人工智能真的能编程吗?研究勾勒出自主软件工程的障碍
WSSWWWSSW
人工智能软件工程
想象一下这样一个未来:人工智能悄然承担起软件开发的繁重工作:重构杂乱无章的代码、迁移遗留系统以及排查竞态条件,这样人类工程师就可以专注于架构、设计以及那些机器仍然无法解决的真正新颖的问题。最近的进展似乎让这个未来近在咫尺,但麻省理工学院计算机科学与人工智能实验室(CSAIL)以及其他几家合作机构的研究人员发表的一篇新论文指出,要实现这个潜在的未来,需要认真审视当前面临的挑战。这篇题为《面向软件工程
- GPU 之后,IMU 登场:AI 发展的下一次飞跃
你早晨醒来,手机上的大模型帮你写完邮件、翻译合同,却依旧不能帮你把厨房里洒掉的牛奶擦干。你戴上的AR眼镜知道“那里有杯子”,却抓不到它——AI会说不会做。是不是哪里少了一截?人工智能(AI)的发展历程中,我们见证了从简单的数据处理到复杂的语言生成能力的飞跃。然而,尽管AI在虚拟世界中表现出色,它在物理世界中的表现却相对滞后。为了填补这一空白,AI正在进入一个新的发展阶段:行动驱动时代。在本文中,我
- YOLOv13_SSOD:基于超图关联增强的半监督目标检测框架(原创创新算法)
YOLOv13_SSOD:基于超图关联增强的半监督目标检测框架项目背景随着深度学习技术的快速发展,目标检测在各个领域都取得了显著的进展。然而,现有的监督学习方法在实际应用中面临着标注数据稀缺、泛化能力不足等挑战。特别是在火灾烟雾检测、工业质检等特定场景中,获取大量高质量标注数据的成本极高。为了解决这一问题,本项目基于最新发布的YOLOv13架构,结合EfficientTeacher半监督学习框架,
- USB串口通信、握手协议、深度学习等技术要点
深度学习教程,
深度学习人工智能网络协议
基于OpenMV的智能车牌识别系统:从硬件到算法的完整实现前言本文将详细介绍一个基于OpenMV微控制器的智能车牌识别系统的设计与实现。该系统集成了嵌入式视觉处理、串口通信协议、深度学习OCR识别等多种技术,实现了从图像采集到车牌识别的完整流程。系统架构概述整体设计思路该车牌识别系统采用分布式架构设计,将计算密集型任务与嵌入式控制分离:┌─────────────┐USB串口通信┌────────
- python学习路线(从菜鸟到起飞)
突突突然不会编了
python学习开发语言
以下是基于2025年最新技术趋势的Python学习路线,综合多个权威资源整理而成,涵盖从零基础到进阶应用的全流程,适合不同学习目标(如Web开发、数据分析、人工智能等)的学习者。路线分为基础、进阶、实战、高级、方向拓展五个阶段,并附学习资源推荐:一、基础阶段(1-2个月)目标:掌握Python核心语法与编程思维,熟悉开发环境。环境搭建安装Python3.10+,配置PyCharm或VSCode开发
- 语音识别开源项目推荐:GitHub热门仓库盘点
AGI大模型与大数据研究院
AI大模型应用开发实战语音识别开源githubai
2024年必看!GitHub热门语音识别开源项目全解析:从入门到实战关键词语音识别(ASR)、开源项目、GitHub、Whisper、FunASR、PaddleSpeech、深度学习摘要想象一下:开车时只需说一句话就能自动发消息,听英文演讲时实时获得中文翻译,给视障人士读文本时精准转换——这些场景的背后,语音识别(AutomaticSpeechRecognition,ASR)技术正在改变我们与机器
- Python训练 + Go优化 + C#部署:端到端AI模型的跨语言实践
威哥说编程
人工智能学习资料库pythongolangc#
在现代AI应用中,如何高效地训练、优化、并最终部署AI模型是一项复杂且具有挑战性的任务。在这一过程中,选择合适的编程语言和工具可以显著提高效率和系统的性能。Python作为AI领域的主流语言,具有丰富的深度学习框架(如PyTorch和TensorFlow),在模型训练方面处于领先地位。然而,针对计算密集型任务(如数据预处理、加密等),Go语言因其高效的并发处理和出色的性能,成为优化计算的理想选择。
- yolov8涨点系列之替换幽灵卷积GhostConv
没脾气的小玩家
yolov8涨点系列YOLO目标检测
文章目录核心思想主要步骤优势yolov8.yaml文件增加CBAMyolov8.yamlyolov8.yaml将Conv卷积替换成GhostConv 幽灵卷积(GhostConv)是一种新颖的卷积操作方法,旨在解决传统卷积神经网络中参数量和计算量过大的问题,尤其适用于资源受限的设备。以下是对幽灵卷积的详细介绍:核心思想 常规的卷积操作会产生大量的特征图,其中存在一定的冗余信息。幽灵卷积的核心思
- DL00478-涡轮叶片缺陷检测数据集yolo格式1300张左右
涡轮叶片缺陷检测数据集yolo格式1300张左右涡轮叶片缺陷检测数据集YOLO格式解析:提升研究与论文写作的关键要点在研究涡轮叶片缺陷检测的过程中,数据集的选择和格式处理是一个至关重要的环节。特别是当你打算通过卷积神经网络(CNN)等深度学习模型进行缺陷检测时,数据集的标注和格式化直接影响到模型的训练效果和论文的质量。本文将重点探讨涡轮叶片缺陷检测数据集的YOLO格式,并分析如何利用这一格式为研究
- 京东零售重磅开源 | OxyGent:像搭乐高一样组装AI团队,实现群体智能
京东零售技术
零售开源人工智能
京东零售Oxygen团队正式开源发布多智能体协作框架——OxyGent。这一创新框架致力于帮助开发者高效组装多智能体协作系统,实现智能体间的无缝协作、弹性扩展与全链路可追溯。推动人工智能从“单点突破”迈向“群体智能”时代。OxyGent已在开源社区正式上线。开源地址:https://github.com/jd-opensource/OxyGent官网地址:https://oxygent.jd.co
- 具身智能的视觉-语言导航综述
24年2月来自曲阜师范、华东师大和哈工大的论文“Vision-LanguageNavigationwithEmbodiedIntelligence:ASurvey”。作为人工智能领域的长期愿景,具身智能的核心目标是提升智体与环境的感知、理解和交互能力。视觉-语言导航(VLN)作为实现具身智能的重要研究路径,致力于探索智体如何利用自然语言与人进行有效沟通,接收并理解指令,并最终依靠视觉信息实现精准导
- 具身智能:从理论到实践的跨越
具身智能(EmbodiedAI)的概念起源与发展是一个跨越半个多世纪的学术探索历程,其核心思想在不同学科的交叉碰撞中逐渐成型。以下从理论源头、技术奠基、术语演进三个维度展开解析,揭示这一概念的学术脉络与产业价值:一、理论源头:从图灵的哲学构想到认知科学的具身化转向1.图灵的"感官机器"设想(1950年)在人工智能奠基性论文《计算机器与智能》中,图灵提出了两种智能发展路径:抽象计算路径:如国际象棋等
- Epoch
老兵发新帖
人工智能
在深度学习和机器学习中,Epoch(轮次或周期)是一个核心训练概念,指模型在整个训练数据集上完成一次完整遍历的过程。以下是关于Epoch的详细解析:一、核心定义基本含义Epoch表示模型将所有训练数据完整学习一次的过程。例如:若训练集有10,000个样本,则1个Epoch即模型用这10,000个样本训练一轮。与相关概念的关系Batch(批次):数据集被分割成的小组(如每批32个样本)。Iterat
- 生命3.0时代,面对人工智能时代的到来,我们可以做些什么
笃定的沙丁鱼
生命的定义生命的定义有很多,最为人所熟知的是在生物学上的定义,即生命是蛋白质存在的一种形式。但是,这种定义可能不太适用于未来的智能机器和外星文明,我们不能将我们对未来生命的思考局限在过去遇到过的物种,所以需要将生命定义得更广阔一些:生命是一个能保持自身复杂性并能进行复制的过程。复制的对象并不是由原子组成的物质,而是能阐明原子是如何排列的信息,这种信息由比特组成。换句话说:我们可以将生命看作一种自我
- 深度学习图像分类数据集—百种病虫害分类
AI街潜水的八角
深度学习图像数据集深度学习分类人工智能
该数据集为图像分类数据集,适用于ResNet、VGG等卷积神经网络,SENet、CBAM等注意力机制相关算法,VisionTransformer等Transformer相关算法。数据集信息介绍:百种病虫害识别分类,训练集45095张,验证集7508张,测试集22619张具体类别为以下:insect_classes=["rice_leaf_roller","rice_leaf_caterpillar
- Java 并发包之线程池和原子计数
lijingyao8206
Java计数ThreadPool并发包java线程池
对于大数据量关联的业务处理逻辑,比较直接的想法就是用JDK提供的并发包去解决多线程情况下的业务数据处理。线程池可以提供很好的管理线程的方式,并且可以提高线程利用率,并发包中的原子计数在多线程的情况下可以让我们避免去写一些同步代码。
这里就先把jdk并发包中的线程池处理器ThreadPoolExecutor 以原子计数类AomicInteger 和倒数计时锁C
- java编程思想 抽象类和接口
百合不是茶
java抽象类接口
接口c++对接口和内部类只有简介的支持,但在java中有队这些类的直接支持
1 ,抽象类 : 如果一个类包含一个或多个抽象方法,该类必须限定为抽象类(否者编译器报错)
抽象方法 : 在方法中仅有声明而没有方法体
package com.wj.Interface;
- [房地产与大数据]房地产数据挖掘系统
comsci
数据挖掘
随着一个关键核心技术的突破,我们已经是独立自主的开发某些先进模块,但是要完全实现,还需要一定的时间...
所以,除了代码工作以外,我们还需要关心一下非技术领域的事件..比如说房地产
&nb
- 数组队列总结
沐刃青蛟
数组队列
数组队列是一种大小可以改变,类型没有定死的类似数组的工具。不过与数组相比,它更具有灵活性。因为它不但不用担心越界问题,而且因为泛型(类似c++中模板的东西)的存在而支持各种类型。
以下是数组队列的功能实现代码:
import List.Student;
public class
- Oracle存储过程无法编译的解决方法
IT独行者
oracle存储过程
今天同事修改Oracle存储过程又导致2个过程无法被编译,流程规范上的东西,Dave 这里不多说,看看怎么解决问题。
1. 查看无效对象
XEZF@xezf(qs-xezf-db1)> select object_name,object_type,status from all_objects where status='IN
- 重装系统之后oracle恢复
文强chu
oracle
前几天正在使用电脑,没有暂停oracle的各种服务。
突然win8.1系统奔溃,无法修复,开机时系统 提示正在搜集错误信息,然后再开机,再提示的无限循环中。
无耐我拿出系统u盘 准备重装系统,没想到竟然无法从u盘引导成功。
晚上到外面早了一家修电脑店,让人家给装了个系统,并且那哥们在我没反应过来的时候,
直接把我的c盘给格式化了 并且清理了注册表,再装系统。
然后的结果就是我的oracl
- python学习二( 一些基础语法)
小桔子
pthon基础语法
紧接着把!昨天没看继续看django 官方教程,学了下python的基本语法 与c类语言还是有些小差别:
1.ptyhon的源文件以UTF-8编码格式
2.
/ 除 结果浮点型
// 除 结果整形
% 除 取余数
* 乘
** 乘方 eg 5**2 结果是5的2次方25
_&
- svn 常用命令
aichenglong
SVN版本回退
1 svn回退版本
1)在window中选择log,根据想要回退的内容,选择revert this version或revert chanages from this version
两者的区别:
revert this version:表示回退到当前版本(该版本后的版本全部作废)
revert chanages from this versio
- 某小公司面试归来
alafqq
面试
先填单子,还要写笔试题,我以时间为急,拒绝了它。。时间宝贵。
老拿这些对付毕业生的东东来吓唬我。。
面试官很刁难,问了几个问题,记录下;
1,包的范围。。。public,private,protect. --悲剧了
2,hashcode方法和equals方法的区别。谁覆盖谁.结果,他说我说反了。
3,最恶心的一道题,抽象类继承抽象类吗?(察,一般它都是被继承的啊)
4,stru
- 动态数组的存储速度比较 集合框架
百合不是茶
集合框架
集合框架:
自定义数据结构(增删改查等)
package 数组;
/**
* 创建动态数组
* @author 百合
*
*/
public class ArrayDemo{
//定义一个数组来存放数据
String[] src = new String[0];
/**
* 增加元素加入容器
* @param s要加入容器
- 用JS实现一个JS对象,对象里有两个属性一个方法
bijian1013
js对象
<html>
<head>
</head>
<body>
用js代码实现一个js对象,对象里有两个属性,一个方法
</body>
<script>
var obj={a:'1234567',b:'bbbbbbbbbb',c:function(x){
- 探索JUnit4扩展:使用Rule
bijian1013
java单元测试JUnitRule
在上一篇文章中,讨论了使用Runner扩展JUnit4的方式,即直接修改Test Runner的实现(BlockJUnit4ClassRunner)。但这种方法显然不便于灵活地添加或删除扩展功能。下面将使用JUnit4.7才开始引入的扩展方式——Rule来实现相同的扩展功能。
1. Rule
&n
- [Gson一]非泛型POJO对象的反序列化
bit1129
POJO
当要将JSON数据串反序列化自身为非泛型的POJO时,使用Gson.fromJson(String, Class)方法。自身为非泛型的POJO的包括两种:
1. POJO对象不包含任何泛型的字段
2. POJO对象包含泛型字段,例如泛型集合或者泛型类
Data类 a.不是泛型类, b.Data中的集合List和Map都是泛型的 c.Data中不包含其它的POJO
- 【Kakfa五】Kafka Producer和Consumer基本使用
bit1129
kafka
0.Kafka服务器的配置
一个Broker,
一个Topic
Topic中只有一个Partition() 1. Producer:
package kafka.examples.producers;
import kafka.producer.KeyedMessage;
import kafka.javaapi.producer.Producer;
impor
- lsyncd实时同步搭建指南——取代rsync+inotify
ronin47
1. 几大实时同步工具比较 1.1 inotify + rsync
最近一直在寻求生产服务服务器上的同步替代方案,原先使用的是 inotify + rsync,但随着文件数量的增大到100W+,目录下的文件列表就达20M,在网络状况不佳或者限速的情况下,变更的文件可能10来个才几M,却因此要发送的文件列表就达20M,严重减低的带宽的使用效率以及同步效率;更为要紧的是,加入inotify
- java-9. 判断整数序列是不是二元查找树的后序遍历结果
bylijinnan
java
public class IsBinTreePostTraverse{
static boolean isBSTPostOrder(int[] a){
if(a==null){
return false;
}
/*1.只有一个结点时,肯定是查找树
*2.只有两个结点时,肯定是查找树。例如{5,6}对应的BST是 6 {6,5}对应的BST是
- MySQL的sum函数返回的类型
bylijinnan
javaspringsqlmysqljdbc
今天项目切换数据库时,出错
访问数据库的代码大概是这样:
String sql = "select sum(number) as sumNumberOfOneDay from tableName";
List<Map> rows = getJdbcTemplate().queryForList(sql);
for (Map row : rows
- java设计模式之单例模式
chicony
java设计模式
在阎宏博士的《JAVA与模式》一书中开头是这样描述单例模式的:
作为对象的创建模式,单例模式确保某一个类只有一个实例,而且自行实例化并向整个系统提供这个实例。这个类称为单例类。 单例模式的结构
单例模式的特点:
单例类只能有一个实例。
单例类必须自己创建自己的唯一实例。
单例类必须给所有其他对象提供这一实例。
饿汉式单例类
publ
- javascript取当月最后一天
ctrain
JavaScript
<!--javascript取当月最后一天-->
<script language=javascript>
var current = new Date();
var year = current.getYear();
var month = current.getMonth();
showMonthLastDay(year, mont
- linux tune2fs命令详解
daizj
linuxtune2fs查看系统文件块信息
一.简介:
tune2fs是调整和查看ext2/ext3文件系统的文件系统参数,Windows下面如果出现意外断电死机情况,下次开机一般都会出现系统自检。Linux系统下面也有文件系统自检,而且是可以通过tune2fs命令,自行定义自检周期及方式。
二.用法:
Usage: tune2fs [-c max_mounts_count] [-e errors_behavior] [-g grou
- 做有中国特色的程序员
dcj3sjt126com
程序员
从出版业说起 网络作品排到靠前的,都不会太难看,一般人不爱看某部作品也是因为不喜欢这个类型,而此人也不会全不喜欢这些网络作品。究其原因,是因为网络作品都是让人先白看的,看的好了才出了头。而纸质作品就不一定了,排行榜靠前的,有好作品,也有垃圾。 许多大牛都是写了博客,后来出了书。这些书也都不次,可能有人让为不好,是因为技术书不像小说,小说在读故事,技术书是在学知识或温习知识,有
- Android:TextView属性大全
dcj3sjt126com
textview
android:autoLink 设置是否当文本为URL链接/email/电话号码/map时,文本显示为可点击的链接。可选值(none/web/email/phone/map/all) android:autoText 如果设置,将自动执行输入值的拼写纠正。此处无效果,在显示输入法并输
- tomcat虚拟目录安装及其配置
eksliang
tomcat配置说明tomca部署web应用tomcat虚拟目录安装
转载请出自出处:http://eksliang.iteye.com/blog/2097184
1.-------------------------------------------tomcat 目录结构
config:存放tomcat的配置文件
temp :存放tomcat跑起来后存放临时文件用的
work : 当第一次访问应用中的jsp
- 浅谈:APP有哪些常被黑客利用的安全漏洞
gg163
APP
首先,说到APP的安全漏洞,身为程序猿的大家应该不陌生;如果抛开安卓自身开源的问题的话,其主要产生的原因就是开发过程中疏忽或者代码不严谨引起的。但这些责任也不能怪在程序猿头上,有时会因为BOSS时间催得紧等很多可观原因。由国内移动应用安全检测团队爱内测(ineice.com)的CTO给我们浅谈关于Android 系统的开源设计以及生态环境。
1. 应用反编译漏洞:APK 包非常容易被反编译成可读
- C#根据网址生成静态页面
hvt
Web.netC#asp.nethovertree
HoverTree开源项目中HoverTreeWeb.HVTPanel的Index.aspx文件是后台管理的首页。包含生成留言板首页,以及显示用户名,退出等功能。根据网址生成页面的方法:
bool CreateHtmlFile(string url, string path)
{
//http://keleyi.com/a/bjae/3d10wfax.htm
stri
- SVG 教程 (一)
天梯梦
svg
SVG 简介
SVG 是使用 XML 来描述二维图形和绘图程序的语言。 学习之前应具备的基础知识:
继续学习之前,你应该对以下内容有基本的了解:
HTML
XML 基础
如果希望首先学习这些内容,请在本站的首页选择相应的教程。 什么是SVG?
SVG 指可伸缩矢量图形 (Scalable Vector Graphics)
SVG 用来定义用于网络的基于矢量
- 一个简单的java栈
luyulong
java数据结构栈
public class MyStack {
private long[] arr;
private int top;
public MyStack() {
arr = new long[10];
top = -1;
}
public MyStack(int maxsize) {
arr = new long[maxsize];
top
- 基础数据结构和算法八:Binary search
sunwinner
AlgorithmBinary search
Binary search needs an ordered array so that it can use array indexing to dramatically reduce the number of compares required for each search, using the classic and venerable binary search algori
- 12个C语言面试题,涉及指针、进程、运算、结构体、函数、内存,看看你能做出几个!
刘星宇
c面试
12个C语言面试题,涉及指针、进程、运算、结构体、函数、内存,看看你能做出几个!
1.gets()函数
问:请找出下面代码里的问题:
#include<stdio.h>
int main(void)
{
char buff[10];
memset(buff,0,sizeof(buff));
- ITeye 7月技术图书有奖试读获奖名单公布
ITeye管理员
活动ITeye试读
ITeye携手人民邮电出版社图灵教育共同举办的7月技术图书有奖试读活动已圆满结束,非常感谢广大用户对本次活动的关注与参与。
7月试读活动回顾:
http://webmaster.iteye.com/blog/2092746
本次技术图书试读活动的优秀奖获奖名单及相应作品如下(优秀文章有很多,但名额有限,没获奖并不代表不优秀):
《Java性能优化权威指南》

![]() 表示第k个DC-CNN块中的卷积层C生成的特征图。这样,将多个特征映射连接起来,然后通过一个
表示第k个DC-CNN块中的卷积层C生成的特征图。这样,将多个特征映射连接起来,然后通过一个![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第1张图片](http://img.e-com-net.com/image/info8/741c8f65ec374b719bdc4aa72cefb3eb.jpg)
 q表示要预测的类别数,
q表示要预测的类别数,![]() 介于 0 和 1 之间。
介于 0 和 1 之间。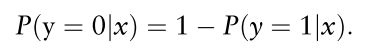

![]()
![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第2张图片](http://img.e-com-net.com/image/info8/7944bf18ef644b9386ed10aa174b3918.jpg)
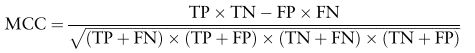
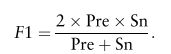
![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第3张图片](http://img.e-com-net.com/image/info8/0a45dde7b85b4224a4b0880b8ed14fa5.jpg)
![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第4张图片](http://img.e-com-net.com/image/info8/64a8b1846d694617920255352d4536fa.jpg)
![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第6张图片](http://img.e-com-net.com/image/info8/815272993f754e75b7035e7cb7f407df.jpg)
![[论文阅读]使用深度学习方法预测蛋白质磷酸化位点DeepPhos: prediction of protein phosphorylation sites with deep learning(二)_第5张图片](http://img.e-com-net.com/image/info8/00d191b7806a4ced8d29e27b8ed12263.jpg)