农作物病虫害识别技术的发展综述
最近读了Recent advances in image processing techniques for automated leaf pest and disease recognition – A review这篇文献之后,让我对农作物的病虫害识别有了一个更全面、更深入的理解了。下面就让我们走进这篇文献:
摘要
快速准确的植物病虫害检测对提高农业生产率是特别重要的,传统上依赖于人类专家来诊断由疾病、害虫、营养缺乏或极端天气引起的植物异常。然而这样是昂贵、费时且不切实际的。为了应对这些挑战,利用图像处理技术进行植物病虫害识别已经成为了一个热门的研究课题。//这篇文章中,总结了用于农作物病虫害识别的图像处理和机器学习技术。//文中显示了10多种神经网络结构在识别准确率、召回率、准确率、特异性、F1-评分、训练时长和存储要求等方面的识别性能。//随后提出了在传统以及移动/嵌入式计算机环境中部署的最合适架构的建议。//还讨论了一些尚未解决的挑战,这些挑战需要解决,以便开发实用的自动植物病害识别系统,用于野外条件。
1.图像处理技术(IPT)在农作物病虫害识别中的应用
感染疾病的植物一般会出现明显的标记或者损伤。专业人员一般通过目视检查或对植物样本进行实验室测试来诊断,但是这些方法具有一定的局限性:
- 对疾病的诊断都是需要专业知识的,一般的农民可能没有具备相应的知识进行诊断;
- 专业诊断人员的训练是耗时且昂贵的;
- 农民和专业人员可能不能正确的识别非本地的病虫害;
- 对于一些具有视觉上相似特征的病虫害需要高水平的专业知识,这种情况下,即使是专业人员也有可能会因为疲劳、照明不足、视力不好而做出错误的诊断。另外,个别专家是一小组疾病的专家;
- 持续监测对于早期疾病检测和防止疾病传播是必要的,这是乏味的、耗时的、昂贵的和低效的;
- 除了实验室设备的高成本以外,实验室测试也是具有破坏性的。因为它们涉及到从野外收集植物样本并将其带到实验室进行分析。此外,将样品运送到实验室可能也会受到检疫限制。
IPT(图像处理技术)的优点:
- 基于作物叶、茎、花和果实的图像,可以快速而准确地识别作物疾病;
- 疾病的严重程度可以通过计算变形或者变色区域相对于整个叶子、果实或者花的大小来估计;
- 跟踪植物的疾病进程,这对于检测感染阶段等细节和识别大多数人类观察者无法辨别的症状至关重要。IPT技术还将帮助研究人员评估实验室正在调查的新作物品种的抗病性特征;
- 通过IPT收集的信息可以快速廉价地传播给偏远地区的其他人;
- 正确的诊断将导致更经济地使用杀虫剂,这将降低生产成本,同时保护环境,提高进入欧盟等监管严格但利润丰厚的市场的机会;
- 加强了与人类专家的联系,可以从偏远地区咨询他们,而不需要推广人员亲自访问每个农场。
在作物病虫害检测中使用IPT是一个旨在克服这些限制的活跃研究领域。数码相机和计算硬件的能力和可用性不断提高,加上成本不断降低,这意味着IPT有望在这一领域为人类专业知识提供一种可能的替代方案。
第2.1节表明,早期的作品依赖于经典的图像处理程序和从叶子图像中“手工制作”的特征提取。然后使用这些特征来训练浅层分类器算法,如支持向量机(SVM)、主成分分析(PCA)、最大似然分类(MLC)、最近邻(KNN)、朴素贝叶斯(NB)、决策树(DT)、随机森林(RF)和人工神经网络(ANN)。
最近的工作集中在深度学习卷积神经网络(CNN)架构的开发,以自动执行特征提取和图像分类。这一趋势是由三个主要因素促成的;更大数据集的可用性、多核图形处理单元(GPU)适应深度神经网络的训练以及支持软件库的开发,如英伟达公司的计算统一设备架构(CUDA)。第2.2节讨论了基于深度学习的疾病识别技术。
1.1基于手动特征提取的病虫害识别
- 图像捕捉:合适的相机收集摄影信息(或者来自于网络上的图片或者是来自于专业人员采集的农作物病虫害数据集);
- 图像预处理(提高图像质量):图像尺寸调整、滤波、色彩空间转换和直方图均衡化;
- 图像分割:首先将对象(叶子、果实或者花朵)与背景分割,再将健康组织与患病组织分离;
- 特征提取(利于病虫害的准确分类):提取的特征主要是纹理(能量、对比度、同质性和相关性)、形状、大小和颜色。纹理特征可以使用统计方法提取,如局部二值模式(LBP)、灰度共生矩阵(GLCM)、颜色共生矩阵(CCM)和空间灰度相关矩阵(SGLDM)。纹理特征也可以使用基于模型的方法提取,如自回归和马尔可夫随机场(MRF)模型。
- 识别与分类:为MLA(机器学习算法)提供特征向量,并训练其对与待识别的每种疾病相关的特征进行分类。然后,训练好的算法可用于从野外捕获的新图像中识别特征。分类处理给定的输入特征向量与在训练期间学习的不同类别之一的匹配。设计者可以使用一个或一个以上的学习算法进行训练和分类,并融合算法的结果。
下表简要概述了利用手工特征提取和浅层分类器进行叶部病害自动识别的最新研究工作。它总结了预处理技术、使用的特征和分类器以及在各自研究中开发的算法的性能。
| Article | Crop | Dataset | Pre-processing techniques | Features used | Classifier | Performance |
|---|---|---|---|---|---|---|
| [7] | Wheat | 3500 Field images | Colour constancy normalization,automatic segmentation SLIC and manual cropping of the region of interest (RoI) using the tactile screen of the mobile device and Chan-Vese algorithm,RGB to Lab* and HSI conversion | Colour,Texture | RF,NB | Auc:0.82 Acc:0.78 Sensitivity:0.81 Specificity:0.75 |
| [13] | Potato | 300 Lab images | RGB to Lab* conversion,thresholding to remove background and healthy parts of the leaves | Colour,Texture | Multiclass SVM | Accuracy: 0.937 Precision: 0.95 Recall: 0.95 F1 score: 0.95 |
| [19] | Grapes,Wheat | 185 Field images | Resizing, cropping, median filtering,RGB to Lab* conversion,segmentation using K-means clustering, hole filling using morphological operations | Colour,Shape,Texture | BP(N) | BP(N):Wheat acc: 100% Grapes acc: 100% PCA + BP(N):Wheat acc: 100% Grapes acc: 97.14% |
| [21] | Alfalfa | 899 Lab images | Resizing, RGB to grayscale, Lab* and HSV conversion, segmentation using K-means, Fuzzy C-means and K-median clustering | Colour,Shape,Texture | SVM,NB,LDA,Logistic regression analysis and regression tree.PCA used for feature dimension reduction | SVM:97.64% training acc,94.74% testing acc,PCA/NB:80% acc |
| [25] | Potato | 150 Field images | RGB to grayscale conversion,Histogram equalization,segmentation using K-means clustering | Texture | SVM with CS optimization | SVM with CS:98.5% acc,basic SVM:72% acc |
| [30] | 未指明 | 284 Lab images | Resizing, median filtering, RGB to Lab* conversion,segmentation using K-means clustering | Colour,Shape,Texture | Two serially connected SVM classifiers | 87.8% acc |
| [58] | Grapes | 137 Field images | Cropping, resizing, Gaussian filtering, RGB to Lab* and HSV conversion, segmentation using K-means clustering | Colour,Texture | SVM | 88.89% acc(93.33% - downy), 83.33% powdery mildew) |
| [36] | Grapes | 137 Field images | Cropping, resizing, Gaussian filtering, RGB to Lab* and HSV conversion, segmentation using K-means clustering | Colour,Texture | Fused SVM + ANN classifiers | 100% acc |
| [55] | Betel | 20 scanned images | Resizing, RGB to HSV conversion,Otsu segmentation on H channel | — | — | 100% precision 83.33% recall |
| [61][62] | Banana,Beans,Lemon,Rose | 106 Lab and field images | Image cropping, smoothing filtering,RGB to HSI conversion,segmentation using K-means clustering compared to using GA for separation of healthy and diseased tissue | Colour,Texture | GA + SVM Compared to:GA + MDC、K-means + MDC | 86.54% acc(K-means + MDC), 93.63% acc(GA + MDC), 95.71% acc(GA + SVM) |
| [63] | Tomato | 383 Field images | Segmentation for background removal using Otsu thresholding | Colour,Shape,Texture | DT | 97.3% acc |
| [64] | Tomato | 3535 Lab images(PlantVillage dataset) | Segmentation by colour thresholding, hole filling for mask refinement | Texture (SIFTwithGEV distribution for dimensionality reduction | SVM | 84.7% acc,9000 predictions per second |
| [65] | Tomato | 3535 Lab images(PlantVillage dataset) | Segmentation by colour thresholding, hole filling for mask refinement | Texture (SIFTwith Johnson SB distribution for dimensionality reduction) | SVM | 85.1% acc,5700 predictions per second |
| [79] | Rice | 650 field images | Resizing to 300x450 pixels, RGB to HSV conversion, threshold S component at 90 to obtain background removal mask, k-means clustering using H component to separate infected region | Colour,Texture | DNN-JOA validated against DNN, ANN and DAE | Accuracy: 94.25% F1-score: 88.74% Precision: 81.24% FDR: 19.64% FPR: 4.42% FNR: 8.94% TPR: 83.7% TNR: 94.04% NPV: 94.14% |
| [83] | Sunflower | 未指明 | Median filtering, resizing images,segmentation using thresholding and PSO, RGB to HSI conversion | Colour, Texture | MDC | 98% acc |
| [84] | 未指明 | 276 (6 from planet natural + 270 from crowdAI.org) field images | Segmentation using RGA. Compared to GA and K-means clustering | — | BRBFNN compared to SVM and GA | Segmentation specificity: RGA: 0.8558 GA: 0.8139 k-means: 0.7914 Segmentation sensitivity: RGA: 0.8705 GA: 0.8244 k-means: 0.802 Classification Vpc:BRBFNN: 0.8621 SVM: 0.8337 GA: 0.8113Classification Vpe:BRBFNN: 0.1118 SVM: 0.1665 GA: 0.1933 |
| [85] | 18 species | 48 (training), 46 (testing) field images | Segmentation using GrabCut algorithm, RGB to HSV conversion | LBP histograms | One-class SVM classifiers with nearest support vector strategy for conflict resolution | 95% test set acc |
总结:基于上表中的信息,很明显,疾病识别算法的成功取决于许多变量,这些变量取决于系统设计者的判断。这些包括选择要使用的预处理和分割技术,采用哪个颜色空间,提取哪些特征,最后使用哪个学习算法进行分类。当试图使用手工制作的特征提取和浅分类器进行自动植物病害识别时,没有办法先验地判断预处理、特征提取或分类算法的哪个组合将产生最佳结果,从而导致繁琐的试错方法[7,13,19,21,25,30,36,55,58,61–65,79,83–85]。此外,手工制作的特征提取方法只有在有限和受约束的设置下才能成功,当操作条件略有变化时就会失败[14,15,22,59]。也已经注意到,分割技术给出不可靠的结果,特别是在复杂背景存在的情况下,并且损伤没有良好定义的边缘,而是逐渐与叶子的健康部分合并[14,88]。此外,一些用于分类的最佳特征不能用目前可用的任何已知数学工具手工提取。
1.2基于深度学习网络自动特征提取的病虫害识别
近年来,卷积神经网络(CNN)在图像识别任务中作为特征提取器和分类器表现突出,如Image-Net挑战。这一想法已经扩展到农业应用,以完成诸如疾病识别、害虫识别、杂草检测、水果和花卉计数以及水果分类和分级等任务。自2015年以来,大多数利用IPTs进行叶部疾病识别的研究都利用了深度学习。LeCun等人将深度学习定义为一种表示学习方法,算法通过一系列优化而不是语义特征来找到表示数据的最佳方式。通过这个学习过程,不需要进行特征工程,因为特征是自动提取的。深度学习将推动农业在疾病诊断、害虫检测、质量管理、营销、自动化、机器人和大数据领域的进步。
训练神经网络需要由数千幅图像组成的大数据集。不幸的是,在植物病害识别领域,这样大而多样的数据集还没有被组装起来供研究人员使用。目前,迁移学习是训练鲁棒的CNN分类器进行植物病害识别的最有效方法。迁移学习使预先训练的卷积神经网络能够通过用较小的数据集重新训练它们来适应,这些数据集的分布不同于以前从头开始训练网络所用的较大数据集。事实上,研究表明,使用在ImageNet数据集上预先训练的CNN模型,然后对它们进行再训练以识别叶部疾病,会产生更好的结果。
下表简要概述了利用深度学习在作物病虫害识别方面进行的最新的研究工作。对每个研究中使用的数据集以及各自提出的算法的性能也提供了简要描述。
| Article | CNN architecture | Validation | Deep learning framework | Crop | Dataset | Performance metric |
|---|---|---|---|---|---|---|
| [8] | Faster R-CNN | Different feature extractor CNNs: AlexNet, VGG-16, GoogLeNet, ZFNet, ResNet-50, ResNet-101 and ResNetXt-101 | Caffe | Tomato | 5000 self-acquired field images representing 9 classes of pests and diseases | mAP (0.8306) |
| [9] | CaffeNet (AlexNet) | none | Caffe | Apple, pear, grape,cherry, peach | 4483 Self-acquired field images | Acc(96.3%) |
| [14] | LeNet | none | Deeplearning4j | Banana | PlantVillage (3700 field images) | Acc(0.9861), precision(0.9867),recall (0.986),F1-score (0.9864) |
| [16] | GoogLeNet | none | Matlab | 14 crops | 1575 self-acquired field images | Acc(94%) |
| [18] | AlexNet | MLP | Caffe | Apple | 1450 Self-acquired lab images | Acc(96.6%) |
| [20] | Custom CNN (AlexNet precursor with cascade inception) | SVM, BP ,AlexNet, GoogLeNet, ResNet-20,VGG16 | Caffe | Apple | 1053 self-acquired lab images | Acc(97.62%) |
| [22] | Modified LeNet | SIFT + RBF-SVM, background suppressing Gabor energy filtering + RBF-SVM,uLBP + RBF-SVM | Matlab | Olive | Self-acquired lab images | Accuracy (98.6%), Matthew’s correlation coefficient(0.9798),F1-score (0.9689),Precision (0.9882),Recall (0.9718) |
| [26] | AlexNet, GoogLeNet | SVM, Random Forest | DIGITS | Tomato | PlantVillage | Acc(99.185%), Macro precision (98.529%),Macro recall (98.532%), Macro F score (98.518%) |
| [27] | AlexNet, DenseNet169,Inceptionv3, ResNet34,SqueezeNet, VGG13 | none | pyTorch | 14 crops, 26 diseases | PlantVillage | Acc(0.9976) |
| [28][29] | Custom CNN | none | Caffe | Cucumber | Self-acquired field images(7320 good condition images& 7520 bad condition images) | Acc(82.3%), sensitivity (79.9%), specificity (92.6%) |
| [31] | AlexNet, GoogLeNet | none | Caffe | 14 crops, 26 diseases | PlantVillage | F1-score (0.9934),Precision (0.9935),Recall (0.9935),Acc(0.9935) |
| [33] | Custom ensemble CNN | none | Keras on Theano | Maize | 1796 self acquired field images (1028 infected with NLB and 768 healthy) | Acc(0.967) |
| [35] | Faster R-CNN (modified) | Faster R-CNN (unmodified) | Matlab | Sugar beet | 155 self-acquired images (38healthy, 20 mild, 35 severe,62 mixed mild and severe infection) | Sensitivity (95.48%),Specificity (95.48%),Acc(95.48%) |
| [69] | MobileNets | none | Caffe | Tomato | PlantVillage | Acc(90.3%) |
| [70] | Inceptionv3, ResNet-50,VGG19, Xception | Transfer learning (TF),Feature tuning 25, 50, 75,100%, No TF | Keras | Soybean | 300 high resolution field images divided into 3000 superpixel images | Accuracy (99.04%),Learning error (0.049) |
| [68] | DCGAN, ProGAN and StyleGAN for generation of synthetic training images Custom CNN – PDNet withYolo/AlexNet detector and 32 layer residual classifier network | AlexNet, Vgg19, Inceptionv3,DenseNet201, ResNet152 | Not stated | 12 crop species, 42 diseases | 18,334 PlantVillage lab images + 79,265 self-acquired field images (Plant disease dataset) | Detection mAP (0.9165) Recognition acc(0.9367) |
| [67] | Faster R-CNN with VGG16 feature extractor | none | Caffe | Tomato | 8927 self-acquired images representing 9 anomalies and one class for background | mAP (96%) |
| [37] | GPMNet – (GoogLeNet) | none | Matlab | Grape | 14,180 self-acquired lab sub-images | Acc(94.3%),AuC (0.984) |
| [52] | INAR-SSD | Faster R-CNN, SSD, R-SSD | Caffe | Apple | 2029 lab and field images | mAP (78.8%)Detection speed (23.13 FPS) |
| [53] | AlexNet | none | Not stated | Rice | 227 self-acquired images(both pest and disease) | Acc(91.23%) |
| [54] | Modified Cifar10 | Basic Cifar10 and GoogLeNet CNNs | Caffe | Maize (8 diseases + healthy) | 500 images | Acc(98.9%) |
| [56] | Custom CNN | VGG16, VGG19, Inceptionv3,ResNet-50 | Keras on Theano | Apple | PlantVillage (healthy & black rot images) | Acc(90.4%) |
| [57] | Custom CNN | none | Not stated | Soybean | PlantVillage | Acc(99.21%),Recall(0.99),Precision (0.99),F1-Score (0.99) |
| [71] | AlexNet, SqueezeNet | none | Caffe on Nvidia Jetson | Tomato | Plantvillage | Acc(0.943) |
| [72] | Custom CNN | BP , SVM, PSO | Matlab | Rice | 500 self-acquired field images representing 10 rice diseases | Acc(95%),Missing report rate (0),False report rate (0) |
| [73] | Modified Inceptionv3 | Inceptionv3 | Keras on TensorFlow | Tomato | PlantVillage | Acc(0.971) |
| [74] | CaffeNet up to fc layers + hand crafted features + SVM classifier | Hand crafted + SVM, CNN,fc6/7 + SVM | Caffe | Trees – 57 species | PlantNet, Flavia and LeafSnap + self-acquired images | Acc(99.68%) |
| [88] | Custom 3 channel CNN | Image processing technology(IPT), Global-Local Singular Value Decomposition(GLSVD), SVM, Sparse Representation Based Classification (SRC) | Matlab | Tomato, cucumber | PlantVillage (tomato), 500self-acquired images(cucumber) | Accuracy (94.15% for cucumber and 91.16% for tomato) |
| [89] | AlexNet, ResNet-50, ResNet-101 | SVM, BP | Caffe | Pests | 550 self-acquired field images (10 species each having 55 images) | Acc(98.67%) |
| [106] | VGG16, AlexNet | none | Matlab | Tomato | PlantVillage (6 tomato diseases+ healthy) | Acc(96.19%) |
| [120] | Modified ResNet-50 | Shallow classifiers in [7], Re-sNet-50 with fully convolutional dense layer | TensorFlow | Wheat | 8178 self-acquired images(3338 Rust, 2744 Septoria,1568 Tan Spot, 1116 healthy) | AuC (0.97),Sensitivity (0.91), Specificity(0.95),Balanced acc(0.93) |
| [107] | Modified CIFAR10-quick CNN | LeNet-5, AlexNet, VGG16, BP,Bayesian, SVM, KNN classifiers | Matlab | Tea | 144 self-acquired images (36 images each of healthy, tea leaf blight, tea bud blight, tea red scab) | Accuracy (92.5%),Loss (0.002) |
| [123] | AlexNet, VGG16 | Fine tuning, training from scratch, CNN feature extractors + SVM/LDA, CNN feature extractors + RNN | Not stated | 14 crop species, 87 tree species obtained from 3 public datasets | PlantVillage (54,306 images),Flavia (1907 images),Swedish leaf dataset (1125),UCI leaf dataset (443 images) | Acc Flavia –VGG16 + LDA(99.10%),Swedish leaf – VGG16(99.11%),UCI leaf – AlexNet + LDA(96.2%),PlantVillage – VGG16 (99.8%) |
| [124] | Custom CNN (MCNN) | PSO, SVM, RBFNN | TensorFlow | Mango | 2200 images (512 healthy mango, 558 infected mango,520 other leaves healthy, 610 other leaves infected) | Accuracy (97.13%),Missing report rate (2.87),False report rate (0) |
通过上表我们可以知道PlantVillage数据集是该领域应用最广泛的数据集。常用的系统性能指标是准确度、精密度、灵敏度/召回、特异性和F1score。在所有将深度学习算法与手工算法进行比较的研究中,深度学习提供了更高的识别精度。Caffe和Matlab是应用最广泛的深度学习框架。Keras、pyTorch、TensorFlow和Theano框架在该领域也受到了一些关注。
还可以得到以下的重要结论(论文中有讲到,在这里把精华提取出来了):
- 在训练模型时,比起增加训练epochs的数目,增加数据对识别性能的贡献更大[28];
- 数据增强(比如:一种训练图像是条件比较好的,而另外一种是条件好的和差的混合的,这样一来后面训练得到的模型的性能更好)可以提高识别的性能[29];
- 与MLP(多层感知机)和人类专家相比,CNN的识别率更高[18];
- 使用彩色图像训练的模型比使用灰度图像训练的模型具有更好的性能[14];
- 与从零开始的训练相比,迁移学习产生了更好的效果[53];
- 即使是在嵌入式应用中,先在传统GPU上训练模型,然后将训练后的模型部署到嵌入式平台进行推理任务,也可以获得较高的精度性能[71];
- 增加网络层数可以提高识别精度,但是在简单的串联结构中,仅仅增加层数并不能保证更高的识别精度,这是因为随着层数的增加,梯度消失了。残差体系结构克服了层数增加而梯度消失的问题[56,89];
- 在设计用于植物异常识别的深度学习模型时,需要尝试不同的超参数设置和网络拓扑结构,以找到性能最佳的架构。如果仔细考虑网络的设计和适当的训练超参数的选择,小型CNN架构可以达到与更复杂模型相当的性能水平。不幸的是,除了反复试验,没有已知的方法可以做到这一点[54,57,72,88,106,107];
- 结合几个CNN分类器可以产生更好的识别精度结果,并且可以从非常高分辨率的图像中检测疾病症状[33];
- 深度学习检测器网络有能力识别同一植物上的多种感染,这种方式对植物上病变的大小和位置以及感染阶段的类内变异具有鲁棒性,使用检测器的一个主要缺点是,通过标记感兴趣区域周围的边界框来进行数据集注释是一个非常劳动密集的过程[8,67,52];
- 在移动电话上实现紧凑的深度CNN用于植物病害识别是可能的[69];优化算法的选择对模型的整体精度影响不大。此外,在不同学习速率下的实验表明,通过降低学习速率,以牺牲较长的训练时间为代价,可以获得更高的准确率[114];
- 在无人机和机器人上部署叶病识别系统是可能的[70];
- 人工生成训练图像有很大的潜力来解决大数据集不可用的问题。然而,人工野外条件图像的生成一直没有得到解决[68];
- 由于植物病虫害识别的数据集目前的多样性比较差,因此只需要一个比较简单紧凑的CNN模型就足够了,在训练的过程中可以通过这(隐藏层输出可视化、特征可视化、语义字典和注意力地图)四种技术来查看CNN中的权重是否有冗余,如果有就可以将冗余的部分给去除掉,以此来获得一个与原模型一样性能的简单紧凑的更瘦的模型[73];
1.3基于手动特征提取和深度学习结合的病虫害识别
许多作者[22,74]在认识到深度cnn比手工特征提取方法的优越性的同时,仍然试图通过结合这两种特征提取方法来提高分类器的性能。他们表明,在全连接层从CNN获取特征向量结果,并将其与手工制作的特征向量相结合,可以获得更准确的分类器性能。
Cruz等人在[22]中提出了一种利用深度学习与数据融合的方法检测Olive Quick Decline Syndrome (OQDS)的技术。作者对CNN进行了修改,将额外手工制作的顺序统计量、纹理、形状和几何关系特征向量与CNN的全连接层进行连接,如下图所示。有人提出,这将使网络更好地泛化,加速模型的收敛,即使在数据集很小的时候。将深度学习模型的性能与手工技术结合径向基函数支持向量机(RBF-SVM)分类器进行比较。与手工制作的方法相比,CNN在所有场景中都表现出了优越的识别性能,CNN在这些场景中接受了200个或更多的训练。

[74]使用Treelogy叶片数据集,实验了不同的特征提取器分类器组合,如下表所示。从输入图像中提取特征可以是手工提取,也可以是使用改进的AlexNet CNN。通过手工提取15个特征,得到56x1个特征向量。从CNN得到的每个特征向量的大小为4096x1。作者表明,将手工制作的特征向量与第一全连接层(fc6)的特征向量融合,可以获得最高的识别精度。然而,将第二层全连接层(fc7)的特征向量与手工制作的特征向量融合,与单独使用fc7相比,精度较低。

下图表明,番茄在最多的研究中占有重要地位。苹果、葡萄、玉米和大豆也很受这一领域研究人员的欢迎。只有三项研究考虑了对害虫的识别。Fuentes等[8,67]考虑了发生在番茄作物上的害虫,而Cheng等[89]考虑了发生在各种作物上的10种害虫。诸如木薯、咖啡、茶、洋葱、百香果、香蕉、梨、椰子和橄榄等作物分别被考虑在一项研究中。C公司的研究成果也在此基础上展开,他们的研究主要集中在树木/农作物的叶片识别上。然而,到目前为止,利用深度学习技术进行树木病害识别的研究还没有发表。

2.十种不同CNN体系结构在农作物病虫害识别上的性能比较
研究比较了10种深度学习模型的性能:AlexNet, GoogLeNet, VGG16, ResNet-101, DenseNet201 [125], Inceptionv3, InceptionResNetv2 [126], ShuffleNet [127], SqueezeNet和mobilenet利用PlantVillage数据集进行叶病识别任务[78]。[27]和[128]报道了使用该数据集的类似比较研究。然而,与之前的研究相比,本研究考虑了更多的CNN模型和性能指标。
-
材料和方法
本比较研究使用的数据集包含54305幅叶片图像,代表14种作物和26种疾病。数据集中的所有图像都是在统一背景下的可控条件下捕获的,大小调整为256x256像素,并组织为38类。将数据集中的每个类分为三个子集;训练集、验证集和测试集的比例分别为70%、15%和15%。因此,训练集包含38009张图像,而验证集和测试集各有8147张图像。为了避免偏差,这些集合被用来训练、验证和测试所有10个CNN模型。 -
实验装置
实验在一台运行在Windows 10上的AMD Ryzen 7 2700 CPU,64GB RAM和Nvidia RTX 2060 GPU的计算机上进行,使用了Matlab2019b中的深度学习工具箱。采用了100%的微调策略来重新训练已经在ImageNet数据集上预先训练过的模型。这意味着预先训练的网络中的任何一层都不会被冻结,因此反向传播算法将能够修改网络中的所有权值和偏差。所有模型使用相同的超参数进行30个epoch的训练,如下表所示。没有进行数据增强。

-
结果与讨论
所有10个CNN模型的验证精度和交叉熵损失如下图所示。可以看出,所有的模型在第15次训练的时候都进行了收敛,达到了98.5%以上的准确率。DenseNet201、VGG16和ResNet-101等较深的模型比AlexNet和SqueezeNet等较浅的网络获得了最高的精度。与较浅的网络相比,较深的网络通常也会在更少的epochs收敛。

各网络的测试集性能如表6所示。DenseNet201在每个指标上都优于所有其他架构。尽管可学习参数比VGG16少67.7%,但ResNet-101的性能与VGG16相当(见表7)。此外,VGG16的训练成本非常高,训练时间超过129 h,而ResNet-101的训练时间为24.48 h。DenseNet201体系结构中连接的密集特性可能导致了训练时间比Inception-V3长的原因,尽管Inception-V3的参数比DenseNet201有更多的参数。
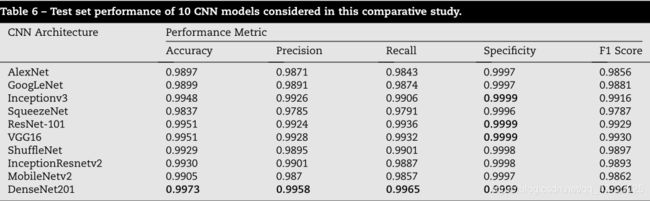

在较大的CNN模型中,DenseNet201、Inceptionv3和ResNet-101似乎最适合实现基于深度学习的叶部疾病识别系统。DenseNet201具有最佳性能和最少存储需求的优势,但缺点是在三种体系结构中训练时间最长。另一方面,Inceptionv3是三个模型中训练时间最少的,不需要像ResNet-101那样多的内存空间。虽然ResNet-101型号的存储要求是三种型号中最高的,但其精度仅次于DenseNet201。此外,ResNet-101可以在训练DenseNet201所需时间的四分之一内进行训练。[73]和[120]中的研究表明,与原始体系结构相比,改进的Inceptionv3和ResNet体系结构可以分别获得相似或更好的性能。因此,在选择这三种网络时应该仔细考虑,因为每种体系结构都有一些优点,但也有一些局限性。
在计算资源受限的移动和嵌入式应用中,小型CNN架构是理想的。ShuffleNet、SqueezeNet和MobileNets可以考虑部署在这类应用中,因为它们的存储要求低,训练时间短,同时仍然可以实现高精度。特别是,与SqueezeNet和MobileNets相比,ShuffleNet具有更好的性能,同时将存储需求限制在6.3MB。在计算能力严重受限的应用程序中,SqueezeNet是一个有吸引力的选择,因为它的最小存储要求为4.6MB,快速训练时间为2.19h。MobileNets似乎没有提供任何优于ShuffleNet和SqueezeNet的优势。在三种紧凑的体系结构中,MobileNets的存储要求最高,培训时间最长,但与ShuffleNet相比,性能较低。
3.现有文献中的问题汇总
- 缺乏足够大的数据集
缺乏大的、标注良好的、高度可变性的图像数据集是训练植物病害识别深度学习系统的主要障碍。目前,PlantVillage数据集和植物病害症状图像数据库(PDDB)数据集仍然是仅有的大型免费可用的数据集。针对这一问题,人们提出了Crowd sourcing和免费共享已经可用的数据集。 - 数据增强
在缺乏大数据集的情况下,数据增强已经被证明可以显著提高模型的性能。除了常规的使用图像平移、裁剪、翻转、镜像等方式进行数据增强外,还可以使用GANs人工生成更多的图像用于训练。常常是在统一的背景下生成树叶图像,需要更多的工作来人工生成具有复杂背景和变化的姿态和照明的图像。目前还没有开发出适用于病变区域的作物叶片图像自动生成的技术。 - 图像分割
对复杂背景下的叶片进行分割可以提高识别精度。有人建议,在移动应用程序中,用户可以在疾病识别之前,手动裁剪感兴趣区域周围的叶片图像。然而,这限制了叶病识别技术的自动化及其在无人机和机器人中的应用。此外,一个没有经验的用户可能仍然不能从这样一个系统中受益,他/她必须手动突出感兴趣的区域。因此,需要开发可靠的自动分割技术来去除背景和叶子隔离。 - 识别具有相似特征的病虫害
如果不同异常表现出的症状在视觉上非常相似,分类器可能无法区分它们。利用多种信息源,如地理位置、天气趋势、作物发育阶段和历史病虫害发病率数据,增加可见线索,将有助于获得更准确的预测。目前,没有文献报道将这些补充信息纳入疾病诊断过程。 - 识别植物其他部分的病虫害
迄今为止,研究人员一直专注于检测和识别叶子上部的疾病。利用茎、果和花的图像进行病害识别,目前还没有得到足够的重视。有研究人员利用更快的R-CNN网络检测番茄植株不同位置多种疾病的技术。这些技术虽然非常精确,但需要对数据集进行艰苦的标记和注释。在我们看来,这将限制这种方法的可伸缩性。于是有研究人员建议开发一种检测器,可以使用简单的图像进行训练,然后应用于检测和识别复杂图像中的害虫和疾病。 - 用于疾病检测的紧凑深度学习模型的开发
目前提出的基于深度学习的解决方案大多依赖于著名的CNN架构,如AlexNet、googlenet、VGG、ResNet和Inceptionv3。这主要是由于缺乏足够大的数据集来训练定制的CNN架构从无到有进行植物病害识别。众所周知的体系结构通过使用迁移学习技术提供了一种解决训练数据量不足问题的方法。然而,这些著名的CNN模型对于植物病害识别任务的计算要求很高,而且过于复杂。因此,我们要去实现一种不过度拟合,精确且紧凑的模型。紧凑的CNN模型是非常可取的,特别是在嵌入式、机器人和移动应用中,这些应用需要实时性能和低计算成本。【意思就是用尽量少的神经元去实现最优的识别效果,而不是只考虑最优的识别效果。】
4.本文献的总结
- 综述了近年来利用IPT技术进行植物病虫害识别的研究进展,通过不同设计的性能,我们可以知道,深度学习技术已经逐步取代了使用手工制作的特征训练的浅分类器;
- 在有足够数据集可供训练的情况下,深度学习技术能够获得更高的病虫害识别准确率,于是本文讨论了收集具有高可变性、数据增强、迁移学习和CNN激活地图可视化的大数据集对提高分类精度的重要性;
- 对10种最先进的CNN模型在叶病识别任务上的性能进行比较评估,并比较已经被量化的7项性能指标;DenseNet201、ResNet-101和Inception-V3架构最适合正常计算环境中使用的模型,而ShuffleNet和SqueezeNet是最适合移动和嵌入式应用的架构;
- 强调了现有文献的不足之处,以指导该领域未来的研究工作;
- 为了能够在移动和嵌入式平台上采用这些技术,有必要开发仍然能够实现高精度的紧凑CNN模型;
- 今后的研究工作还应侧重于开发可靠的消除背景技术和纳入地理位置、发病率历史和天气趋势等形式的数据,以提高疾病识别系统的准确性和可靠性;
- 识别疾病症状发生在植物的其他部分,如水果、花和茎还没有被研究人员广泛的解决;
