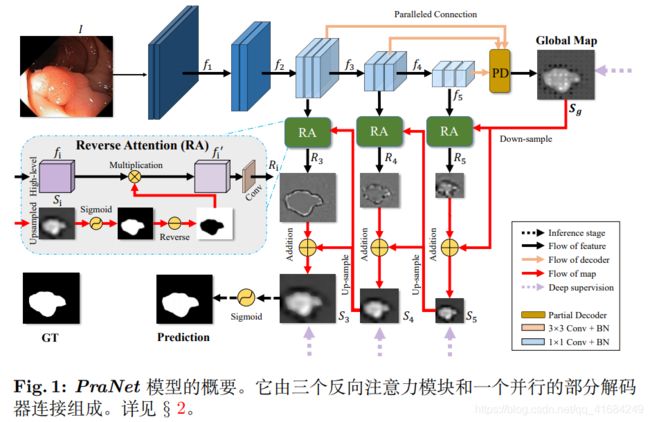
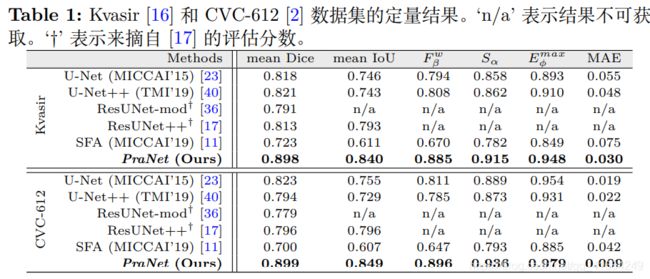
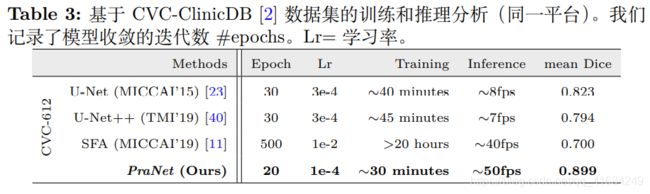
- 21天刷题计划之10.1—统计大写字母个数(Java语言描述)
justlikeu777
21天刷题计划java基础算法基础
题目描述:找出给定字符串中大写字符(即’A’-‘Z’)的个数接口说明原型:intCalcCapital(Stringstr);返回值:int输入描述:输入一个String数据输出描述:输出string中大写字母的个数示例1输入add123#$%#%#O输出1分析:获取输出的字符串,将字符串转换成字符数组,遍历字符数组并判断是否为大写字母即可。importjava.util.Scanner;publ
- 也谈一下 30+ 程序员的出路
写编程的木木
langchain产品经理python开发语言大模型
前言前两天和一个前端同学聊天,他说不准备再做前端了,准备去考公。不过难度也很大。从20152016年那会儿开始互联网行业爆发,到现在有7、8年了,当年20多岁的小伙子们,现在也都30+了大量的人面临这个问题:大龄程序员就业竞争力差,未来该如何安身立命?先说我个人的看法:除非你有其他更好的资源,否则没有更好的出路认真搞技术,保持技术能力,你大概率不会失业(至少外包还在招人,外包也不少挣…)考公之我见
- 【优秀文章】7月优秀文章推荐
优秀文章智能自主运动体与人工智能技术——环境感知、SLAM定位、路径规划、运动控制、多智能体协同作者:fpga和matlabC++之红黑树认识与实现作者:zzh_zao【手把手带你刷好题】–C语言基础编程题(十)作者:草莓熊Lotso飞算JavaAI:从“码农”到“代码指挥官”的终极进化论作者:可涵不会debug前端网页开发学习(HTML+CSS+JS)有这一篇就够!作者:一颗小谷粒
- 用 AI “一句话生成代码”,用创意兑换灵码潮品:技术人的夏日狂欢季来了
人工智能
在AI技术迅猛发展的2025年,我们正式推出“通义灵码编程智能体挑战季”,以“码力觉醒”为主题,打造一场融合技术探索与潮流文化的开发者盛宴。活动以体验MCP服务、Qwen3大模型及记忆功能的智能编程助手为核心,通过“小游戏开发”和“MCP场景实践”两大趣味赛道,降低AI技术门槛,让开发者轻松体验“一句话生成代码”的魔力。活动亮点抢先看:零门槛参与:新老用户均可参与,完成任务即领限量定制棒球帽!趣味
- 蛋白质结构预测/功能注释/交互识别/按需设计,中国海洋大学张树刚团队直击蛋白质智能计算核心任务
hyperai
蛋白质作为生命活动的主要承担者,在人体生理功能中扮演关键角色。然而传统研究面临结构解析成本高昂、功能注释严重滞后、新型蛋白质设计效率低下等挑战。近年来,生命科学对蛋白质复杂特性解析的需求日益迫切,大数据、深度学习、多模态计算等技术的突破性发展,为构建蛋白质智能计算体系提供了全新的发展契机。蛋白质智能计算体系的构建,使得蛋白质在大规模功能注释、交互预测及三维结构建模等领域取得显著成果,为药物发现与生
- 【心灵鸡汤】深度学习技能形成树:从零基础到AI专家的成长路径全解析
智算菩萨
人工智能深度学习
引言:技能树的生长哲学在这个人工智能浪潮汹涌的时代,深度学习犹如一棵参天大树,其根系深深扎入数学与计算科学的沃土,主干挺拔地承载着机器学习的核心理念,而枝叶则繁茂地延伸至计算机视觉、自然语言处理、强化学习等各个应用领域。对于初入此领域的新手而言,理解这棵技能树的生长规律,掌握其形成过程中的关键节点和发展阶段,将直接决定其在人工智能道路上能够走多远、攀多高。技能树的概念源于游戏设计,但在学习深度学习
- 【秋招算法】2025 届搜广推方向求职历程(SSP、头部计划)
秋冬无暖阳°
搜广推等—算法面经面试职场和发展算法
【秋招算法】2025届搜广推方向求职历程(SSP、头部计划)文章目录【秋招算法】2025届搜广推方向求职历程(SSP、头部计划)1.背景2.日常实习3.暑期实习3.1暑期BG3.2暑期记录4.秋招4.1秋招BG4.2转正4.3头部4.4提前批4.5正式批5.面试记录5.1Coding5.2其他高频编程题5.3常见八股、面经6.关于搜广推1.背景关于日常实习、暑期实习、提前批,秋招、春招、补招何为大
- 2025年渗透测试面试题总结-2025年HW(护网面试) 31(题目+回答)
独行soc
2025年护网面试职场和发展安全linux护网渗透测试
安全领域各种资源,学习文档,以及工具分享、前沿信息分享、POC、EXP分享。不定期分享各种好玩的项目及好用的工具,欢迎关注。目录2025年HW(护网面试)311.自我介绍2.渗透测试流程(五阶段模型)3.技术栈与开发经历4.自动化挖洞实践5.信息搜集方法论6.深度漏洞挖掘案例8.SQL注入实战技巧9.AWVS扫描与防御10.CSRFvsSSRF核心差异11.SSRF正则绕过技术12.虚拟主机识别原
- 【计算机毕业设计】基于Springboot的办公用品管理系统+LW
枫叶学长(专业接毕设)
Java毕业设计实战案例课程设计springboot后端
博主介绍:✌全网粉丝3W+,csdn特邀作者、CSDN新星计划导师、Java领域优质创作者,掘金/华为云/阿里云/InfoQ等平台优质作者、专注于Java技术领域和学生毕业项目实战,高校老师/讲师/同行前辈交流✌技术范围:SpringBoot、Vue、SSM、HLMT、Jsp、PHP、Nodejs、Python、爬虫、数据可视化、小程序、安卓app、大数据、物联网、机器学习等设计与开发。主要内容:
- ShardingSphere-JDBC 详解
csdn_tom_168
ApacheShardingSphere数据库ShardingSphereJDBC学习
ShardingSphere-JDBC(原Sharding-JDBC)是ApacheShardingSphere的核心模块之一,定位为轻量级Java框架,在Java的JDBC层提供分库分表、读写分离、数据加密、影子库等分布式数据库增强能力。它直接操作JDBC接口,对应用透明,集成成本极低。以下是ShardingSphere-JDBC的详解:一、核心功能数据分片:分库分表:将逻辑上的大表(库)拆分成
- MongoDB + Voyage AI 详解:重塑数据库与AI的协同范式
csdn_tom_168
NoSQL数据库mongodb人工智能AI
MongoDB+VoyageAI详解:重塑数据库与AI的协同范式2025年2月,MongoDB官方宣布收购VoyageAI,这一举措标志着数据库与人工智能技术的深度融合迈入新阶段。通过整合VoyageAI的先进AI检索与嵌入模型能力,MongoDB旨在重新定义AI时代的数据库架构,为企业构建智能应用提供端到端的数据基础设施。一、收购背景与技术战略1.行业趋势驱动AI数据挑战:随着生成式AI与大语言
- HarmonyOS5.0仓颉引擎与盘古大模型:个性化作业批改系统架构设计与实现
H老师带你学鸿蒙
系统架构HarmonyOS5.0鸿蒙华为仓颉教育
人工智能与边缘计算的融合正在重塑教育评价体系。本文将展示如何基于HarmonyOS5.0仓颉并发引擎和盘古大模型,构建新一代智能作业批改系统。系统架构全景graphTDA[学生端设备]-->|提交作业|B[仓颉边缘处理]B-->C[盘古大模型分析]C-->D[个性化反馈生成]D-->E[学生终端]D-->F[教师仪表盘]subgraphHarmonyOS分布式系统B-->|设备协同|G[教室平板集
- 阿里云瑶池数据库 Data Agent for Meta 正式发布,让 AI 更懂你的业务!
数据库观点资讯人工智能
背景随着生成式人工智能(GenerativeAI)从概念验证迈向规模化商业落地,AIAgent已成为企业核心业务流程的重要组成部分。然而,当模型调用日益便捷时,核心痛点已不再是模型本身,而是集中在一个关键要素上:数据。AIAgent的落地瓶颈已从技术能力转向高质量、高相关性、安全合规的数据供给。企业面临的核心挑战在于:数据孤岛导致知识库分散,通用大模型难以理解专业业务传统数据管理依赖人工开发维护,
- 【TVM 教程】如何处理 TVM 报错
ApacheTVM是一个深度的深度学习编译框架,适用于CPU、GPU和各种机器学习加速芯片。更多TVM中文文档可访问→https://tvm.hyper.ai/运行TVM时,可能会遇到如下报错:---------------------------------------------------------------AnerroroccurredduringtheexecutionofTVM.F
- 【PaddleOCR】OCR文本检测与文本识别数据集整理,持续更新......
博主简介:曾任某智慧城市类企业算法总监,目前在美国市场的物流公司从事高级算法工程师一职,深耕人工智能领域,精通python数据挖掘、可视化、机器学习等,发表过AI相关的专利并多次在AI类比赛中获奖。CSDN人工智能领域的优质创作者,提供AI相关的技术咨询、项目开发和个性化解决方案等服务,如有需要请站内私信或者联系任意文章底部的的VX名片(ID:xf982831907)博主粉丝群介绍:①群内初中生、
- 多模态大模型的技术应用与未来展望:重构AI交互范式的新引擎
zhaoyi_he
重构人工智能
一、引言:为什么多模态是AI发展的下一场革命?过去十年,深度学习推动了计算机视觉和自然语言处理的飞跃,但两者的发展路径长期割裂。随着生成式AI和大模型时代的到来,**多模态大模型(MultimodalFoundationModels)**以统一的建模方式处理图像、文本、音频、视频等多源数据,重塑了“感知-认知-决策”链条,为AGI迈出关键一步。OpenAI的GPT-4o、Google的Gemini
- 从0到1打造创始人IP:创客匠人如何用内容构建商业护城河
创客匠人老蒋
创始人IP创客匠人IP变现大数据知识付费
创始人IP为何成为企业破局的关键引擎?在知识付费赛道竞争白热化的当下,创客匠人创始人老蒋以“IP新商业架构师”的身份,将个人IP与企业品牌深度绑定,走出了一条差异化路径。当传统企业还在纠结流量成本时,老蒋通过输出“成事心法”“商业认知”等干货内容,在公众号、短视频等平台积累精准用户,其“正确的事做长期”理念,正是创客匠人9年深耕行业的缩影。这种将创始人个人影响力转化为企业信任背书的模式,让创客匠人
- 行业锦标赛激励数据集(2008-2023)
数据皮皮侠AI
人工智能大数据物联网矩阵动态规划
1771行业锦标赛激励数据集(2008-2023)数据简介坚持创新驱动发展,要强化企业创新主体地位,发挥企业家在技术创新中的重要作用。作为企业组织内部最具有影响力的角色,高级管理人员拥有企业经营管理的自由裁量权,对企业战略决策及由此产生的经营绩效具有举足轻重的影响。合理的薪酬契约安排是促进员工努力工作并提高企业绩效的重要手段。效率视角下的锦标赛理论主要关注企业内部薪酬差距的激励效应,但随着信息技术
- 移动conda虚拟环境的安装目录
方法1:重新创建环境(推荐)(1)导出环境配置(生成environment.yml):condaactivateold_env#激活原环境condaenvexport>environment.yml#导出配置(llmtuner):~$condaenvexport>environment.yml(llmtuner):~$tail-fenvironment.yml-websockets==15.0.1
- 使用 C++ 实现 MFCC 特征提取与说话人识别系统
whoarethenext
c++开发语言mfcc语音识别
使用C++实现MFCC特征提取与说话人识别系统在音频处理和人工智能领域,C++凭借其卓越的性能和对硬件的底层控制能力,在实时音频分析、嵌入式设备和高性能计算场景中占据着不可或缺的地位。本文将引导你了解如何使用C++库计算核心的音频特征——梅尔频率倒谱系数(MFCCs),并进一步利用这些特征构建一个说话人识别(声纹识别)系统。Part1:在C/C++中计算MFCCs直接从零开始实现MFCC的所有计算
- OneCode技术架构深度解析:自主UI体系、注解驱动与全栈开发的协同优势
低代码老李
OneCode产品介绍OneCode实战软件行业架构ui
引言:低代码平台的技术基石在AIGC与数字化转型的双重驱动下,企业级低代码平台已从简单的界面搭建工具演进为全栈业务开发环境。OneCode作为国内领先的低代码开发平台,其核心竞争力源于三大技术支柱:自主可控的UI体系、注解驱动的开发模式和端到端的全栈支持能力。这三大支柱形成有机整体,使OneCode在开发效率、系统集成和业务适应性方面建立起显著优势。本文将深入剖析这些技术特性的实现原理与应用价值,
- 样本量计算:配对样本定量资料——平均值法
今天介绍的是配对样本定量资料采用平均值法的样本量计算。先来看一下案例。一、案例为明确某种新的训练计划是否能显著提高运动员的100米短跑成绩,欲招募一批志愿者,分别记录运动员在进行新训练计划前后的100米短跑成绩(秒)。据早期研究,两配对样本差值的标准差为5秒,若接受新的训练计划前后的100米短跑成绩平均值差为3秒,问至少需要招募多少志愿者?运动员的100米短跑成绩属于连续性数据。经正态性检验,成绩
- ImportError: /nvidia/cusparse/lib/libcusparse.so.12: undefined symbol: __nvJitLinkComplete_12_4
爱编程的喵喵
Python基础课程pythonImportErrortorchnvJitLink解决方案
大家好,我是爱编程的喵喵。双985硕士毕业,现担任全栈工程师一职,热衷于将数据思维应用到工作与生活中。从事机器学习以及相关的前后端开发工作。曾在阿里云、科大讯飞、CCF等比赛获得多次Top名次。现为CSDN博客专家、人工智能领域优质创作者。喜欢通过博客创作的方式对所学的知识进行总结与归纳,不仅形成深入且独到的理解,而且能够帮助新手快速入门。 本文主要介绍了ImportError:/home/
- 原神4.8版本双号升级计划角色数据列表
妖为邻
csscss3前端原神4.8版本数据列表2个号升级计划角色数据
原神4.8版本升级计划数据表*{margin:0;padding:0;box-sizing:border-box;body{background:#1c3b5c;}a{color:#e6a23c;}}header{width:99vw;height:40px;display:flex;justify-content:space-between;align-items:center;backgrou
- SharePlex for Oracle应用系统高可用和容灾方案
dsg_gulibin
【正Dataguardrmanoracle数据库服务器constraints数据备份产品
第1章前言在企业信息化进程不断加快的今天,保持业务的连续性是企业用户进行数据存储时必须考虑的重要方面。灾难的出现可能导致生产停顿、客户满意度降低,减少企业的竞争力。如何安全、可靠、完整地保存数据,实现系统的灾难恢复是市场竞争的需要,更是进一步提高服务水平和改善服务质量、提升业务支撑能力的重要技术手段。“911”事件使大家更加谨慎地审视自己的应用系统。据有关数据表明,接近50%的公司需要关键业务24
- 机器人动力学模型及其线性化阻抗控制模型
机器人动力学模型机器人动力学模型描述了机器人的运动与所受力和力矩之间的关系。这个模型考虑了机器人的质量、惯性、关节摩擦、重力等多种因素,用于预测和解释机器人在给定输入下的动态行为。动力学模型是设计机器人控制器的基础,它可以帮助我们理解机器人如何响应控制指令,并优化机器人的运动性能。具体来说,机器人动力学模型通常由一组微分方程组成,这些方程描述了机器人各关节的加速度、速度和位置与施加在关节上的力和力
- 【机器学习&深度学习】多分类评估策略
一叶千舟
深度学习【理论】深度学习【应用必备常识】大数据人工智能
目录前言一、多分类3大策略✅宏平均(MacroAverage)✅加权平均(WeightedAverage)✅微平均(MicroAverage)二、类比理解2.1宏平均(MacroAverage)2.1.1计算方式2.1.2适合场景2.1.3宏平均不适用的场景2.1.4宏平均一般用在哪些指标上?2.1.5怎么看macroavg指标?2.1.6宏平均值低说明了什么?2.1.7从宏平均指标中定位模型短板
- 网络安全相关专业总结(非常详细)零基础入门到精通,收藏这一篇就够了
网络安全工程师教学
兼职副业黑客技术网络安全web安全安全人工智能网络运维
一、网络工程专业专业内涵网络工程是指按计划进行的以工程化的思想、方式、方法,设计、研发和解决网络系统问题的工程,一般指计算机网络系统的开发与构建。该专业培养具备计算机科学与技术学科理论基础,掌握网络技术领域专业知识和基本技能,在计算机、网络及人工智能领域的工程实践和应用方面受到良好训练,具有深厚通信背景、可持续发展、能力较强的高水平工程技术人才。学生可在计算机软硬件系统、互联网、移动互联网及新一代
- 动态链接库:技术赋能,打通“最后一公里”的商业模式
超级晒盐人
经验分享学习方法人工智能教育电商程序员创富
序言-飞跃大山的故事阿登是一位热爱骑行的冒险者,他计划骑行穿越一片壮丽的山脉,去探索那片未知的风景。然而,当他查看路线图时,他发现其中有一座几乎不可能翻越的高峰。这座山峰不仅陡峭无比,还充满了危险的地形,即使是经验最丰富的骑行者也望而却步。阿登知道,仅凭自己的力量,很难完成这次骑行计划。于是,他开始寻找解决方案。在一次偶然的机会中,他听说了一位名叫阿飞的飞行员,他提供一种独特的服务——用直升机帮助
- OpenCV 图像操作:颜色识别、替换与水印添加
目录引言代码实现1.导入必要的库2.图像加法3.图像直接相加4.颜色加权加法5.HSV颜色空间转换概念作用6.查找颜色范围对应的像素点7.与运算-生成掩膜8.添加水印9.主函数总结引言在计算机视觉领域,OpenCV是一个强大的库,提供了丰富的图像操作功能。本文将详细介绍如何使用OpenCV进行图像加法、颜色加权加法、HSV颜色空间转换、颜色范围查找、与运算生成掩膜以及添加水印等操作,并给出相应的P
- web报表工具FineReport常见的数据集报错错误代码和解释
老A不折腾
web报表finereport代码可视化工具
在使用finereport制作报表,若预览发生错误,很多朋友便手忙脚乱不知所措了,其实没什么,只要看懂报错代码和含义,可以很快的排除错误,这里我就分享一下finereport的数据集报错错误代码和解释,如果有说的不准确的地方,也请各位小伙伴纠正一下。
NS-war-remote=错误代码\:1117 压缩部署不支持远程设计
NS_LayerReport_MultiDs=错误代码
- Java的WeakReference与WeakHashMap
bylijinnan
java弱引用
首先看看 WeakReference
wiki 上 Weak reference 的一个例子:
public class ReferenceTest {
public static void main(String[] args) throws InterruptedException {
WeakReference r = new Wea
- Linux——(hostname)主机名与ip的映射
eksliang
linuxhostname
一、 什么是主机名
无论在局域网还是INTERNET上,每台主机都有一个IP地址,是为了区分此台主机和彼台主机,也就是说IP地址就是主机的门牌号。但IP地址不方便记忆,所以又有了域名。域名只是在公网(INtERNET)中存在,每个域名都对应一个IP地址,但一个IP地址可有对应多个域名。域名类型 linuxsir.org 这样的;
主机名是用于什么的呢?
答:在一个局域网中,每台机器都有一个主
- oracle 常用技巧
18289753290
oracle常用技巧 ①复制表结构和数据 create table temp_clientloginUser as select distinct userid from tbusrtloginlog ②仅复制数据 如果表结构一样 insert into mytable select * &nb
- 使用c3p0数据库连接池时出现com.mchange.v2.resourcepool.TimeoutException
酷的飞上天空
exception
有一个线上环境使用的是c3p0数据库,为外部提供接口服务。最近访问压力增大后台tomcat的日志里面频繁出现
com.mchange.v2.resourcepool.TimeoutException: A client timed out while waiting to acquire a resource from com.mchange.v2.resourcepool.BasicResou
- IT系统分析师如何学习大数据
蓝儿唯美
大数据
我是一名从事大数据项目的IT系统分析师。在深入这个项目前需要了解些什么呢?学习大数据的最佳方法就是先从了解信息系统是如何工作着手,尤其是数据库和基础设施。同样在开始前还需要了解大数据工具,如Cloudera、Hadoop、Spark、Hive、Pig、Flume、Sqoop与Mesos。系 统分析师需要明白如何组织、管理和保护数据。在市面上有几十款数据管理产品可以用于管理数据。你的大数据数据库可能
- spring学习——简介
a-john
spring
Spring是一个开源框架,是为了解决企业应用开发的复杂性而创建的。Spring使用基本的JavaBean来完成以前只能由EJB完成的事情。然而Spring的用途不仅限于服务器端的开发,从简单性,可测试性和松耦合的角度而言,任何Java应用都可以从Spring中受益。其主要特征是依赖注入、AOP、持久化、事务、SpringMVC以及Acegi Security
为了降低Java开发的复杂性,
- 自定义颜色的xml文件
aijuans
xml
<?xml version="1.0" encoding="utf-8"?> <resources> <color name="white">#FFFFFF</color> <color name="black">#000000</color> &
- 运营到底是做什么的?
aoyouzi
运营到底是做什么的?
文章来源:夏叔叔(微信号:woshixiashushu),欢迎大家关注!很久没有动笔写点东西,近些日子,由于爱狗团产品上线,不断面试,经常会被问道一个问题。问:爱狗团的运营主要做什么?答:带着用户一起嗨。为什么是带着用户玩起来呢?究竟什么是运营?运营到底是做什么的?那么,我们先来回答一个更简单的问题——互联网公司对运营考核什么?以爱狗团为例,绝大部分的移动互联网公司,对运营部门的考核分为三块——用
- js面向对象类和对象
百合不是茶
js面向对象函数创建类和对象
接触js已经有几个月了,但是对js的面向对象的一些概念根本就是模糊的,js是一种面向对象的语言 但又不像java一样有class,js不是严格的面向对象语言 ,js在java web开发的地位和java不相上下 ,其中web的数据的反馈现在主流的使用json,json的语法和js的类和属性的创建相似
下面介绍一些js的类和对象的创建的技术
一:类和对
- web.xml之资源管理对象配置 resource-env-ref
bijian1013
javaweb.xmlservlet
resource-env-ref元素来指定对管理对象的servlet引用的声明,该对象与servlet环境中的资源相关联
<resource-env-ref>
<resource-env-ref-name>资源名</resource-env-ref-name>
<resource-env-ref-type>查找资源时返回的资源类
- Create a composite component with a custom namespace
sunjing
https://weblogs.java.net/blog/mriem/archive/2013/11/22/jsf-tip-45-create-composite-component-custom-namespace
When you developed a composite component the namespace you would be seeing would
- 【MongoDB学习笔记十二】Mongo副本集服务器角色之Arbiter
bit1129
mongodb
一、复本集为什么要加入Arbiter这个角色 回答这个问题,要从复本集的存活条件和Aribter服务器的特性两方面来说。 什么是Artiber? An arbiter does
not have a copy of data set and
cannot become a primary. Replica sets may have arbiters to add a
- Javascript开发笔记
白糖_
JavaScript
获取iframe内的元素
通常我们使用window.frames["frameId"].document.getElementById("divId").innerHTML这样的形式来获取iframe内的元素,这种写法在IE、safari、chrome下都是通过的,唯独在fireforx下不通过。其实jquery的contents方法提供了对if
- Web浏览器Chrome打开一段时间后,运行alert无效
bozch
Webchormealert无效
今天在开发的时候,突然间发现alert在chrome浏览器就没法弹出了,很是怪异。
试了试其他浏览器,发现都是没有问题的。
开始想以为是chorme浏览器有啥机制导致的,就开始尝试各种代码让alert出来。尝试结果是仍然没有显示出来。
这样开发的结果,如果客户在使用的时候没有提示,那会带来致命的体验。哎,没啥办法了 就关闭浏览器重启。
结果就好了,这也太怪异了。难道是cho
- 编程之美-高效地安排会议 图着色问题 贪心算法
bylijinnan
编程之美
import java.util.ArrayList;
import java.util.Collections;
import java.util.List;
import java.util.Random;
public class GraphColoringProblem {
/**编程之美 高效地安排会议 图着色问题 贪心算法
* 假设要用很多个教室对一组
- 机器学习相关概念和开发工具
chenbowen00
算法matlab机器学习
基本概念:
机器学习(Machine Learning, ML)是一门多领域交叉学科,涉及概率论、统计学、逼近论、凸分析、算法复杂度理论等多门学科。专门研究计算机怎样模拟或实现人类的学习行为,以获取新的知识或技能,重新组织已有的知识结构使之不断改善自身的性能。
它是人工智能的核心,是使计算机具有智能的根本途径,其应用遍及人工智能的各个领域,它主要使用归纳、综合而不是演绎。
开发工具
M
- [宇宙经济学]关于在太空建立永久定居点的可能性
comsci
经济
大家都知道,地球上的房地产都比较昂贵,而且土地证经常会因为新的政府的意志而变幻文本格式........
所以,在地球议会尚不具有在太空行使法律和权力的力量之前,我们外太阳系统的友好联盟可以考虑在地月系的某些引力平衡点上面,修建规模较大的定居点
- oracle 11g database control 证书错误
daizj
oracle证书错误oracle 11G 安装
oracle 11g database control 证书错误
win7 安装完oracle11后打开 Database control 后,会打开em管理页面,提示证书错误,点“继续浏览此网站”,还是会继续停留在证书错误页面
解决办法:
是 KB2661254 这个更新补丁引起的,它限制了 RSA 密钥位长度少于 1024 位的证书的使用。具体可以看微软官方公告:
- Java I/O之用FilenameFilter实现根据文件扩展名删除文件
游其是你
FilenameFilter
在Java中,你可以通过实现FilenameFilter类并重写accept(File dir, String name) 方法实现文件过滤功能。
在这个例子中,我们向你展示在“c:\\folder”路径下列出所有“.txt”格式的文件并删除。 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16
- C语言数组的简单以及一维数组的简单排序算法示例,二维数组简单示例
dcj3sjt126com
carray
# include <stdio.h>
int main(void)
{
int a[5] = {1, 2, 3, 4, 5};
//a 是数组的名字 5是表示数组元素的个数,并且这五个元素分别用a[0], a[1]...a[4]
int i;
for (i=0; i<5; ++i)
printf("%d\n",
- PRIMARY, INDEX, UNIQUE 这3种是一类 PRIMARY 主键。 就是 唯一 且 不能为空。 INDEX 索引,普通的 UNIQUE 唯一索引
dcj3sjt126com
primary
PRIMARY, INDEX, UNIQUE 这3种是一类PRIMARY 主键。 就是 唯一 且 不能为空。INDEX 索引,普通的UNIQUE 唯一索引。 不允许有重复。FULLTEXT 是全文索引,用于在一篇文章中,检索文本信息的。举个例子来说,比如你在为某商场做一个会员卡的系统。这个系统有一个会员表有下列字段:会员编号 INT会员姓名
- java集合辅助类 Collections、Arrays
shuizhaosi888
CollectionsArraysHashCode
Arrays、Collections
1 )数组集合之间转换
public static <T> List<T> asList(T... a) {
return new ArrayList<>(a);
}
a)Arrays.asL
- Spring Security(10)——退出登录logout
234390216
logoutSpring Security退出登录logout-urlLogoutFilter
要实现退出登录的功能我们需要在http元素下定义logout元素,这样Spring Security将自动为我们添加用于处理退出登录的过滤器LogoutFilter到FilterChain。当我们指定了http元素的auto-config属性为true时logout定义是会自动配置的,此时我们默认退出登录的URL为“/j_spring_secu
- 透过源码学前端 之 Backbone 三 Model
逐行分析JS源代码
backbone源码分析js学习
Backbone 分析第三部分 Model
概述: Model 提供了数据存储,将数据以JSON的形式保存在 Model的 attributes里,
但重点功能在于其提供了一套功能强大,使用简单的存、取、删、改数据方法,并在不同的操作里加了相应的监听事件,
如每次修改添加里都会触发 change,这在据模型变动来修改视图时很常用,并且与collection建立了关联。
- SpringMVC源码总结(七)mvc:annotation-driven中的HttpMessageConverter
乒乓狂魔
springMVC
这一篇文章主要介绍下HttpMessageConverter整个注册过程包含自定义的HttpMessageConverter,然后对一些HttpMessageConverter进行具体介绍。
HttpMessageConverter接口介绍:
public interface HttpMessageConverter<T> {
/**
* Indicate
- 分布式基础知识和算法理论
bluky999
算法zookeeper分布式一致性哈希paxos
分布式基础知识和算法理论
BY
[email protected]
本文永久链接:http://nodex.iteye.com/blog/2103218
在大数据的背景下,不管是做存储,做搜索,做数据分析,或者做产品或服务本身,面向互联网和移动互联网用户,已经不可避免地要面对分布式环境。笔者在此收录一些分布式相关的基础知识和算法理论介绍,在完善自我知识体系的同
- Android Studio的.gitignore以及gitignore无效的解决
bell0901
androidgitignore
github上.gitignore模板合集,里面有各种.gitignore : https://github.com/github/gitignore
自己用的Android Studio下项目的.gitignore文件,对github上的android.gitignore添加了
# OSX files //mac os下 .DS_Store
- 成为高级程序员的10个步骤
tomcat_oracle
编程
What
软件工程师的职业生涯要历经以下几个阶段:初级、中级,最后才是高级。这篇文章主要是讲如何通过 10 个步骤助你成为一名高级软件工程师。
Why
得到更多的报酬!因为你的薪水会随着你水平的提高而增加
提升你的职业生涯。成为了高级软件工程师之后,就可以朝着架构师、团队负责人、CTO 等职位前进
历经更大的挑战。随着你的成长,各种影响力也会提高。
- mongdb在linux下的安装
xtuhcy
mongodblinux
一、查询linux版本号:
lsb_release -a
LSB Version: :base-4.0-amd64:base-4.0-noarch:core-4.0-amd64:core-4.0-noarch:graphics-4.0-amd64:graphics-4.0-noarch:printing-4.0-amd64:printing-4.0-noa