DPABI详细使用教材——数据准备、预处理流程、数据分析流程
dpabi必看内容
1、 DPABI(用于脑成像的数据处理和分析的工具箱)的下载和安装步骤
2、DPABI详细使用教材——数据准备、预处理流程、数据分析流程
3、DPABISurf的安装及使用(windows系统)
4、 DPABISurf使用教材
5、如何使用DPABI 进行核磁共振数据结果统计分析?
下面介绍 ,DPABI详细使用教材——数据准备、预处理流程、数据分析流程
严老师的官方教程:官网
DPABI版本:V4.3_200401
MATLAB版本:2018a
目录
一、数据准备
二、预处理
三、数据分析
四、关于预处理流程
五、其他设置
一、数据准备
(1)如果是数据的DICOM格式,整理成以下格式(注意文件夹的命名方式):
![]()
如果觉得不是很清楚,可以在官网下载Demo数据(http://rfmri.org/DemoData),参照里面的文件格式进行整理即可。
(2)如果数据是nii格式,整理成以下格式:
![]()
按照nii的文件格式进行整理,整理结果如下:
![]()
注意: 我的fMRI数据在一开始都是4D的nii.gz格式,结果发现在预处理途中会出现以下报错,后来把4D的nii.gz格式全部转成3D的nii格式后解决问题。严老师在视频中说4D-nii格式也是可以接收的,但在预处理的时候还是会出问题,具体还不清楚什么原因,因此建议把fMRI数据全部转换为3D-nii格式,或者直接从DICOM格式开始处理,这样可以避免以下报错。
如何将4D-nii格式转化为3D-nii格式
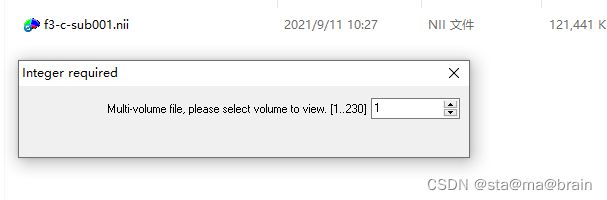
下面所放置的f3-c-sub001.nii为4D-nii文件,其为230个时间点。
1、首先下载工具箱,地址为:4D-nii格式转化为3D-nii格式
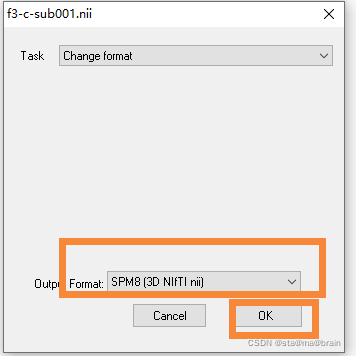
第二步,点击输出格式(Output Format)为:SPM8(3D NIFTI nii)
第三步,把fMRI的4D-nii文件拖到打开的工具箱中即可完成格式的转换
二、预处理
在MATLAB中输入“dpabi”,回车,会陆续出现以下界面,陆续点击DPARSF 5.0→DPARSF Advanced Edition,即会出现DPARSF处理界面。
![]()
![]()
![]()
首先需要输入整理好的数据,操作流程如上图所示:
①选择同时包含FunImg和T1Img数据的总目录
②输入数据的Time Point和TR(Time Point就是fMRI的volume数)
③如果输入的是DICOM数据,则勾上;如果直接输入nii数据,则把勾去掉
④填写下面的Starting Directory Name:如果输入的是****DICOM*数据,这里不用改,FunRaw就是开始执行程序的文件夹;如果输入的是nii数据,开始执行文件要改成FunImg*,然后回车,后面会弹出一些提示,看看直接跳过即可。
蓝色框框处是Template parameter,也就是处理流程的模板,一般直接用默认的就可以,不用动,这里也直接使用默认处理流程。后面会专门介绍里面每个处理流程有什么区别。下图是成功输入nii数据后的界面:
![]()
如果成功输入数据,在Participants框框里面会自动读取被试个数,如果没有成功读取,可以检查一下上面的四个步骤,尤其是下面的Starting Directory Name有没有填错。
下面是对每个预处理功能的介绍,由于DPARSF排版比较密集,下面将分开4大部分来做笔记。
PART 1
![]()
①Remove first 10 time points 必填
被试进入磁共振扫描时,梯度磁场的稳定需要一段时间,被试适应也需要一段时间,因此前几个时间点的图像噪声较多,通常选择去除4个时间点以上,默认是去除前10个。
②Slice timing 必选
层时间矫正。对大脑进行扫描时一般采用隔层扫描的方式,如先扫1,3,5,7,9层,再扫2,4,6,8,10层,也就是第1、3层的扫描时间比较早,而第2层的扫描时间比较晚,因此同一脑区的扫描时间差异较大,需要用一些方法把它们矫正到同一时间点扫描,这就是Slice timing。
③Slice number 必填
总的层数,根据自己实验数据的基本信息填写。
④Slice order 必填
扫描顺序,根据自己实验数据的基本信息填写,然后按照matlab的矩阵表达方式输入即可。
如扫描顺序是:1,3,5,7,9,11,13,2,4,6,8,10,12,则输入[1:2:13,2:2:12]。
(要留意是先扫奇数层还是先扫偶数层,如西门子机器的设置:若总层数是奇数时,会从奇数层先开始扫描,如总层数为33,扫描顺序为1,3,5,……,2,4,6,……;若总层数是偶数时,则会从偶数层先开始扫描,如总层数为32,扫描顺序为2,4,6,……,1,3,5,……)
⑤reference slice 必填
参考层,一般选择最中间那一层作为参考,如扫描层数是:1,3,5,7,9,11,13,2,4,6,8,10,12,参考层则为13(总层数为偶数时可以选择中间任意一层作为参考层)。
⑥Realign 必选
头动矫正。在实验过程中被试会有一定的头部运动,需要用一些算法进行头动矫正。做完这一步后,头部运动的结果会保存在RealigParameter文件夹下。
![]()
在后面的实验过程中需要先根据头动排除被试,头动较为明显的被试认为噪声过大,需要排除。打开RealigParameter文件夹后,可以看到有如下文件:
![]()
接着打开 ExcludeSubjectsAccordingToMaxHeadMotion.txt 后可以看到以下头动统计信息:
![]()
可以看到没有被试的头动超过3mm、旋转角度超过3°,认为三个被试的头动噪声都在接受范围以内。这个排除准则主要根据实验需要进行选择,若需要十分精确地统计,可以把排除标准升高,一般最低的排除标准是3mm和3°,下面更高的标准有2mm和2°,1.5mm和1.5°等,若不能符合最低的头动标准,那么这些被试需要被剔除。
![]()
这两个文件是所有样本的头动参数,tsv文件用excel打开。其中最后一列mean FD_Jenkinson 可以相对更好地反映头动情况,在进行组间比较的时候可以作为头动协变量把它去除。
⑦Voxel-Specific head motion 一般不选
计算很大但是作用不是很大,所以一般不选。
PART 2
![]()
①Reorient Fun* 和Reorient T1* 根据需要选择
带星号的选项都需要手动调整。这两步是手动调整功能像和结构像,包括头部的旋转、平移、原点定位等,手动摆正整个图像。好处是可以增加配准(coregistration)、分割(segmentation)和标准化(normalization)的准确度。当然如果扫描员在扫描过程中操作比较认真,一般已经基本全部对准,那么可以不选择这两项,直接跳过。
如果选择了这两项,在跑程序中途会弹出这样的界面(一个是功能像一个是结构像):
![]()
![]()
上面两张图所示被试的大脑位置和标准模板是差不多的,如果差得比较多可以通过左下的一系列参数进行调整,另外还需要把蓝色的十字架中心放在前联合的位置(和上图蓝色线所示的位置差不多),以配准原点,这一步直接用鼠标在图上点就可以。右下角QC score是对这个扫描图像的评分/评价,比如这个被试是否长了肿瘤,扫描有没有什么问题等等都可以选择评分,并进行备注,后面会专门生成一个QC文件以方便查看每一个被试的信息。最后按“Reorient image”即会自动跳到下一个被试图像。
![]()
这个功能好处是可以一个个被试进行查看并手动调整歪了的图像,但缺点是这一步会在中途直接跳出调整图像的询问窗口,如果不一个个看完就无法进入下一步。当被试太多时,每个功能/结构像都要单独看一遍,消耗的时间较长。因此若手上的数据已经初步看过或已经知道数据基本没有问题的情况下,可以直接跳过这两步。当然如果有被试的图像如下图,与标准模板相差极大,就必须执行手动调整。
![]()
②Auto mask 最好选上
For checking EPI coverage and generating group mask.
③Crop T1 一般不选,看情况
用于砍掉脖子的结构,用的很少,如果使用DICOM数据开始处理时一般不用选,如果是NIFTI格式的看情况而定,一般也已经处理好,所以很少用到这个功能。
④Bet 必选
Skullstrip,去除头皮结构,在配准时减少无关结构的影响,可以提高配准精度。(对于Linux和Mac,要运行这一步需要先安装FSL)。
⑤T1 coreg to Fun 必选
把T1像配到功能像。
⑥segment和New segment+DARTEL 必选
分割。Segment是用SPM12中的old segment,New segment+DARTEL 把图像分成灰质、白质、脑脊液,用于下一步配准到MNI空间,一般选择New segment+DARTEL。
这一步会生成以下文件夹:
![]()
打开其中一个被试的文件,会有一系列的VBM信息(voxel-based morphometry),对结构功能都感兴趣的分析可以查看这些文件:
![]()
⑦Affine regularization in East Asian/European 选European
对图像进行调整,East Asian目前只有几十个人的模板,所以一般选择European更好。
PART 3
![]()
①Nuisance covariates regression 必选
把一些噪声协变量去除,包括全局信号、灰质白质中的噪声等。
②Polynomial trend 必填
一般填1,去除线性漂移。
③Head motion 必选
调整头动参数,Rigid-body 6,很早以前会用,Derivative 12在前几年会用,现在更常用Friston 24(普遍接受),Voxel-specific 12用的不是很多,计算量很大但效果不显著。因此选择Friston 24参数模型即可。
④Head motion scrubbing regression 看情况而定
在使用Friston 24调整头动参数后,审稿人还是觉得有问题时,此时可以选上scrubbing,也可以把scrubbing的结果放在辅助材料中,说明是否使用scrubbing对结果都没有影响即可,但审稿人绝大部分不会抓头动的问题,另外scrubbing会破坏时间的连续性,在后面的处理中可能会出现一些问题,所以一般可以不选。
⑤Nuisance regressors (WM, CSF, Global)
去除白质、脑脊液等噪声信号。点开后弹出窗口如下:
![]()
WM、CSF必选,Mask有两种:
a) 使用SPM中的先验模板
b) 使用分割结果中的灰质、白质、脑脊液密度来区分
一般使用默认的SPM先验模板即可。
![]()
方法也有两种:
a) Mean:把WM/CSF信号单独平均,得到两个WM/CSF的平均时间序列作为协变量去除
b)CompCor:把WM和CSF的mask叠加起来,通过PCA提取前n个(默认是前5个)主成分作为协变量去除
![]()
global signal的设置:
a) 关于global signal是否去除有两个选项,两个都不选时则不去除global signal,如果选Both with & without…时会把去除global signal和不去除global signal都做一遍,最后的结果文件中会有标注。
b) Mask同样有两个,一般默认选择SPM先验模板
⑥Other covariates 看情况而定
其他协变量,若有其他特殊的需要去除的协变量可以勾选。
⑦Filter (Hz) 必选
绿色框的都是滤波,功能是相同的,只是执行顺序不同。如果不计算ALFF/fALFF,仅对图像进行预处理,则直接在上面选择,如果要计算ALFF/fALFF,则滤波需要放在ALFF/fALFF之后选。一般滤波范围为0.01~0.1Hz,也可以根据需要设置。
⑧Add mean back 看情况选择**(重点!!!!教程视频里没有提到!)**
如果后面打算直接分析ALFF/fALFF/ReHo等(仅与时间序列有关的分析)时,可以不选。因为前面在去除协变量时有一个减均值的步骤,而在计算时间序列之间的差异时,是否减去/保留均值都是不影响结果的,因此可以直接不选。
但如果需要对预处理后的整个图像(针对的是图像,而不是时间序列)进行后续分析(如ICA等)时,则必须要选上添加均值,否则会无法进行下一步的运算。(http://rfmri.org/faq 具体官方解释请看第4条)
后续需要对图像进行分析时,如果没有选Add mean back,观察到的图像是下图这样的:
![]()
![]()
因为没有把均值加回来,看到的都只是差异值,根本看不出来图像的样子(但是对时间序列的计算不影响)。如果把Add mean back选上加回来之后,图像才会变回来:
![]()
只有这种完整的功能图像才能够进行下一步针对图像的分析。
PART 4
![]()
①Normalize 根据需要调整
标准化。每个人的大脑具有个体差异,因此需要统一标准化到同一个空间后才可以进行相互比较。Bounding box表示装这个大脑的空间大小,一般使用默认参数即可。Voxel size同样一般默认使用[3 3 3]即可,如果自己的数据voxel是3.5或3.7mm采集的,也是使用[3 3 3]即可;若数据是使用multiband技术采集的,采集的精度会比较高,voxel已经达到2mm时则可以改成[2 2 2]。
②Normalize的方法选择
Normalize by using EPI template:把功能像配到EPI模板;
Normalize by using T1 image unified segmentation:先对T1做分割,分成白质、灰质、脑脊液,再把这些信息用到功能图像上去配准;
Normalize by DARTEL:一般使用得最多,先把结构像配到功能像空间,然后把结构像分割成灰质、白质、脑脊液,同时也生成了一些转换的矩阵,此时可以生成一个组的template,比如一个组有200个被试,也就是基于这200个被试生成一个group template,然后所有人都配到这个group template上,最后再标准化到MNI空间。
③smooth 根据情况选择
平滑。平滑的好处是可以减少配准的不准确性,提高信噪比。**注意如果要计算ReHo、Degree Centrality这两个指标,在计算之前不要选平滑,否则会影响结果。**如果仅仅是对图像进行预处理,则默认选smooth,平滑参数FWHM默认为[4 4 4]即可。
④Default mask 必选
用来确定数据处理时是在哪一个空间上进行的。
default mask:基于SPM5中的先验模板计算(默认)
User-defined mask:如需要可以在后面“…”选择前面自己生成的group mask来计算
Warp the mask into individual space:把所有的模板变到个体空间中去计算,如果不熟悉里面的参数不要选这一项。在输入文件那一步中,一开始可以选择不同的处理流程template parameter,在里面会有对应的处理流程帮助我们自动选上/不选该参数。(每个处理流程template parameter的解释在后面,这里都是直接使用默认的处理流程。)
⑤ Detrend 不选
前面(蓝色框)已做过polynomial trend去除线性漂移,所以这里可以不选。
⑥ Nuisance covariates regression 视情况而定
因为去除噪声协变量这一步即可以在前面很早就做(前面的绿色大框框,推荐在前面先去除噪声协变量的做法),也可以在配到标准空间之后再做,在这里可以根据自己需要的预处理顺序进行选择,后面这一个Nuisance covariates regression和前面的参数是一样的,点了这一步之后前面对应的参数会变成蓝色,然后根据提示设置参数即可。注意,如果normalize的时候选择了Normalize by using EPI template时,必须要在标准MNI空间做Nuisance covariates regression,也就是需要先配准到MNI空间,再去除协变量,点后面的绿色框。
至此,所有预处理流程已经选择完毕,如果仅需要对图像进行预处理,则设置到此即可,后面的勾勾可以全部删掉,直接跳到(五、其他设置)中查看如何启动预处理程序。
三、数据分析
FPARSF还有数据分析的功能,如计算ALFF、功能网络等等。
![]()
①ALFF/fALFF
ALFF是把时间序列变到频域,然后计算低频段的能量。fALFF则是用ALFF的全频段作为分母,然后normalize一下,再计算低频的能量。如果要计算ALFF/fALFF,滤波要放在ALFF/fALFF之后,选了之后上面蓝色框内的数字会变蓝色,提示参数设置和上面的蓝色框是一样的,且频率一般是0.1~0.01Hz,也可以根据需要设置,如研究特定频段的能量变化等等。
②Scrubbing
这里用的比较少,如果必须要做Scrubbing一般都选前面的head motion scrubbing regressors,除非有特殊需求,也可以在这里做。
③ReHo
局部一致性。一个节点和邻居节点的一致性,其可重复性比较高,是一个比较常用的指标。前面已提到如果要计算ReHo,那么在前面不能选择smooth,因此在计算完ReHo之后可以在这里选择smooth ReHo,如果还需要计算其他指标如Degree centrality,则可以在最后再选择Smooth Derivatives(下面的绿色框),功能是一样的,点击之后前面的FWHM参数会变蓝,提示可以在蓝色的地方设置相关参数。
④Degree centrality
某个体素/节点和其他所有体素/节点之间的连接/关系,点击后需要输入一个Threshold参数,即低于多少的功能连接我们认为是假的,默认是0.25,可根据需要设置。
![]()
⑤Functional connectivity
使用最多的功能连接。定义感兴趣区ROI后,把这些ROI与全脑所有区域做相关,即可以得到一个功能连接图。
Extract ROI time courses
提取感兴趣区的时间序列,如可以提取33个默认网络的ROI做小世界网络分析等等。
Define ROI
**在计算前两两项时,必须先在这里定义ROI。**如选择杏仁核等作为种子点来进行功能网络的分析等等。点开后会弹出以下窗口:
![]()
前面的Multiple labels in mask file表示在某个mask里面会有多个label,如某个mask有90个脑区(90个label),选择该选项之后会把90个ROI都提取出来,如果不选这一项,则仅提取有值的部分ROI,没有值的都统一默认为不是ROI,最终的ROI可能小于90个。然后可以根据自己需要选择下面的任意一种模板,当然还能选择最后一项Define other ROI,这需要根据自己读到的文献,自己研究的ROI坐标点在哪里,半径是多少,把这些信息输入即可。任意点开一项后则出现下面的ROI List窗口:
![]()
里面可以根据自己的需要添加各种ROI模板,如添加sphere信息确定的模板:
![]()
根据自己阅读的文献,在ROI List中输入x,y,z和半径即可,可以输入很多行,每一行是一个ROI,最后的Talarirach to MNI可以把早期文章的Talarirach坐标转换到现在更常用的MNI坐标。
或者其他的mask模板、种子序列模板等等,这些都基于自己所读的文献来确定。
⑥Define ROI interactive*
带星号的选项,需要自己手动选择。这一项主要是神经外科的医生使用,如医生知道哪个脑区有问题,可以直接用鼠标点。
⑦Normalize to symmetric template
这一步是在做镜像对称功能连接之前所必须的,因为人脑的左右半球并不是完全对称的,因此需要先标准化到一个更对称的模板中之后,再做体素镜像同伦连接VMHC。
至此,所有数据分析的功能已介绍完毕,可以根据自己的实验需要选择对应的分析方法。
四、关于预处理流程
前面一直提到这里使用的是template parameter中的默认处理流程,下面将对其他的处理流程做一个简单的介绍。
![]()
①默认,所有流程都在配准到MNI标准空间后,在MNI空间中计算;
②在原始空间计算,在所有预处理和指标计算全部结束以后,最后再配到标准空间
③术中磁共振(医生使用)
④VBM计算,没有扫功能磁共振,但想要计算VBM时可以使用,可以直接出来VBM的结果。(voxel-based morphometry,基于体素的形态学分析)
⑤Blank,所有清空,根据自己需要设置,自己想做哪一步就选哪一步即可。
五、其他设置
![]()
①Parallel workers #
做并行处理,由计算机/服务器中核的数量而定,核越多,计算速度越快。如是4核的计算机,填4即可。
②Functional session #
若功能像扫描了多个session,可以选择该项。并且需要把数据整理成如下格式:
Session 1 数据:FunRaw
Session 2 数据:S2_FunRaw
Session 3 数据:S3_FunRaw
……
③Starting directory name
这一个选项可以随意选择开始执行的地方,如想要换一个滤波的频段,那么前面做过的部分可以不用重复做,在修改滤波参数后,直接在这里输入从滤波这一步开始执行程序即可。如何输入对应的文件名字?在输入框内按一下回车,会弹出以下说明窗口:
![]()
根据以上说明,如需要从滤波开始执行程序,只需要在框框内输入FunImgARWSD即可,FunImgARWSD表示FunImg中已经处理完A,R,W,S,D步骤了,下面将直接开始执行F-Filter步骤。
④Save / Load
Save可以把当前设置的参数保存成一个mat文件,下次需要重复分析时可以直接Load进来,就不用每次处理时都重新设置参数,也可以对之前的设置进行查看。(不主动点save也可以,该软件已设置成每次都会自动保存一个mat文件以便查看。)
最后,点击RUN,开始执行程序。
如何使用DPABI 进行核磁共振数据结果统计分析?
详见博客地址: 如何使用DPABI 进行核磁共振数据结果统计分析?https://blog.csdn.net/xj4math/article/details/123285981