AutoDockFR:具有明确指定结合位点灵活性的蛋白质-配体对接研究进展
微信公众号参考链接:链接
引用原文:Ravindranath, Pradeep Anand et al. “AutoDockFR: Advances in Protein-Ligand Docking with Explicitly Specified Binding Site Flexibility.” PLoS computational biology vol. 11,12 e1004586. 2 Dec. 2015, doi:10.1371/journal.pcbi.1004586
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1004586
摘要:
在基于结构的药物设计中,类药物分子与受体的自动对接是一个必不可少的工具。虽然对受体的灵活性进行建模对于正-确预测配体结合很重要,但仍然具有挑战性。这项工作的重点是一种方法,在这种方法中,通过显式地预先指定一组受体侧链来模拟受体的灵活性。这种方法的挑战包括:1)搜索空间呈指数增长,需要更高效的搜索方法;2)假阳性数量增加,需要为灵活的受体对接定制评分功能。我们介绍了AutoDockFR–AutoDock for Flexible Receptor(ADFR),这是一种基于AutoDock4评分功能的新型对接引擎,它通过一种新的遗传算法(GA)和定制评分功能解决了上述挑战。我们使用Astex多样集验证ADFR,证明其GA的效率和可靠性比AutoDock4中实现的GA提高。我们证明,当配体与需要侧链构象变化的载脂蛋白受体交叉对接时,成功率大大提高。这些交叉对接实验基于两个数据集:1)SEQ17——一个包含17对apo holo结构的受体多样性集;2)CDK2–由一个CDK2载脂蛋白结构和52个已知结合抑制剂组成的配体多样性集。我们表明,当将配体交叉对接到具有多达14条柔性侧链的受体的载脂蛋白构象中时,ADFR在两个数据集上报告的交叉对接配体比AutoDock Vina更正确,在SEQ17上发现的解决方案分别为70.6%和35.3%,在CDK2上发现的解决方案分别为76.9%和61.5%。ADFR在这两个数据集的顶级解决方案数量上也优于AutoDock Vina。此外,我们还表明,在holo复合物中,正确对接的CDK2复合物在配体和移动受体原子之间平均重新产生了79.8%的成对原子相互作用。最后,我们证明了降低受体内能的权重可以提高正确对接姿势的排名,并且当增加侧链灵活性时,AutoDockFR的运行时间可以线性扩展
作者摘要:
对接程序被广泛用于识别与特定受体相互作用以抑制其功能的类药物分子。虽然已知受体在配体结合时改变构象,但大多数对接程序将小分子建模为柔性,而将受体建模为刚性,从而限制了可应用对接的治疗靶点范围。在这里,我们介绍了一个新的对接程序AutoDockFR,它通过允许大量明确指定的受体侧链探索其构象空间来模拟部分受体灵活性,同时为给定配体寻找能量有利的结合姿势。我们表明,通过在不存在配体(即载脂蛋白构象)的情况下,在实验确定的受体构象结合位点中加入受体灵活性,我们实现了更高的对接成功率。以前的方法基于对被认为是柔性的受体部分的先验和明确说明,迄今为止仅限于少量柔性蛋白质侧链(2-5),因此需要事先了解受体侧链在与给定配体结合时发生构象变化的情况。AutoDockFR在识别多达14个柔性受体侧链问题的正确解决方案方面的能力降低了这一要求。
介绍:
基于结构的计算药物设计是计算药物化学的重要工具。对接用于优化已知药物,并通过预测其结合模式和亲和力来识别新的结合物。虽然在对接过程中对配体构象空间的探索是常见的,但由于所需的计算资源,对配体结合的受体灵活性建模仍然是一个重大挑战。最近的综述对基于结构的药物设计中模拟受体灵活性的最新技术进行了出色而详细的分析。总之,受体在配体结合时诱导的运动范围从小的局部调整到大的重排。在对接计算期间,将受体建模为完全灵活的模型在计算上过于昂贵,因为在搜索期间需要探索大量的自由度。相反,提出了许多计算上可行的近似方法,可大致分为以下三类:1)改变相互作用势的方法,其中配体和受体原子之间的排斥势衰减,或代表这些势的网格变形,或者创造一个共识电位来代表受体的各种构象;2) 整体对接方法,使用一组离散的受体构象;3)诱导拟合方法,在对接过程中探索受体构象的变化。根据分类标准,一些方法可能会分为多个类别。潜在的改变方法在计算上是廉价的;然而,他们能够解释的运动范围相当有限。亲和网格的弹性变形已被证明是一种有效的计算方法,可以提高交叉对接配体进入非天然结构的精度。然而,作者观察到,这种方法在一个大受体的情况下失败了。
配体结合需要侧链构象的改变。集成对接方法不需要对现有对接代码进行任何修改,其并行性令人尴尬,并已成功用于设计HIV逆转录酶抑制剂。这种方法的成功率取决于对接配体是否存在合适的受体构象。在使用受体构象来定义在对接过程中结合的受体片段,从而探索受体构象空间的更大子集的方法中,这一限制有所减弱。诱导拟合方法在考虑受体和配体灵活性的策略上各不相同。一些方法依赖于预先计算的低能配体构象,这些构象被放置在受体结构中,或者在对接配体周围重新包装受体侧链,或者调整受体和配体构象以解决冲突。这些技术不需要预先规定受体侧链的柔性,并且可以潜在地改变大量结合位点侧链的构象。幻灯片通过最小旋转和简化评分函数的平均场优化来解决冲突,使其对虚拟筛查研究有效。除了模拟受体侧链的运动外,罗塞塔配体还可以诱导主链构象的变化,但这种方法的计算成本很高。其他方法依赖于受体部分的明确和先验说明,使其具有灵活性。这些方法探索了跨越所有可能的配体旋转和平移的溶液空间,以及配体和受体柔性部分的所有可能构象。ADFR属于这一类,我们称之为“显式方法”,因为它们需要在对接前明确说明受体的柔性部分。虽然这些方法主要关注受体侧链运动,但其中一些方法也包括有限的主干运动。显式方法的主要挑战包括:1)在随受体增加的自由度呈指数增长的解空间中,难以找到全局最小值;2)使用具有内在近似和缺陷的评分函数,评估更多潜在解决方案时产生的误报数量增加,如所述。由于这些局限性,成功使用这些程序的报告仅限于与数量相对较少的柔性受体侧链(通常为2-5条)的对接研究,这就给用户带来了选择移动侧链的负担。
AutoDock是一个广泛使用的停靠程序,允许指定灵活的侧链。然而,当受体侧链变得灵活时,其32个可旋转键的硬编码限制很容易被超过。此外,在AutoDock中实现的遗传算法(GA)对于具有超过20个可旋转键的对接复合体表现不佳。在这里,我们介绍了一个新的对接引擎——AutoDockFR:灵活受体的AutoDock(ADFR),它实现了一种新的遗传算法。我们展示了它在高维溶液空间中的应用,对应于将一个全柔性配体对接到一个具有多达14个明确指定的柔性侧链的受体。虽然ADFR的设计允许包含各种各样的受体运动,但这项工作的重点是发生在受体侧链中的受体运动,其主干运动较小。先前开发的柔性树(FT)数据结构支持对多种层次嵌套分子运动进行编码[29],并首次用于我们早期的对接软件FLIPDock[23]。AutoDockFR取代了FLIPDock,并引入了一种新的、更高效的遗传算法(GA),以及一种新的柔韧性树运动描述符,该运动描述符针对柔韧性受体侧链进行了优化。为ADFR开发的新GA引入了由GA优化的解决方案集合(即总体)的聚类概念。聚类能够维持种群的多样性,并实现有效的终止标准。这种新的遗传算法还实现了一种在遗传算法优化过程中最小化解的新策略。
在本文中,我们对该算法进行了概述,并描述了支持新遗传算法效率的关键概念。我们验证了AutoDock4评分函数的实现,并通过将Astex多样性集的配体重新连接到其天然刚性受体中,量化了其比AutoDock中实现的效率的提高。接下来,我们使用两个数据集,一个强调受体多样性,另一个强调配体多样性,展示了ADFR将柔性配体交叉对接到柔性载脂蛋白受体的能力。第一个数据集(SEQ17)包含17个不同的apo holo受体对。选择这17个系统来代表广泛的受体,并且在其载脂蛋白构象中,配体原子和受体侧链之间至少存在一次严重冲突。我们发现,与AutoDock Vina相比,当每个配体交叉对接到其受体的载脂蛋白构象时,ADFR显著提高了对接成功率,该受体具有多达14条柔性受体侧链。第二个数据集包括细胞周期素依赖性激酶受体(CDK2)的载脂蛋白构象和来自该受体的holo复合物的52个配体。52个配体与受体的载脂蛋白构象对接,具有许多从0到12不等的柔性受体侧链。我们表明,增加柔性侧链的数量可以提高对接成功率,并且当增加柔性受体侧链的数量时,ADFR比AutoDock Vina在运行时线性缩放时获得更好的成功率。对于CDK2交叉对接实验,我们还详细分析了十二个侧链中的配体在载脂蛋白构象中的柔性所引起的构象变化。我们发现,在对接的复合物中,受体侧链移动以重新产生在holo复合物中观察到的平均79.8%的原子成对相互作用。最后,我们表明,在两个交叉对接实验中,降低分数中受体内能的贡献权重会增加正确对接解决方案的排名。
方法:
算法概述:
对接程序的三个主要组成部分是:表示(即对接问题编码为一组待优化变量)、优化这些变量的评分函数以及搜索方法。ADFR将对接问题编码为描述对接解决方案的变量列表,并使用遗传算法(GA)结合Solis Wets局部搜索对AutoDock4力场进行优化。该程序的源代码以及二进制文件和所有用于复制本文中报告的计算的输入文件都可以在线获得[http://adfr.scripps.edu/]
表示法:
在ADFR中,柔性配体与具有柔性侧链的受体对接的问题被编码为一组称为基因组的变量,并表示与以下相关的自由度:(i)配体方向(旋转和平移);(ii)配体构象;(iii)受体构象(图1)。在我们的方法中,配体翻译为基因组增加了三个变量。配体的旋转用四元数来描述,四元数对应四个变量。在欧拉角上使用四元数表示,以避免框架锁奇点和稳定的旋转插值。配体构象编码为配体中可旋转键的扭转角值。因此,一个具有两个可旋转键的配体将为其构象向基因组添加两个变量。受体的构象变化目前仅限于侧链运动。每个柔性侧链将其χ角列表添加到基因组中。例如,当赖氨酸变得灵活时,它会给基因组增加四个变量。图1显示了具有两个可旋转键的配体和具有两个柔性侧链的受体的基因组示例。相关变量:
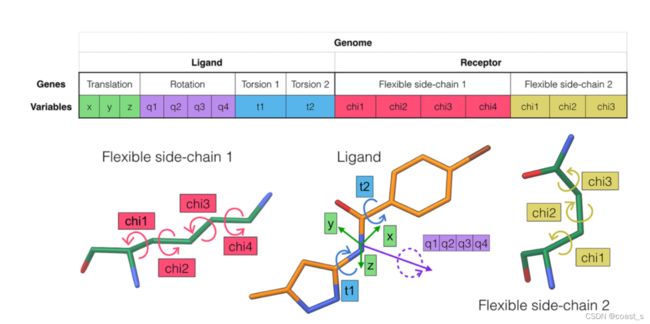
配体(Ligand):variables:变量 rotation:旋转 torsion:扭转
受体(Receptor):flexible side chain 1(柔性侧链)flexible side chain 2(柔性侧链)
图1ADFR用于编码柔性配体对接到具有两个柔性侧链的受体的基因组。该图显示了由ADFR中实施的GA优化的基因组,用于解决将具有两个可旋转键的柔性配体对接到具有两个柔性侧链的受体的问题。基因组是一组需要优化的变量。这些变量的一组给定值构成了一个对接解决方案,也称为个体。变量分为以下基因:配体翻译(把他分为三部分,3个值:x、y、z)、旋转(4个值:四元数)和构象(每个配体旋转键1个扭转角),以及受体构象(每个柔性受体侧链的χ角) 空间 旋转 扭角
n基因组被称为基因,并作为编程对象实现。例如,与配体翻译相对应的三个变量被分组在翻译基因对象中。这种面向对象的方法可以实现特定于基因的操作,用于基因值的初始化、随机化、扰动和突变。例如,配体翻译基因对象的初始化运算符从预定义的集合中随机选择翻译,而柔性受体侧链基因对象的初始化运算符使用从受体的输入构象获得的角度初始化χ角。类似地,翻译基因的突变使用以其当前值为中心的高斯分布修改基因的x、y、z值,而柔性受体侧链对象的突变算子从转子分子库随机选择一组旋转角(带偏差)。这种面向对象的体系结构有助于对搜索空间的各个维度进行集中采样(见下文),这是成功搜索大型解决方案空间的关键功能之一。基因组中变量(即基因型)的一组给定值对应于对接解决方案,可计算受体和配体原子(即表型)的坐标,并用于计算该解决方案的评分函数值。在ADFR中,基因组在运行时根据输入文件中提供的分子灵活性描述进行动态组装。使用AutoDock文件格式(即PDBQT)指定配体,该格式描述配体可旋转键。在对接设置文件中,使用残留物名称(即残留物类型和编号)指定要变为柔性的受体侧链。配体转换仅限于一组称为转换点(见下文)的可能值,这些值存储在对接设置文件中指定的文件中。
下面我们将介绍在AutoDockFR中实现的损失函数和GA,然后介绍支持GA性能的聚焦采样技术:
损失函数 :
AutoDock能量函数[30](等式1)是代表范德华、氢键、静电和去溶剂作用的项的加权和,这些项在原子对之间计算

对于刚性受体,只考虑前两项(即EL-L或配体分子内和EL-RR或配体刚性受体分子间相互作用)。当受体原子变得灵活时,额外的术语(EL-FR、EFR-FR、EFR-RR)会自动包含在评分函数中。可以为评分函数的每个项分配权重。与AutoDock类似,ADFR使用亲和图谱来表示配体或柔性受体原子与刚性受体原子之间的相互作用;因此,EL-RR和EFR-RR项可以通过在亲和映射中插值有效地获得。剩余项(EL-L、EL-FR、EFR-FR)是使用每个非键对原子的显式原子对计算的,不包括1-3个相互作用和1-4个不由可旋转键介导的相互作用。
关联贴图是在与笛卡尔轴对齐的长方体上定义的规则三维网格。这个盒子定义了配体原子可以占据的空间。在停靠之前,使用AutoDockTools套件中的AutoGrid计算亲和贴图,默认栅格贴图间距为0.375Å。为蛋白质内网格点计算的亲和力值呈现剧烈波动,最高值集中在受体原子上,原子中心周围的电位迅速下降(图2A)。我们设计了一个map后处理协议,该协议将受体内网格点上的电位替换为排斥电位,该排斥电位提供指向配体结合表面的梯度(图2B)。虽然此图显示了一个开放式口袋的示例,但相同的协议适用于埋入式空腔。该协议通过提供解决冲突的梯度,以及移除太小而无法容纳配体的掩埋有利空腔(例如陷井空腔),生成便于搜索的地图。

图2 亲和图谱处理 A) AutoDock碳亲和图的横截面。B) 同样的横截面经过贴图处理后,在蛋白质内部形成一个梯度。除了在受体内部产生一个潜在的梯度,这个过程还消除了受体体积内的局部极小值。蛋白质表面外的颜色梯度表示从弱(绿色)到强(蓝色)的良好相互作用。在蛋白质表面内部,颜色梯度表示从低(黄色)到高(红色)的不利相互作用
用于从人群中移除重复的解决方案,从而确保多样性。它还通过自动向下一代添加来自每个集群的最佳解决方案来支持自适应精英主义的实施。在我们的实现中使用聚类可以在搜索过程中同时探索和优化多个极小值。集群还可以实现下面描述的高效终止标准。通过使用最低能量、尚未聚集的溶液作为簇种子,并将相对于簇种子RMSD小于2Å(对于配体原子)的所有溶液添加到簇中来执行聚集。重复此过程,直到所有要聚集的解决方案都属于一个群集。接下来创建一个交配群体,其中包含每个集群中的最佳个体以及所有未集群的个体。每个集群中最好的个体会自动复制到下一代(适应性精英主义)。遗传算法然后选择父母进行交叉、变异和最小化以产生后代,这些后代将与其父母竞争以加入下一代群体。个体生育后代的概率与其得分成正比。选择交配的一对父母在80%以上的时间里会杂交,由此产生的后代会发生变异并最小化。在这种情况下,如果没有发生交叉(20%的时间),父母双方会发生变异并最小化以获得后代。交叉、变异和最小化的实现细节见支持信息[S1文本]。所有被创造的个体都会经历一个快速的最小化步骤。如果被最小化的个体的得分高于参考得分(迄今为止所见的最佳得分),则该个体将经历更积极的最小化,其得分将成为参考得分。最好的两个如果在交配过程中识别出的个体尚未出现在下一代群体中,则将其添加到下一代群体中。一旦下一代的种群完成(即其大小达到传入种群的大小),它就成为GA优化循环中下一代的传入种群。
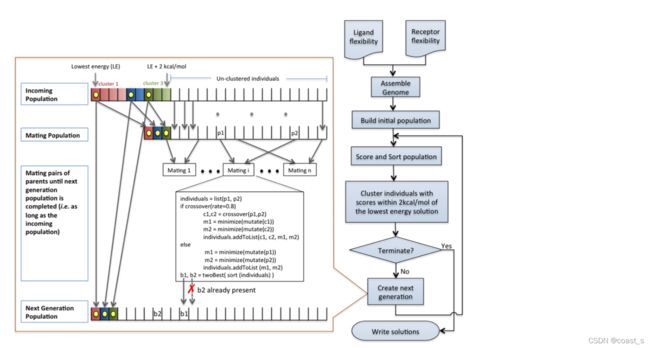
图3 ADFR的总体流程图。配体(即可旋转键)和受体(即柔性侧链)的柔性信息用于组装基因组,由此创建初始溶液列表(即群体)。对总体进行评分、排序,并对排名靠前的解决方案进行聚类。遗传算法用每个簇的最佳解为下一代播种,并通过杂交、变异和最小化交配群体中的个体来完成。当达到其中一个终止标准(最大生成数或评估数)或搜索收敛时,优化停止,此时将写出与最佳解相差1kcal/mol的解
如果连续三代的集群保持不变(即相同数量的集群,每个集群的最佳解决方案的能量保持不变),则整个种群将面临一个积极的最小化。如果集群连续五代保持不变,则认为搜索已收敛,优化停止。如果达到用户指定的限制,如最大生成数或最大评分函数评估数,优化也会停止。在优化停止后,对距离最佳解1kcal/mol以内的解进行聚类,并将每个聚类中的最佳解写入一个文件。
默认情况下,ADFR对接实验执行50个独立的GA进化,每个进化产生一个解决方案。然后对这些解决方案进行集群,以删除重复的解决方案并报告每个集群中得分最高的个体,从而形成对接解决方案的排名列表
解空间的聚焦采样
ADFR计算过程中探索的解空间非常大,减少基因组中任何变量的范围有助于搜索。在ADFR中,我们应用这一原理,将配体翻译的采样减少到更可能产生良好对接姿势的翻译子空间,从而消除已知的配体翻译,使其位于受体内部,或距离受体太近或太远。同样,所谓的“软转子流量计”(见下文)允许ADFR对受体侧链构象进行取样,类似于更频繁地在晶体结构中观察到的构象
平移点:在ADFR中,通过将配体的中心原子(称为根原子)转移到对接盒内的一个点,将配体放入受体中。对于GA中给定的对接解决方案,该翻译存储在配体翻译基因中。当创建初始群体时,群体中个体的翻译基因从一组网格点(即翻译点)中选择。通过分析碳亲和力图谱并选择亲和力为-0.3 kcal/mol或更好的位于受体体积之外的所有点来定义翻译点(图4A)。通过分析Astex多样性集合[33]中85个配合物的网格图,使用以配体几何中心为中心的尺寸为26.6Å的立方体盒,确定了-0.3 kcal/mol的亲和截止值的最佳值。该分析表明,向下采样平移点至1.125Å间距(即默认网格图间距的三倍)和亲和力小于或等于-0.3 kcal/mol的过滤点提供了结合配体中根原子覆盖率和平移点数量减少之间的最佳权衡(见图4B)。更严格的亲和性截止值将进一步减少点数,但会降低覆盖率。该方案从最初的357911个总转换点中平均产生470个转换点(最小值:60,最大值:网格点(713)对应于99.87%的平均折减系数。有趣的是,平移点的数量与对接盒中可触及受体表面积的分数成比例,而不是与盒的体积成比例。因此,对于更大的对接盒,例如用于盲对接实验的对接盒,其折减系数更为显著,其中实际结合区域未知,对接盒包含整个蛋白质结构[34–36]。当配体翻译被减少到初始群体中的一组离散点时,最小值可以自由修改翻译值,从而实现离网翻译。
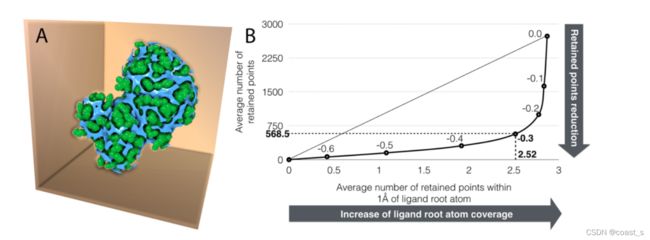
图4A) 平移点。位于蛋白质外部且碳亲和力小于或等于-0.3 kcal/mol的碳亲和力图谱的表面包围点以蓝色显示;蛋白质原子球(vdW半径减小)以绿色显示。这组平移点覆盖了可以容纳配体的凹槽和空腔,并为配体根原子提供了合理的初始放置点。B) 平移点截止值选择对于从0.0到-0.6 kcal/mol的能量截止值,以0.1 kcal/mol的减量,保留的网格点的平均数与配体根原子1Å内保留的网格点的平均数绘制。平均值是在Astex多元数据集中的85个系统上计算的。较低的能量截止值产生较少的平移点,但它们增加了丢弃配体根原子周围点的机会(即减少根的覆盖)。-0.3 kcal/mol的值最接近曲线的拐点,并被选为最佳截止值,以最大限度地减少保留点并最大限度地覆盖配体根原子.
软转子流量计。蛋白质中氨基酸的侧链主要存在于被称为转子异构体的构象子集中。ADFR使用Dunbrack编译的转子流量计库[37]在搜索过程中利用这些信息。rotamer库提供了每个氨基酸最常观察到的χ角及其偏差列表。在对接过程中,软转子异构体突变算子通过随机选择转子异构体(即,一组χ角)并根据库中提供的χ角标准偏差向这些角度添加随机偏差来修改受体侧链构象。本地搜索程序可以自由修改这些角度,从而允许它们潜在地探索0°和360°之间的所有值。因此,转子分子库用于引入代表柔性受体侧链的基因的有偏采样,而不是修剪搜索空间。
数据集:
我们在三个不同的数据集上进行了对接实验。Astex多样性集合用于验证AutoDock4评分函数在ADFR中的实现,并量化ADFR的GA相对于AutoDock使用的GA性能的提高。虽然AutoDock的性能已经使用其他数据集(如Astex清洁集)进行了基准测试[38],但我们在研究中选择使用Astex多样化集,因为它是最近才开发的,专门用于解决Astex清洁集的缺点[33]。Astex多样化套件包含85个精心策划的蛋白质-配体复合物,与Astex清洁套件没有重叠,最适合测试对接程序。我们定义了另外两个数据集,用于评估将柔性配体对接到具有明确指定柔性侧链的受体的载脂蛋白构象时的交叉对接成功率,特别是在需要解决严重冲突以正确对接配体的情况下
SEQ17交叉-对接设置。该数据集专门用于测试ADFR修改受体侧链构象的能力,从而在apo受体中实现正确的配体结合。该数据集来自SEQ数据集[39],其中包含多种受体的apo-holo对。首先,在holo复合物中确定了与配体相互作用的受体侧链(即在4Å内至少有一个成对相互作用的侧链)。然后,这些氨基酸的主链原子被用于将载脂蛋白结构叠加到全结构上,从而得到配体在载脂蛋白构象中的大致位置。接下来,来自与配体碰撞的重叠apo结构的侧链被确定为配体的重原子和受体的重原子之间的距离在对应于这两个原子的范德华半径之和的一半的距离内的侧链。这一选择产生了35个复合物。我们进一步消除了5个复合物,其中碰撞涉及主链或Cβ原子,因为这些碰撞不能通过侧链构象变化来解决。我们对剩下的30个配合物将配体重新对接到其刚性天然配合物中,并选择了最后17个系统,AutoDock Vina和ADFR都成功地将配体重新对接(RMSD<2Å)。最后一次减少是为了消除因一个或另一个程序中的评分功能限制而可能导致重新对接失败的复合物。在Cα位置观察到holo和apo复合物之间的配体相互作用的残基的最大偏差为2.19Å。在该数据集中,除了β-乳球蛋白(apo:1BSQ,holo:1GX9)。在这个系统中,载脂蛋白构象中的Leu87与配体发生冲突。该残基位于结合后重新排列的环区,导致相对于载脂蛋白结构的Cα偏差为8.23Å 。
CDK2交叉对接装置。这个交叉对接数据集的建立是为了提供大量与同一受体结合的配体。它是利用从蛋白质数据库(PDB;[40])检索到的细胞周期蛋白依赖性激酶2(CDK2)催化结构域的结构构建的。CDK2是一种参与细胞周期调节的激酶,因此可用于癌症治疗。该数据集旨在测试不同数量的侧链对一系列不同配体之间不同范围的相互作用的影响,这些配体与单个靶点结合。选择了一种高分辨率的载脂蛋白结构(4EK3,分辨率为1.34Å)以及52种配体结合的holo结构,其中一个或多个与配体相互作用的侧链相对于载脂蛋白结构呈现不同的构象。52个全息结构通过叠加主链原子与apo结构对齐,在与配体相互作用的侧链的Cα位置之间产生高达2Å的偏差。支持信息(S1表)中报告了主干偏差的详细分析。本研究中使用的PDB ID的完整列表见支持信息(S2表)
容器:
可旋转配体键由ADFR从AutoDock和AutoDock Vina使用的PDBQT文件中获得。通过在输入配置文件中列出相应的氨基酸,在ADFR计算中指定柔性受体侧链。
本文中报告的所有RMSD值均使用匈牙利匹配算法计算[41]。该算法的开源Python实现(http://software.clapper.org/munkres/)用于在计算RMSD的两个绑定姿势中找到相同类型原子之间的最佳配对。支持信息(S2文本)中描述了实现的细节。在运行对接之前,使用AutoDock Vina随机化函数对输入配体结构的位置、方向和扭转进行随机化。这可以防止搜索算法中对初始构象的可能偏差。柔性侧链在初始群体中不是随机的,以便从合理的初始受体构象开始。当载脂蛋白构象交叉对接时,这种选择不会在受体构象上产生有利的偏差。AutoDock和ADFR使用的群体规模基于以下启发:50+10×Lv,其中Lv是基因组中与配体相关的变量数量,即4(旋转)+3(转换)+NLRB(配体旋转键的数量)。支持信息(S3文本)中提供了3个数据集的结构准备细节。
Astex重新对接:AutoDock使用默认参数运行,但以下情况除外:1)包含1-4个交互,2)通过上述启发式方法获得的总体大小,以及3)每次停靠总共50次GA运行。对于每个综合体,分别执行两次AutoDock运行:一次默认能量评估次数为250万次(即AD2.5M),另一次更彻底地搜索2500万次评估(即AD25M)。AutoDock在达到指定的评估次数时终止其GA进化。ADFR是使用默认参数运行的,默认参数为50 GA运行、从上述启发式中获得的种群规模,以及包含1-4个相互作用。使用AutoGrid生成以结合配体为中心的立方网格盒(一侧26.6Å,即一侧71个点,间距为0.375Å)的亲和图谱。这些图谱用于使用AutoDock对接配体。如上文所述对图谱进行处理,以获得ADFR图谱并提取翻译点。
交叉对接。ADFR和AutoDock Vina对接计算均使用其默认参数进行。在ADFR评分函数中,受体EREC=(EFR-FR+EFR-RR)的内能按1/NFS的系数进行加权,其中NFS是柔性侧链的数量。我们使用以配体为中心的对接网格盒,大小为71×71×71点,标准分辨率为0.375Å,以便包含所有柔性侧链。ADFR和AutoDock Vina使用了相同的对接盒。通过如上所述处理AutoGrid图谱,在碳亲和图谱中识别翻译点,并生成ADFR图谱。
SEQ17。为了将SEQ17配体交叉对接到载脂蛋白受体中,我们确定侧链是柔性的,即载脂蛋白受体侧链重原子在距离配体重原子4.0Å的Cβ之外。所选柔性侧链的数量从6个到14个不等,具有11到36个可旋转的χ角。这组配体有1到16个可旋转键。支持信息(S3表)中报告了选择灵活的PDB ID和侧链。使用叠加在holo受体上的载脂蛋白结构和以holo复合物中配体几何中心为中心的对接盒计算AutoGrid图谱。AutoDock Vina以默认设置运行。
CDK2。通过将与holo复合物中配体相互作用的残基定义为与任何配体重原子4Å范围内Cβ之外至少一个重原子的残基,通过将与配体相互作用的残基制成表格来选择柔性受体侧链。由此产生的相互作用模式在数据集中差异很大,配体接触4到12个侧链之间的任何位置。在交叉对接过程中,我们选择了以下3组柔性侧链:1)名为FS4的配体(2R3I)接触的最小侧链数:Ile10、Lys33、Phe82、Leu134;最大的一组相互作用侧链(2FVD),FS12:Ile10、Val18、Lys33、Val64、Phe80、Phe82、Gln85、Asp86、Lys89、Asn132、Leu134、Asp145);最后是FS10,含有10种最常与配体相互作用的氨基酸:Ile10、Val18、Lys33、Val64、Phe80、Phe82、Asp86、Lys89、Leu134、Asp145。我们首先将52个配体交叉连接到刚性载脂蛋白结构中,然后连接到具有4、10和12个柔性侧链的载脂蛋白结构中。柔性侧链组FS4、FS10和FS12分别为基因组贡献了10、22和27个受体变量。配体中可旋转键的数量从0到13不等。使用apo结构计算以(25.8,27.6,27.5)为中心的对接盒的自动栅格地图。AutoDock Vina设置使用了相同的网格框定义,并使用默认搜索设置(Vina8,穷举设置为8)执行对接,然后运行更穷举的搜索,穷举设置为20和200(分别命名为Vina20和Vina200)
结果:
Astex多样化集合重新对接
ADFR和AutoDock4将Astex多样性组的配体重新对接到其刚性受体中,成功率如下:ADFR:74%,使用2Å的RMSD截止值,AD2.5M:77.65%和AD25M:73%。我们使用这些对接运行来验证AutoDock4评分功能的实现,并比较这两个程序的搜索引擎的性能。使用ADFR对AutoDock解决方案进行评分,反之亦然,结果相同。此外,85种溶液中有76种(89.4%)在ADFR和AD2确定的最低能量溶液之间的能量差小于0.5 kcal/mol。5米或25米。对于76和80个系统(89.4%和94.1%),两个程序都确定了相同的对接姿势(ADFR解决方案与AD2.5M和AD25M解决方案之间的RMSD分别小于2.0Å)。这些结果有力地证明了这两个项目探索相同的能源前景,从而验证了我们对AutoDock4评分功能的实现,并实现了直接的比较AutoDock4和ADFR中实现的GAs。我们使用以下三个属性比较了GA实现的性能:1)程序找到的最佳分数(即最低能量)表明了搜索方法的威力。2) 搜索引擎的效率取决于它找到解决方案的速度。对于GA算法,这与目标函数(即评分函数)的评估次数相对应。最后,3)由于GAs是随机算法,因此使用不同的随机种子数执行多次运行。GA运行识别相同最佳解决方案的次数,衡量算法的可靠性。比较用于验证两个项目探索相同能源景观的解决方案表明,两种搜索技术具有相同的能力。通过将AutoDock的评估数量从250万增加到2500万,没有发现显著的能源改善,这一事实证实,AutoDock在250万次评估之后确定了全球最小值,并且两个项目在确定几乎相同的解决方案时达到了一致。图5A显示了差异大于0.5 kcal/mol的9个系统的能量差异。只有一个系统的差异大于2.0 kcal/mol。图5B显示,在ADFR中实现的GA更有效,因为它识别了与AutoDock相同的解决方案,但每次GA进化仅使用平均81万次能量评估。只有三个综合体需要250多万次评估。每个系统所需的评估数量与基因组中变量的数量或ADFR和AutoDock获得的分数之间的能量差异均无相关性。图5C比较了2种气体的可靠性。在这个图中,85个复合物是基于50次跑步中的分数进行分类的,对于50次跑步,最终姿势与得分最高的姿势之间的误差在2.0ÅRMSD以内。AutoDock在2500万次评估中表现出更高的可靠性。然而,ADFR发现59种具有高可靠性的配合物(即蓝色和绿色条)的溶液更可靠,而AD25M和AD2分别为54和40。5米。相反,ADFR(6个复合物)中发现的低可靠性复合物(即红条)数量少于AD25M(12个复合物)和AD2。5米(15个综合体)。总体而言,ADFR中的GA实现比AutoDock中的GA实现更高效、更可靠,其终止标准能够有效限制对接期间使用的能量评估数量,同时在需要时分配更多评估。

图5 Astex多样化集合重新对接 A) 条形图描述了ADFR和AD2发现的最低能量溶液之间的能量差异。5M(暗)、ADFR和AD25M(亮)。负值表示ADFR解决方案的低能量。仅显示两个差值至少有一个大于0.5 kcal/mol的配合物。1R1H是ADFR发现的唯一比AutoDock更好的解决方案(即差值>2 kcal/mol)的复合物
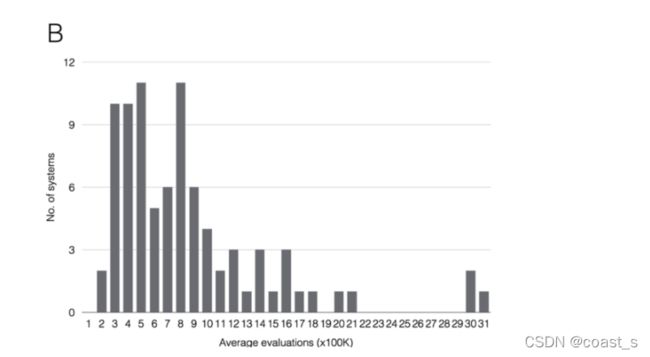
B) 该直方图显示了ADFR在导致最低能量解决方案的GA进化中执行的评分函数的评估次数分布
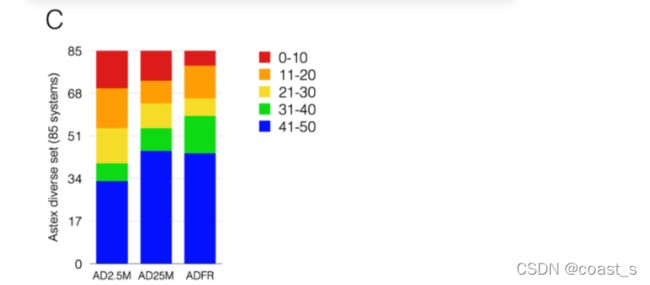
C) 每个对接由50个GA进化组成,每个进化产生一个解决方案。50种溶液以2Å的RMSD截止值聚集。在该图中,85个复合物根据最低能量溶液的簇大小进行分格,表明50次GA运行中有多少次确定了与50次运行中发现的最低能量位相对应的位姿,即GA的可靠性
交叉对接实验
AutoDock有32个可旋转键的硬编码上限,防止在用于灵活交叉对接的两个数据集上与ADFR进行直接比较。此外,已知在AutoDock中实现的遗传算法对于超过20个可旋转键的问题会失去效率。AutoDock Vina对其可以搜索的可旋转键的数量没有实现限制,并且它使用了与ADFR相同的柔性侧链的显式表示,最后,它在高维搜索方面比AutoDock具有更好的性能。因此,它为比较柔性配体与具有明确指定柔性侧链的受体对接的成功率提供了很好的参考。对于全息重对接,配体原子的实验结构和对接结构之间的RMSD值为2.0Å被广泛接受,用于识别正确对接的姿势。当将配体对接到载脂蛋白结构中时,配体的参考位置是通过叠加holo和载脂蛋白受体结构获得的。排列受两种受体构象之间的差异以及用于叠加的原子子集的影响。为了减少这些近似值,我们在apo交叉对接实验和分析中将RMSD截止值放宽至2.5Å。因此,溶液的秩为1表示最低能量溶液的RMSD小于2.5ÅRMSD,而秩为N(N>1)表示报告了N-1个假阳性溶液
SEQ17数据集 表1总结了SEQ17交叉对接结果。对于刚性交叉对接,AutoDock Vina报告了一个系统的正确解决方案:(1IKG)和两个系统的ADFR(1IT8和1Z6P)。1IT8的载脂蛋白构象中的结合袋允许其小刚性配体(RMSD 1.26Å)的翻译,这足以解决配体中的嘧啶部分与Phe229侧链之间的冲突。1Z6P配体的硝基苯甲酰基和邻苯二甲酸部分分别与Arg193和Arg310的氨基发生严重冲突。通过旋转这些基团产生1.86Å的配体RMSD,这些冲突可以在受体载脂蛋白构象的结合口袋中解决。当与柔性受体侧链对接时,排名从9提高到2。1IKG的配体通过AutoDock Vina正确对接,与Ser62的羟基氧发生严重冲突,并且非常接近Thr301主链羰基氧。AutoDock Vina通过旋转和翻译肽为配体找到解决方案(等级3,RMSD为2.27Å)在配体中键合以避免冲突,并在侧链变软时使其成为RMSD 2.45Å的1级溶液。当在载脂蛋白构象中与配体相互作用的侧链变得灵活时,两种方案的成功率都明显提高。AutoDock Vina找到了4个排名靠前的解决方案(23.5%),ADFR找到了5个(29.4%)。AutoDock Vina报告了另外两种解决方案,排名第4和第6,共有6种受体(35.3%)。ADFR报告了另外4个系统的排名为2和3(52.9%),另外3个系统的解决方案排名为14(70.6%)。表1还提供了ADFR和AutoDock Vina的最佳正确解决方案和最佳错误解决方案(Δ分数)之间报告分数的差异。这些差异为解决方案的排名提供了一定程度的可信度。一个小的Δ分数表示存在一个能量非常接近的替代姿势。
如果这一差异低于评分函数的内在误差,那么这两种解决方案是无法区分的,它们的相对排名也无法提供信息。另一方面,较大的Δ分数表明排名信息更丰富。考虑到排名前十的解决方案,ADFR成功率为52.9%,AutoDock Vina成功率为35.3%。支持信息(S4表)中提供了最佳评分配体姿势的RMSD值,以及最佳评分溶液和最低能量正确对接溶液(如果不同)的RMSD值
表1:SEQ17与受体侧链交叉对接形成载脂蛋白构象
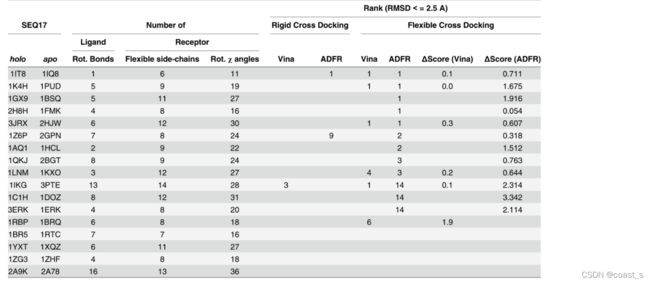
该表列出了数据集中17个载脂蛋白和全结构对的PDB ID、配体中可旋转键的数量、柔性受体侧链的数量以及相应的可旋转χ角的数量。报告了RMSD小于2.5Å的溶液的等级。空单元格表示在此RMSD截止值内未找到任何溶液
表2:0,4,10,12柔性受体侧链的ADFR和AutoDock Vina的交叉对接结果比较

左侧部分(“柔性侧链”)显示了在每次对接中被视为柔性的侧链的数量以及相应的可旋转χ角的数量。右部分报告了ADFR和AutoDock Vina发现的最低能量溶液的配体RMSD小于2.5Å(排名1)或正确对接的溶液在前10个溶液中(排名<10)的系统数量(和百分比)。
CDK2.我们将来自CDK2数据集的52个配体交叉对接到载脂蛋白结构中,该结构具有不同程度的受体柔性,从0到12个柔性侧链不等。交叉对接结果总结在表2中。该表显示了排名靠前的解决方案(排名1)正确的复合物百分比,以及排名靠前的十个解决方案中正确解决方案的系统百分比。ADFR在所有测试中都优于AutoDock Vina。在刚性交叉对接中,相对较少的自由度(7到20之间)不太可能是AutoDock Vina性能较低的原因。因此,更好的ADFR结果表明,与AutoDock Vina中实现的功能相比,AutoDock评分功能可能对口袋形状中的小扰动不太敏感。随着结合位点受体灵活性的增加,两个程序的整体对接性能都有所提高。所有柔性侧链组(FS4、FS10和FS12)的结果表明,使用“最佳排名”和“前10名结果”指标,ADFR始终获得更好的成功率。支持信息(S5表)中提供了最佳得分姿势和最佳得分正确对接姿势(如果不同)的RMSD值。AutoDock Vina耗尽性的增加并未显示出成功率的改善,在某些情况下,结果甚至更糟。
效率和复杂性考虑:与AutoDock不同,AutoDock Vina使用的评分功能与ADFR不同。此外,它没有报告该评分功能的评估次数,因此无法在效率方面进行直接比较。在XEON-EMT处理器上,在单核上与12个柔性受体侧链对接的平均壁时间为:AutoDock Vina的耗竭性为8,平均壁时间为1.85小时;Python实现ADFR的平均壁时间为8.5小时。AutoDock Vina的运行时间随着RCD、FCD4、FCD10和FCD12的柔性受体侧链数量的增加而呈指数增长,平均时间分别为1.8、13.0、61.2和111.3分钟。另一方面,对于FCD4、FCD10和FCD12,ADFR的运行时间从RCD的平均4.2小时线性增加到每个GA进化的4.8、7.3和8.6小时。图6显示了两个程序的柔性对接运行时间(FS4、FS10和FS12)与刚性对接运行时间的比率,使用在52个系统上计算的平均运行时间。使用带有默认搜索设置(Vina8)的AutoDock Vina,对接到带有12条柔性侧链的接收器所需的时间是对接到刚性接收器所需时间的60多倍。更高的耗竭性设置Vina20和Vina200将运行时间分别比默认的Vina8增加了2倍和20倍以上(支持信息–S7表)。另一方面,具有12条柔性侧链的ADFR运行时间仅为刚性对接运行时间的两倍。
受体侧链运动:每个柔性受体侧链在输入结构的构象中开始对接(即交叉对接中的载脂蛋白构象)。这种构象在对接过程中由变异算子和局部搜索过程进行修改。突变使用χ角和转子流量计库的偏差。另一方面,局部搜索程序可以自由地修改这些χ角。柔性受体侧链的子集也可以通过交叉操作在个体之间交换。我们分析了在FS12对接运行期间通过遗传算法优化的所有种群,以深入了解在进化过程中改变轮状体状态的侧链数量。如果侧链的至少一个χ角偏离输入结构至少50°,则认为侧链构象发生了变化。该值对应于ADFR使用的转子流量计库中χ角的最小差异。对所有52个复合体的所有50次运行中所有世代的数据进行分析,揭示了GA的一个有趣的涌现特性。虽然可以看到最多12个修饰侧链的个体,但非常罕见,但每个个体的平均修饰侧链数为5.6(共12个)。图7显示了GA连续世代中修饰侧链数量的典型分布。该图显示,该数量从0(在初始种群中)快速上升到平台。这一行为与Gaudreault及其同事[39]的分析一致,他们的分析报告称,只有5条或更少的侧链在配体结合时在60°角截止范围内改变了它们的旋转体构象

图6根据柔性受体侧链的数量调整对接运行时间。Yaxis代表刚性交叉对接运行时的倍数。此图中使用的时间是CDK2交叉对接实验的52个复合体在所有对接运行中的平均值。对于AutoDock Vina,使用与默认耗尽8对应的时间。X轴表示柔性受体侧链的数量。当12个蛋白质侧链变为柔性时,ADFR的伸缩系数为2,而Vina8的伸缩系数为62
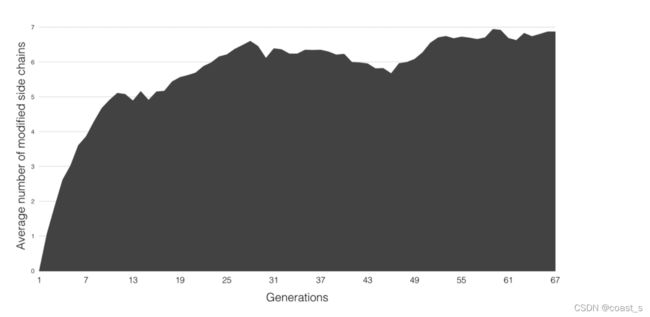
图7在将4EK6配体成功对接到具有12条柔性侧链的相应4EK3 apo受体期间,GA群体中受体侧链的变化频率。该图描绘了在连续几代GA优化过程中,具有修改构象的受体侧链的平均数量的演变。在初始群体中,所有受体侧链均为载脂蛋白构象。在优化种群的个体中,改变轮状体状态的侧链数量在最初几代中迅速增加,并达到一个稳定期。该剖面是典型的,在所有系统的所有运行中都可以观察到
柔性受体侧链与配体的相互作用。虽然我们的交叉对接实验成功的衡量标准是相对于叠加到载脂蛋白结构上的近似配体的配体原子RMSD,但了解柔性受体侧链在结合配体时如何改变构象也很重要。图8提供了apo结构(4EK3)、holo复合物(1YKR)和最佳对接溶液(具有12条柔性侧链的apo受体)之间受体构象变化的示例。图8A显示,apo和holo结构之间的Cα位置没有显著偏差。 载脂蛋白构象中的Lys33和Lys89与完整构象中的配体严重冲突,从而阻止其成功交叉对接到刚性载脂蛋白受体(最佳能量溶液的RMSD值为5.96Å)。此外,Asp132和Glu85被翻转,而剩余的侧链在配体结合时保留其载脂蛋白构象。图8B显示了载脂蛋白结构中的对接配体。所有柔性侧链都已调整位置。特别是,这两种赖氨酸的构象更接近其全构象,从而使配体能够与0.34Å的RMSD结合。图8C显示了与holo复合体对接的解决方案。这两种赖氨酸已接近其完整构象。Gln85构象的差异可能是因为在X射线结构中,氧和氮的电子密度的相似性限制了Asn和Gln的酰胺基分配正确侧链方向的能力。在对接溶液中,由于Gln85与Lys89的相互作用(或缺乏相互作用),Gln85在holo和apo结构之间翻转其氨基。此外,Gln85的胺基在全息结构中与水分子形成氢键。在对接溶液中,尽管Lys89更接近其全构象,但该侧链仍保持接近其载脂蛋白构象,并与配体磺酸基相互作用形成氢键。因此,Gln85采用的构象可能是由于受体能量项的权重降低,以及对接过程中缺少明确的水分子。下面描述了一组成功交叉对接溶液中侧链与配体相互作用的更详细分析。
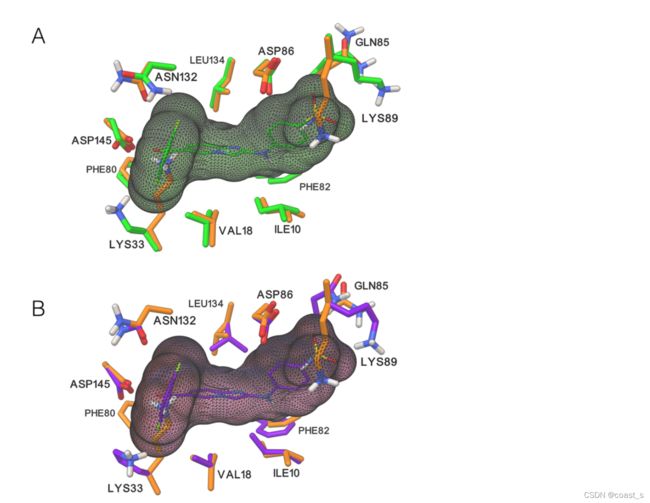

图8 比较apo、holo和成功对接溶液的侧链构象。该图对载脂蛋白(4EK3)、holo复合物(1YKR)和1YKR配体对接溶液的构象进行了两两比较,其中12条柔性受体侧链显示为球状和棒状。A) Apo vs.holo:天然结合配体显示为带有绿色碳原子的棒状物,以及部分透明的绿色分子表面。载脂蛋白构象中的2个赖氨酸侧链与配体占据的空间严重重叠。B) 对接对apo。对接溶液显示为紫色碳原子和部分透明的配体分子表面。载脂蛋白的结构用橙色的碳原子表示。艾尔对接溶液中的2条侧链采用不同于初始apo构象的构象。它们中的大多数都满足于对应于微小调整的构象,而另一些则采用实质上不同的构象来解决空间冲突(Lys33和Lys89)。C) 对接溶液(紫色碳原子)与全息受体(绿色碳原子)一起显示。配体完美对接(晶体结构的RMSD为0.34Å),受体侧链改变其构象,以适应正确结合模式下的配体结合
我们考虑了不同的指标,以评估对接12个柔性侧链(FS12计算)的apo结构时诱导配合建模的成功性。我们发现,移动受体原子的RMSD不能很好地测量受体侧链运动的性质(见讨论部分)。相反,我们将holo复合体中的原子成对相互作用制成表格,并分析43个系统中这些相互作用的恢复率,其中ADFR成功地生成了至少一个正确的溶液(RMSD<2.5Å)。我们将一对移动配体的受体原子定义为相互作用,如果它们彼此的距离在5Å以内。在配对原子相互作用时,考虑了配体和受体原子的对称性。图9显示了用于该分析的43个配合物在对接溶液中再现的全息成对原子相互作用的百分比。在该热图的一个单元中,100%(绿色)的速率表明配体和侧链原子之间的每一个成对原子相互作用都在对接溶液中重现。平均而言,每个对接姿势都能再现79.8%的成对全息交互,其中至少有57.1%的交互(对于3DDQ)。对于每个姿势,显示其在聚类前的50个解决方案中的排名
在再现全息相互作用模式方面的一些失败可归因于apo和全息之间的主链原子偏差。Ile10、Val18和Lys33的偏差值最高(约2Å)。Lys33和Ile10的侧链具有足够的灵活性来适应这种变化。另一方面,Val18只有一个χ角,使两个碳原子在一个平行于包含配体原子的平面上旋转。因此,Val18上的侧链柔性无法补偿骨架从载脂蛋白结构中观察到的配体转移。
对于45.1%的移动侧链(穿过43个复合体),在全息复合体中观察到的所有相互作用都以对接姿势再现,89.7%的侧链至少再现他们一半的全息互动。在holo结构中,11.2%不与配体接触的受体侧链与配体(灰细胞)产生至少一次相互作用。对接溶液中的大多数额外相互作用由Lys33(14)、Gln85(16)和Asn132(32)解释。对于这些侧链,与配体弱相互作用的益处大于这些侧链在载脂蛋白构象中产生的缩小的受体-受体相互作用

图9以对接姿势再现配体-柔性受体-原子接触的热图。本表中报告的43个系统是ADFR正确报告对接溶液的系统(即配体RMSD<2.5Å)。报告了解决方案在50次GA运行中的排名。白细胞对应于柔性侧链,不与holo或对接复合体中的配体相互作用。灰色细胞表示对接溶液中形成的相互作用,而holo复合体中不存在这种相互作用。剩余的细胞用红到绿的色标着色,表示对接溶液复制的全息相互作用原子对的百分比。绿色细胞(100%)表示配体原子和对应于该细胞的残基的侧链原子之间的每一个成对原子相互作用都在对接溶液中重现。柱状图显示了每个配体在所有12条侧链上复制的全息相互作用的百分比。该配体至少复制了holo复合物中所有相互作用对的57.1%,平均79.8%的相互作用
当移动的受体原子的数量大大超过配体原子时,降低受体内能的影响
当移动的受体原子数量远远超过配体原子时,受体内能成分(EREC=EFR-FR+EFR-RR)占主导地位。在没有纠正的情况下,这导致GA主要优化柔性受体的构象,而不是配体-受体相互作用(ELIG=EL-FR+EL-RR)。我们的结果表明,通过柔性侧链数量的倒数来降低受体贡献EREC的权重(例如,对于FS12集合,EREC/12)会导致正确解的等级整体提高。图10A和10B分别显示了SEQ17和CDK2 FS12交叉对接的第一个正确解决方案的排序秩,没有(蓝色)和(绿色)加权EREC值。我们观察到排名和排名的整体改善,在考虑前10个解决方案时,CDK2数据集的成功率从61.5%提高到76.9%,SEQ17的成功率从35.2%提高到52.9%。同时,通过降低受体内能的权重,对接溶液中的相互作用能显著提高。改善范围为1至7 kcal/mol(图10C和10D),大多数配合物增加3至4 kcal/mol。这一结果支持这样的观点,即在不降低受体内能成分的情况下,GA无法优化配体-受体相互作用,从而增加假阳性率。它还表明,对不同的能量项(即EREC与ELIG与EREC-LIG)应用不同的权重,可以改变搜索引擎的焦点。在对接非常大的配体(如肽)的初步结果中,我们观察到了一种相反的情况,即配体内能占主导地位。因此,需要更复杂的方法来平衡不同原子组的能量贡献。我们目前正在制定一个更详细的方案,用于规范化评分函数的各种术语,并导出适用于评分函数的这些规范化术语的适当权重
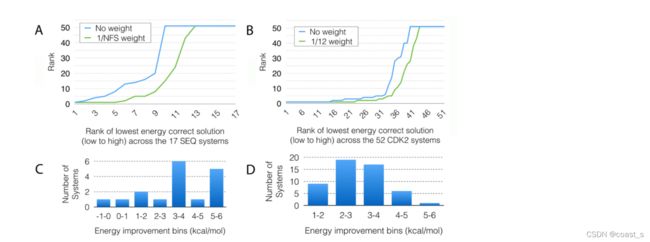
图10降低受体内能的影响。A)和B)对于SEQ17和CDK2 FS12交叉对接计算,在不缩放受体能量(蓝色)和缩放因子为1/NFS(绿色)的情况下,正确对接解决方案的排序,其中NFS是柔性受体侧链的数量。总的来说,降低受体能量的权重可以提高能量最低的正确解决方案的级别。图中的顶部水平线(排名51)表示在50次对接运行中未找到解决方案的数据点。C)和D)受体-配体相互作用能(ER-L)改善的分布,单位为kcal/mol,当受体的内能分别在SEQ17和CDK2 FS12计算的评分函数中被向下加权时
讨论
在本文中,我们介绍了一种新的对接软件AutoDockFR(或ADFR),用于将柔性配体对接到具有明确指定柔性的受体中。我们证明,ADFR在各种对接实验中优于广泛使用的对接程序AutoDock和AutoDock Vina,并引入了两组需要受体侧链发生实质性构象变化的蛋白质复合物,以便将配体成功对接到载脂蛋白构象中。
自3.05版以来,AutoDock可以明确处理柔性受体侧链,但已知其GA在少于20个键可旋转时表现最佳,有效地将此选项限制为1到2个柔性侧链。将柔性配体重新对接到Astex多样性集合中,可以定量评估ADFR中实现的新GA与AutoDock中实现的新GA之间的差异,从而提高效率和可靠性。这种性能的提高是通过以下技术的组合实现的:群体聚类、有效的终止标准,以及知识的编码,如平移点和软转子流量计,帮助遗传算法更快地识别好的解决方案。其他对接软件(如Gold,Fitted)使用类似于平移点和转子流量计的技术来修剪搜索空间。然而,在ADFR中,该信息用于更频繁地对搜索空间中有希望的区域进行采样,同时保留对整个搜索空间的连续采样,而不是对其进行修剪。我们在两个数据集上进行了交叉对接实验。SEQ17数据集关注受体多样性,而CDK2关注配体多样性。这两种情况都是相关的对接场景,选择载脂蛋白构象作为目标提供了与诱导拟合模拟相关的挑战的现实场景
SEQ17数据集由17个受体组成,其中apo构象中的大量侧链运动对于对接配体是必要的。在受体的刚性载脂蛋白构象中观察到的大量失败的交叉对接证实了SEQ17提供了一组具有挑战性的复合物。交叉对接实验表明,增加受体的灵活性可以提高对接成功率。ADFR报告了70.6%的络合物溶液,52.3%的络合物溶液的等级低于10,优于AutoDock Vina 35.3%的成功率。对评分函数的改进可以进一步提高ADFR未来的对接成功率。特别是,在柔性受体侧链中添加可旋转的末端氢原子将提高该数据集上的对接精度,因为大量移动的受体侧链包含此类氢原子
CDK2数据集的交叉对接显示,ADFR的成功率更高,与AutoDock Vina相比有了实质性的改进。这种对接方案不同于SEQ17数据集,因为我们使用一组52个配体对接到受体的单一载脂蛋白构象中。我们表明,当我们以CDK2载脂蛋白受体的构象与0、4、10和12个柔性侧链交叉对接52个CDK2配体时,ADFR在柔性对接的所有情况下都优于AutoDock Vina。我们对受体侧链运动以及增加受体灵活性对对接成功率的影响进行了详细分析
受体侧链运动分析
移动受体原子的RMSD不是了解受体侧链运动的合适指标,原因如下。首先,在许多情况下,包括这里使用的SEQ17和CDK2数据集,只有一小部分与配体相互作用的受体侧链的构象发生了实质性变化。这些侧链对移动受体原子的RMSD的贡献超过了大量侧链保持接近其初始构象的贡献。第二,计算RMSD需要参考构象,这是成功实现的目标构象。理想情况下,当配体对接成载脂蛋白构象时,应诱导全构象。然而,这需要受体具有充分的灵活性。在我们的实验中,只有与配体相互作用的侧链才能移动。这些侧链存在于载脂蛋白构象中;因此,并不总是期望它们实现全息构象。例如,没有与特定配体相互作用的侧链没有理由偏离它们的载脂蛋白构象。此外,即使是很小的骨架扰动也可能改变Cα-Cβ载体,从而潜在地迫使侧链采用替代构象与配体相互作用。基于这些原因,我们使用holo复合物中配体和移动受体原子之间的成对原子相互作用来评估受体侧链的运动。结果表明,平均79.8%的这些原子成对相互作用在对接溶液中恢复,表明柔性受体侧链移动以重新创建在holo复合物中观察到的相互作用模式
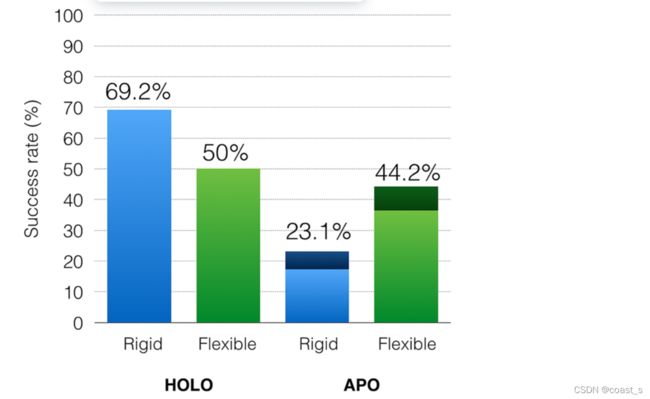
图11当配体与天然holo受体和apo受体对接时,使12个受体侧链具有柔性的影响。当使原生holo受体具有灵活性时,观察到预期的准确性损失,这反映了评分功能和搜索方法的缺陷。然而,增加载脂蛋白受体的灵活性可以提高对接成功率。显示配体RMSD<2Å的全息对接成功率。apo交叉对接的成功率从17.3%增加到36.5%,截止值为2.0ÅRMSD。当使用2.5ÅRMSD截止值(较暗的阴影条)时,成功率从23.1%增加到44.2%
增加受体灵活性对对接成功率的影响
图11显示了ADFR在刚性对接到载脂蛋白和载脂蛋白结构并带有12个柔性侧链时实现的成功率(即顶级溶液的晶体结构RMSD小于2Å(holo)和2.5Å(apo))的CDK2络合物百分比)。在将配体对接到刚性天然holo受体(69.2%)与将其对接到具有柔性侧链(50%)的同一受体之间,可以观察到预期的性能下降。这可归因于模型的缺陷(例如,隐式溶剂、评分函数限制),导致误报超过正确的解决方案评分。然而,在apo结构中加入柔性侧链可以显著改善对接配体的结果,将成功率从23%提高到44%
与将配体对接到天然或非天然holo结构中相比,将配体对接到apo结构中是一项更具挑战性的任务[43],这代表了一种现实场景,即评估候选分子结合到给定结构中的能力。两项交叉对接试验均采用载脂蛋白构象进行。此外,处理多达12条侧链的能力减少了在运行停靠计算之前必须选择哪些侧链应被视为灵活的负担。SEQ17和CDK2数据集代表了特定但相关的受体构象变化类型。对于AutoDock Vina和ADFR来说,它们是一个挑战。比较引言中描述的处理这些数据集上受体灵活性的各种其他方法的优点将很有趣,但超出了本文的范围
方法开发的开放式体系结构。ADFR的开放式体系结构旨在整合各种运动对象,我们正在为局部和全局运动添加运动操作符。这种体系结构支持探索新技术,但python实现在执行时间上表现不佳。ADFR的当前实现比在具有12个柔性侧链的受体中对接时的AutoDok VINA的高度优化的C++代码的平均速度慢230倍。目前,一个GA进化过程平均需要8.5个小时,用于具有12个柔性侧链的受体。因此,此时,ADFR仅适用于能够访问大量计算资源(即大型集群)的用户,以便在不同的处理器上并行执行独立的GA进化。我们正在研究一种C++实现,它将极大地减少执行时间。
总之,我们证明,增加受体的载脂蛋白构象的灵活性可以提高对接成功率,并且ADFR在多达12条柔性侧链的受体上优于AutoDock Vina。对于明确指定受体灵活性的对接方法,处理如此大量的侧链的能力减轻了预测或任意挑选少数需要在配体结合时改变其构象的侧链的负担。评分功能和受体灵活性表现的未来改进可能会进一步提高成功率。特别是,我们计划对可旋转配体键进行集中取样,并公布特定侧链发生构象变化的倾向,以简化搜索。受体侧链上所有末端氢原子的完全柔性表示也将提高评分函数的准确性。最后,添加新的运动描述符,包括局部主干运动以改变Cα-Cβ键方向,以及全局运动以模拟环和域运动,将增加该软件可以成功使用的治疗靶点范围。该软件可在以下网站免费获得开源许可证:http://adfr.scripps.edu/以及重现本文计算所需的所有数据。