【数学建模】2022数维杯国际赛C题 如何利用脑结构特征和认知行为特征诊断阿尔茨海默病(How to Diagnose Alzheimer‘s Disease)
2022数维杯国际赛C题 如何利用脑结构特征和认知行为特征诊断阿尔茨海默病
- 1 前言
- 2 问题重述
- 3 问题一求解
-
- 3.1 数据预处理
- 3.2 模型训练
-
- 3.2.1 逻辑回归
- 3.2.2 SVM
- 3.2.3 KNN
- 3.2.4 决策树
- 3.2.5 XGB
- 3.2.6 LGB
- 3.2.7 CB
- 3.3 各模型的对比
- 4 问题二求解
-
- 4.1 数据处理
- 4.2 数据探索
- 4.3 相关性分析
- 5 问题三求解
-
- 5.1 K值的选择
- 5.2 随机种子选择
- 5.3 Kmeans聚类
- 6 问题四、问题五求解
-
- 6.1 问题四
- 小结
- 附件
竞赛题目:如何利用脑结构特征和认知行为特征诊断阿尔茨海默病
How to Diagnose Alzheimer’s Disease Using Brain Structural Features and Cognitive Behavioral Features
1 前言
本文档主要包括了2022年数维杯C题的求解过程,包含了代码以及运行成果的讲解,下图为本文的中文摘要

2 问题重述
- 问题1: 对附件数据的特征指标进行预处理,考察数据特征与阿尔茨海默病诊断的相关性。
- 问题2:利用所附的脑结构特征和认知行为特征,设计阿尔茨海默病的智能诊断。
- 问题3:首先,将CN、MCI和AD聚类为三大类。然后,针对MCI中包含的三个子类(SMC、EMCI和LMCI),继续将聚类细化为三个子类。
- 问题4:附件中的同一样本包含了不同时间点采集的特征,请将其与时间点的关系进行分析,以揭示不同类别疾病随时间的演化规律。
- 问题5:请查阅相关文献,描述CN、SMC、EMCI、LMCI、AD五类疾病的早期干预及诊断标准。
注意:在本文中首先求解了第二问,然后利用第二问的结果对第一问进行了分析
3 问题一求解
3.1 数据预处理
df = pd.read_csv('./data/ADNIMERGE_New.csv')
df

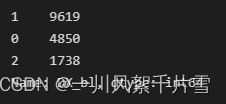
在给定的数据集中包含116列数据,共计16222条样本。在本问题中,我们将DX_bl列作为标签列,其余155列作为特征列。其中标签列共包含5种类别,分别是:CN、SMC、LMCI、EMCI和AD。如下图所示,包含CN样本4850个,SMC样本1416个,LMCI样本5236个,EMCI样本2968个,AD样本1738个。在115个特征中,数值特征共计93个,类别特征共计22个。
missingno为一款缺失值可视化Python工具库,使用它来查看训练集和测试集的缺失值,如下图所示。
# 查看数据缺失值分布概况
import missingno as msn
msn.matrix(df)

上图中,白色部分代表缺失值,空白越多代表缺失越严重。从上图中可以看到,部分特征存在着严重的缺失值,我们在这里删除了缺失值数量大于样本数量百分之30的特征。删除之后数据的缺失值分布如下:
# 删除缺失值大于百分之30的数据
df_dropna = df.dropna(axis=1, thresh=11355)
df_dropna

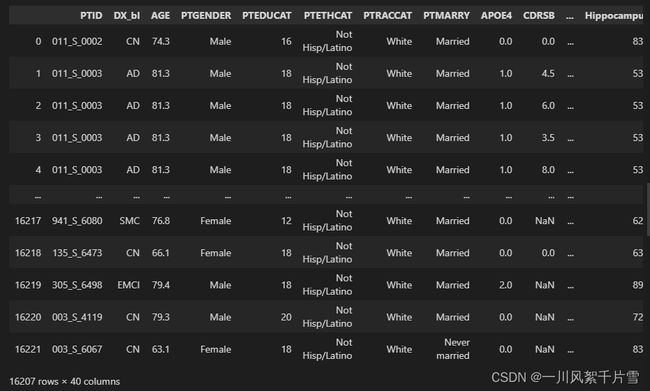
然后,删除DX_bl列和PTMARRY列为空的样本,剩余的数据的形状为:16207 rows × 52 columns。此外,通过对特征内容的进一步分析,人为删除了以下对分类任务没有重要作用的特征:‘RID’, ‘COLPROT’, ‘ORIGPROT’, ‘SITE’, ‘VISCODE’, ‘EXAMDATE’, ‘DX’, ‘EXAMDATE_bl’, ‘FLDSTRENG_bl’, ‘FSVERSION_bl’, ‘IMAGEUID_bl’, ‘update_stamp’。此时,剩余数据的形状为:16207 rows × 40 columns。
# 删除DX_bl列为空的样本
df_dropna = df_dropna.dropna(subset=['DX_bl'])
# 删除PTMARRY列为空的样本
df_dropna = df_dropna.dropna(subset=['PTMARRY'])
# 手动删除部分特征
df_new = df_dropna.drop(columns=['RID','COLPROT','ORIGPROT','SITE','VISCODE',
'EXAMDATE','DX','EXAMDATE_bl','FLDSTRENG_bl','FSVERSION_bl','IMAGEUID_bl','update_stamp'], axis=1)
df_new
首先,对特征’PTID’进行统计,计算得到目前该数据集中共计2410名病人的数据。然后,对特征’PTID’进行LabelEncoder编码,对剩余的类别特征做以下映射。
| 映射方法 |
|---|
| ‘CN’:‘0’, ‘SMC’:‘1’, ‘EMCI’:‘2’, ‘LMCI’:‘3’, ‘AD’:‘4’ |
| ‘Female’:‘0’, ‘Male’:‘1’ |
| ‘Not Hisp/Latino’:‘0’, ‘Hisp/Latino’:‘1’, ‘Unknown’:‘2’ |
| ‘Am Indian/Alaskan’:‘0’, ‘Asian’:‘1’, ‘Black’:‘2’, ‘Hawaiian/Other PI’:‘3’, ‘More than one’:‘4’, ‘Unknown’:‘5’, ‘White’:‘6’, |
| ‘Divorced’:‘0’, ‘Married’:‘1’, ‘Never married’:‘2’, ‘Unknown’:‘3’, ‘Widowed’:‘4’ |
# 统计PTID的个数
df_new['PTID'].value_counts()
Name: PTID, Length: 2410, dtype: int64
# 对PTID进行labelEncode操作
from sklearn import preprocessing
lb = preprocessing.LabelEncoder()
lb =lb.fit(df_new['PTID']) #训练LabelEncoder
df_new['PTID']=lb.transform(df_new['PTID'])
df_new
# 对类别特征进行编码映射
dict = {
'CN':'0', 'SMC':'1', 'EMCI':'2', 'LMCI':'3', 'AD':'4',
'Female':'0', 'Male':'1',
'Not Hisp/Latino':'0', 'Hisp/Latino':'1', 'Unknown':'2',
'Am Indian/Alaskan':'0', 'Asian':'1', 'Black':'2', 'Hawaiian/Other PI':'3', 'More than one':'4', 'Unknown':'5', 'White':'6',
'Divorced':'0', 'Married':'1', 'Never married':'2', 'Unknown':'3', 'Widowed':'4'
}
df_new['DX_bl'] = df_new.DX_bl.map(dict)
df_new['PTGENDER'] = df_new.PTGENDER.map(dict)
df_new['PTETHCAT'] = df_new.PTETHCAT.map(dict)
df_new['PTRACCAT'] = df_new.PTMARRY.map(dict)
df_new['PTMARRY'] = df_new.PTMARRY.map(dict)
再次查看数据的缺失值分布
msn.matrix(df_new)
# 缺失值填充
from sklearn.impute import SimpleImputer
df_new= SimpleImputer().fit_transform(df_new)
df_new = pd.DataFrame(df_new)
df_new
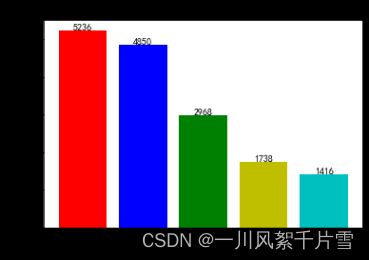
如下图所示,此时各类别疾病的数量为:LMCI: 5236, CN: 4850, EMCI: 2967, AD: 1738, SMC: 1416。,保存数据,记为df_new_PTID.csv
df_new.to_csv('./data/df_new.csv', index=False)
3.2 模型训练
df = pd.read_csv('./data/df_new_PTID.csv')
df
值的注意的是,数据集中包含同一病人的多条样本,为了避免数据发生穿越,因此按照以下规则进行数据划分,如下图所示。
# PTID小于1687的70%的病人划分到训练集
train = df[df['PTID'] < 1687]
test = df[df['PTID'] >= 1687]
训练集:病人ID小于1687的。11425条样本。
测试集:病人ID大于等于1687的,4782条样本。

下边的代码为训练集和测试集数据和标签的划分,另外在本文的实验中,所采用的模型均使用默认参数。
train_data = train.drop(['DX_bl','PTID'],axis=1)
train_target = train['DX_bl']
test_data = test.drop(['DX_bl','PTID'],axis=1)
test_target = test['DX_bl']
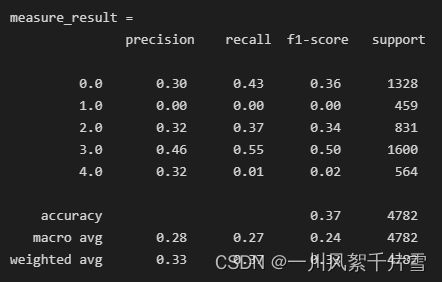
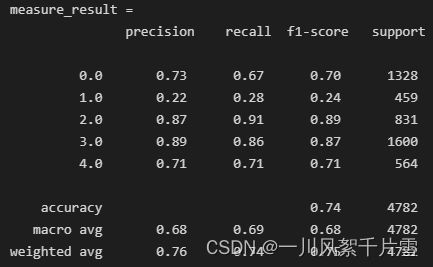
3.2.1 逻辑回归
# 逻辑回归
from sklearn.linear_model import LogisticRegression
clf = LogisticRegression()
clf.fit(train_data, train_target)
test_pred = clf.predict(test_data)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
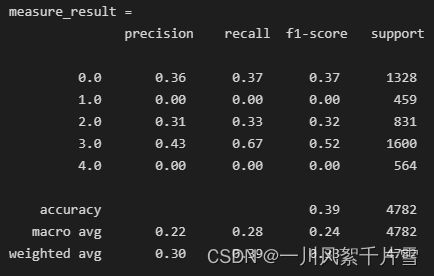
3.2.2 SVM
# 线性可分支持向量机
from sklearn.svm import LinearSVC
clf = LinearSVC(random_state=0, tol=1e-5)
clf.fit(train_data, train_target)
test_pred = clf.predict(test_data)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
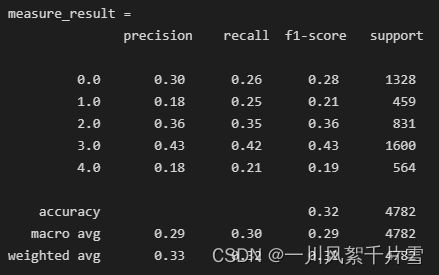
3.2.3 KNN
# KNN分类
from sklearn.neighbors import KNeighborsClassifier
# clf = KNeighborsClassifier(n_neighbors=10)
clf = KNeighborsClassifier()
clf.fit(train_data, train_target)
test_pred = clf.predict(test_data)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
3.2.4 决策树
# 决策树分类
from sklearn.tree import DecisionTreeClassifier
clf = DecisionTreeClassifier()
clf.fit(train_data, train_target)
test_pred = clf.predict(test_data)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
3.2.5 XGB
# xgb
params = {
'objective': 'multi:softmax', 'num_class' : 5
}
dtrain = xgb.DMatrix(train_data, train_target)
num_rounds = 500
model_xgb = xgb.train(params, dtrain, num_rounds)
dtest = xgb.DMatrix(test_data)
test_pred = model_xgb.predict(dtest)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
xgb_feature_importance = pd.DataFrame(list(model_xgb.get_fscore().items()))
xgb_feature_importance.columns = ['Feature', 'Feature importrance']
#设置绘图风格
plt.style.use('ggplot')
#处理中文乱码
#对读入的数据做降序排序
xgb_feature_importance.sort_values(by='Feature importrance', inplace=True, ascending=False)
#取前15行的数据
xgb_feature_importance = xgb_feature_importance.iloc[:15, :] # 取前15行数据
#对读入的数据做升序排序
xgb_feature_importance.sort_values(by='Feature importrance', inplace=True, ascending=True)
#绘制条形图
plt.barh(y = range(xgb_feature_importance.shape[0]), #指定条形图y轴的刻度值
width = xgb_feature_importance['Feature importrance'], #指定条形图x轴的数值
tick_label = xgb_feature_importance['Feature'], #指定条形图y轴的刻度标签
color = 'lightblue', #指定条形图的填充色
)
#添加x轴的标签
plt.xlabel('Feature importrance')
#添加条形图的标题
plt.title('XGB Feature importrance')
#为每个条形图添加数值标签
for y,x in enumerate(xgb_feature_importance['Feature importrance']):
plt.text(x+0.1,y,"%s"%round(x,1),va='center') #round(y,1)是将y值四舍五入到一个小数位
#显示图形
plt.show()
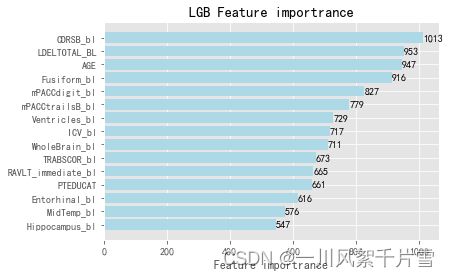
3.2.6 LGB
# lgb
from numpy import true_divide
params = {
'objective': 'multiclass', 'num_class' : 5
}
dtrain = lgb.Dataset(train_data, label=train_target)
cate_features_name = ['PTGENDER','PTETHCAT','PTRACCAT','PTMARRY']
model_lgb = lgb.train(params, dtrain, categorical_feature= cate_features_name)
test_pred = model_lgb.predict(test_data)
preds = test_pred
test_pred = []
for x in preds:
test_pred.append(np.argmax(x))
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
feature_name = pd.DataFrame(model_lgb.feature_name())
feature_importance = pd.DataFrame(model_lgb.feature_importance())
lgb_model_importance = pd.concat([feature_name, feature_importance], axis=1)
lgb_model_importance.columns = ['Feature', 'Feature importrance']
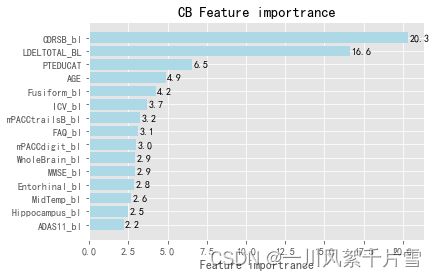
3.2.7 CB
# 因为catboost的类别变量得是整形或者字符型
# 报错信息:cat_features must be integer or string, real number values and NaN values should be converted to str
train_data = train.drop(['DX_bl','PTID'],axis=1)
train_target = train['DX_bl']
test_data = test.drop(['DX_bl','PTID'],axis=1)
test_target = test['DX_bl']
train_data[['PTGENDER','PTETHCAT','PTRACCAT','PTMARRY']] = train_data[['PTGENDER','PTETHCAT','PTRACCAT','PTMARRY']].astype(str)
test_data[['PTGENDER','PTETHCAT','PTRACCAT','PTMARRY']] = test_data[['PTGENDER','PTETHCAT','PTRACCAT','PTMARRY']].astype(str)
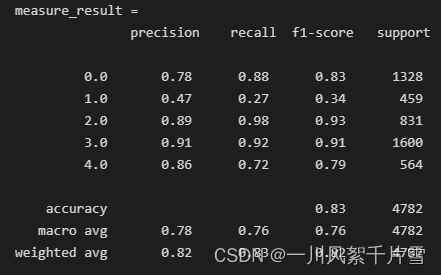
# cb
cat_features_index = [1,3,4,5]
model_cb = cb.CatBoostClassifier()
model_cb.fit(train_data, train_target, cat_features=cat_features_index)
test_pred = model_cb.predict(test_data)
measure_result = classification_report(test_target, test_pred)
print('measure_result = \n', measure_result)
fea_ = model_cb.feature_importances_
fea_name = model_cb.feature_names_
cb_value = pd.DataFrame(fea_)
cb_name = pd.DataFrame(fea_name)
cb_feature_importrance = pd.concat([cb_name,cb_value], axis=1)
cb_feature_importrance.columns = ['Feature', 'Feature importrance']
3.3 各模型的对比
关于模型评价指标,可以详见下面这篇文章
【数学建模】分类问题的几种常见指标(一)——准确率、召回率、F1值
我们将7个模型的accuracy、macro avg和weighted avg中的f1-score进行了可视化分析,如下图所示。
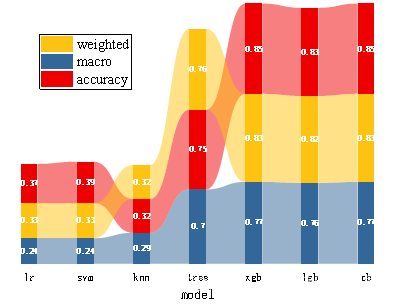 可以看到,基于集成学习的模型均取得了较为令人满意的成绩,accuracy均达到了83%以上。但是在类别1中的分类效果仍然不理想。这可能是模型忽略了SMC这种类别的特征。在现实中,可能该种症状也是难以发现的,由此可见,对阿尔兹海默症的早期预防仍然存在着巨大的挑战。
可以看到,基于集成学习的模型均取得了较为令人满意的成绩,accuracy均达到了83%以上。但是在类别1中的分类效果仍然不理想。这可能是模型忽略了SMC这种类别的特征。在现实中,可能该种症状也是难以发现的,由此可见,对阿尔兹海默症的早期预防仍然存在着巨大的挑战。
4 问题二求解
注意,本问题省略了一些中间步骤
4.1 数据处理
选取上文XGB、LGB、CB中最重要的15个特征的并集+‘PTID’作为新的数据集
保存新的数据集命名为:df_cluster_18F.csv
# 选中其中的18个特征+1个label
df_cluster = df[['AGE','mPACCtrailsB_bl','ICV_bl','LDELTOTAL_BL','WholeBrain_bl',
'CDRSB_bl','MidTemp_bl','TRABSCOR_bl','Entorhinal_bl','mPACCdigit_bl','Fusiform_bl','Ventricles_bl','Hippocampus_bl',
'PTEDUCAT','RAVLT_immediate_bl','FAQ_bl','MMSE_bl','ADAS11_bl','DX_bl']]
df_cluster.to_csv('./data/df_cluster_18F.csv', index=False)
将数据集中的5类标签,保存为3类。
即CN——CN,SMC、EMCI、LMCI——MCI,AD——AD
# 将5类标签转换为3类标签
dict = {
0:0, 1:1, 2:1, 3:1, 4:2
}
df_cluster['DX_bl'] = df_cluster.DX_bl.map(dict)
df_cluster['DX_bl'].value_counts()
df_cluster.to_csv('./data/df_3cluster_18F.csv', index=False)
4.2 数据探索
按照上文方法读取df_3cluster_18F.csv数据,按照下列方式进行划分数据
# PTID小于1687的70%的病人划分到训练集
train = df[df['PTID'] < 1687]
test = df[df['PTID'] >= 1687]
train_data = train.drop(['DX_bl','PTID'],axis=1)
train_target = train['DX_bl']
test_data = test.drop(['DX_bl','PTID'],axis=1)
test_target = test['DX_bl']
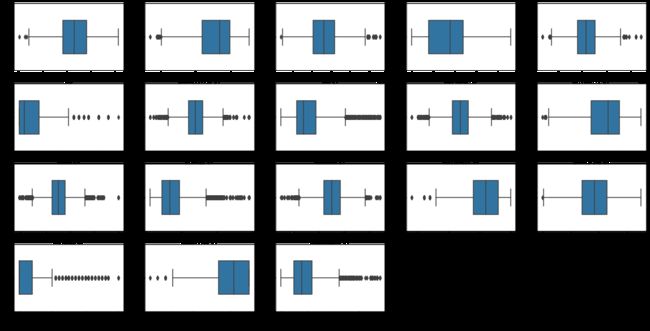
绘制这些变量的箱线图来查看异常值
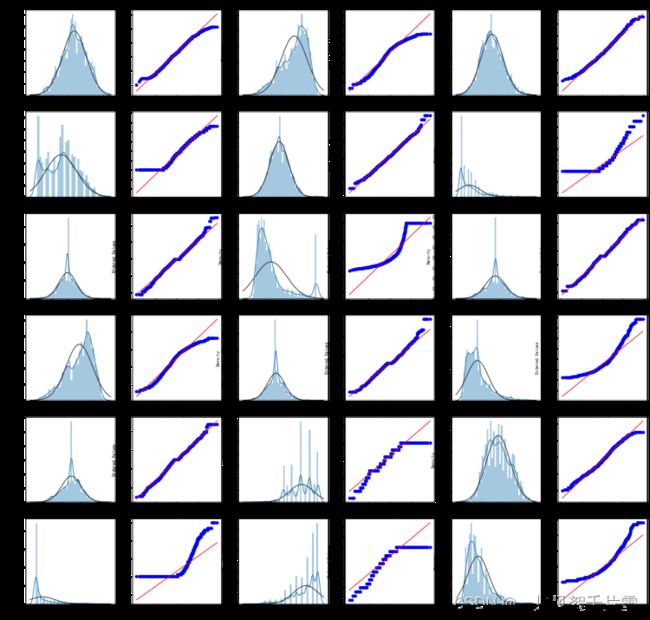
绘制直方图与Q-Q图,来检验这些特征是否符合正态分布,这和后边相关性分析选择何种参数有关。
查看训练集和测试集的分布,如果分布存在不一致的情况,需要对特征进行删除

4.3 相关性分析
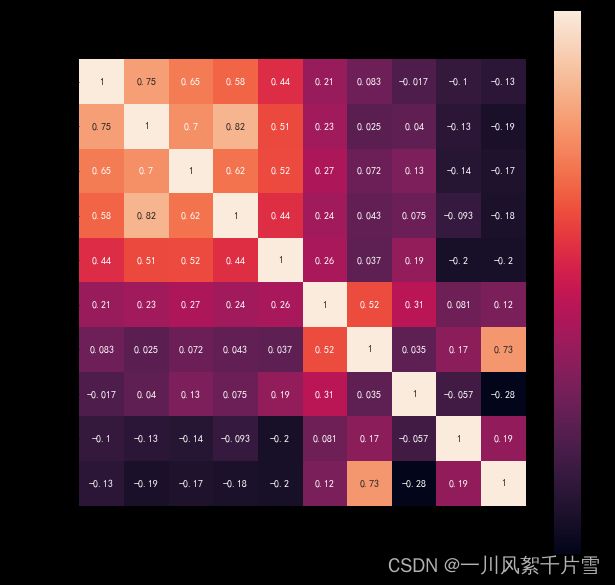
绘制相关性热力图
data_train1 = train.drop(['PTID'],axis=1)
train_corr = data_train1.corr()
train_corr
# 画出相关性热力图
ax = plt.subplots(figsize=(20, 16))#调整画布大小
ax = sns.heatmap(train_corr, vmax=.8, square=True, annot=True)#画热力图 annot=True 显示系数
#寻找K个最相关的特征信息
k = 10 # number of variables for heatmap
cols = train_corr.nlargest(k, 'DX_bl')['DX_bl'].index
cm = np.corrcoef(data_train1[cols].values.T)
hm = plt.subplots(figsize=(10, 10))#调整画布大小
#hm = sns.heatmap(cm, cbar=True, annot=True, square=True)
#g = sns.heatmap(train_data[cols].corr(),annot=True,square=True,cmap="RdYlGn")
hm = sns.heatmap(data_train1[cols].corr(),annot=True,square=True)
plt.show()
同理,绘制相关性大于0.5的特征信息
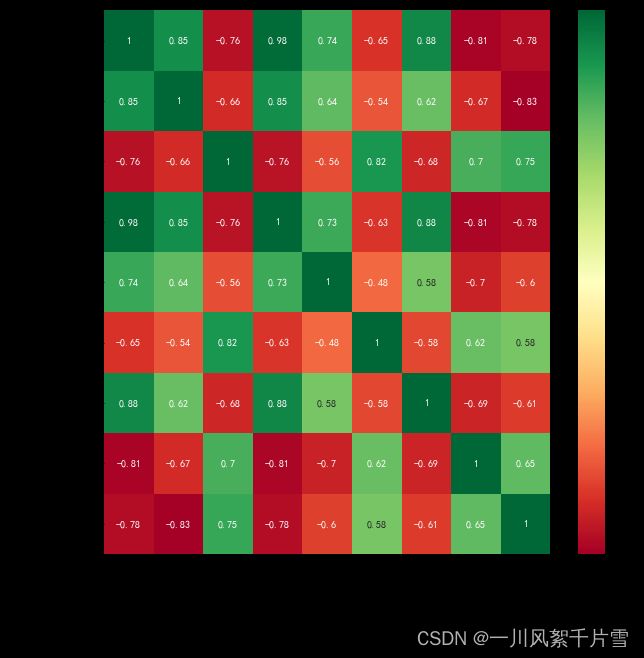
根据上述信息,可以对影响阿尔兹海默症的相关因素进行分析
5 问题三求解
该问题为聚类问题,首先读取df_3cluster_18F.csv数据集
5.1 K值的选择
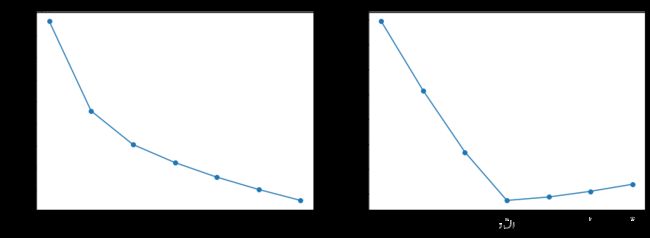
绘制手肘图
data = np.array(train_data)
Scores = [] # 存放轮廓系数
SSE = [] # 存放每次结果的误差平方和
for k in range(2, 9):
estimator = KMeans(n_clusters=k) # 构造聚类器
estimator.fit(data)
Scores.append(silhouette_score(
np.array(df), estimator.labels_, metric='euclidean'))
SSE.append(estimator.inertia_) # estimator.inertia_获取聚类准则的总和
X = range(2, 9)
plt.figure(figsize=(15,5))
plt.subplot(121)
plt.xlabel('k', fontsize=15)
plt.ylabel('SSE', fontsize=15)
plt.plot(X, SSE, 'o-')
plt.subplot(122)
plt.xlabel('k值', fontsize=15)
plt.ylabel('轮廓系数', fontsize=15)
plt.plot(X, Scores, 'o-')
plt.savefig('./img/手肘法.png',dpi=300)
plt.show()
5.2 随机种子选择
我也没有理解这个题目为什么要先聚成3类,这里选择k值为3,随机种子取值范围设定为2000-2025,绘制下图
Scores = [] # 存放轮廓系数
for i in range(2000,2025):
estimator = KMeans(n_clusters=3, random_state=i) # 构造聚类器
estimator.fit(data)
Scores.append(silhouette_score(np.array(df), estimator.labels_, metric='euclidean'))
X = range(2000, 2025)
plt.figure(figsize=(7,5))
plt.xlabel('random_state', fontsize=15)
plt.ylabel('silhouette coefficient', fontsize=15)
plt.plot(X, Scores, 'o-')
plt.xlim(2000, 2025)
plt.savefig('./img/随机种子的确定.png',dpi=300)
plt.show()
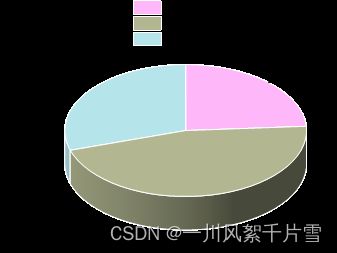
5.3 Kmeans聚类
然后采用k=3,random_state=2009,进行kmeans聚类(其它的聚类方法也可以尝试)
# Kmens 聚类
from sklearn.cluster import KMeans
# 实例化K-Means算法模型,先使用3个簇尝试聚类
cluster = KMeans(n_clusters=3, random_state=2009)
# 使用数据集train_data进行训练
cluster = cluster.fit(train_data)
# 调用属性labels_,查看聚类结果
cluster.labels_
labels_pred = cluster.labels_
import collections
data_count2=collections.Counter(labels_pred)
data_count2
聚类结果如下:
计算此时的ARI指数为:0.012940512409904181
采用PCA对数据进行降维,采用相同方式进行聚类,可视化结果如下
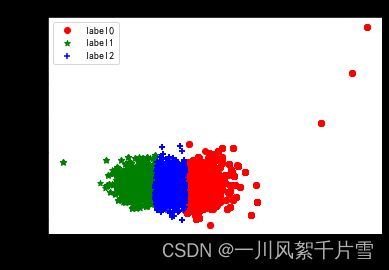
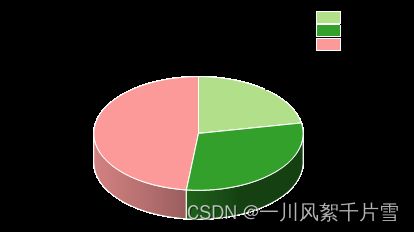
第二小问的结果同上,直接展示结果:

6 问题四、问题五求解
6.1 问题四
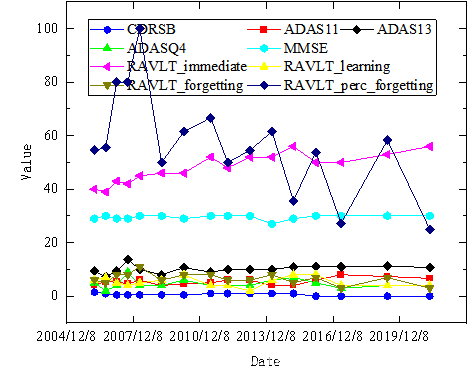
该问题我采用了可视化的方法进行了分析,可以先绘制出各特征随时间的变化情况,然后对图片上的趋势自己进行描述。
分别选取了4种类型病人进行分析,其中SMC的PTID为135_S_5113,EMCI的PTID为007_S_2394,LMCI的PTID为021_S_0178,AD的PTID为027_S_4938。在这里,选取了9个特征分别为:CDRSB、ADAS11、ADAS13、ADASQ4、MMSE、RAVLT_immediate、RAVLT_learning、RAVLT_forgetting和RAVLT_perc_forgetting进行分析,分别对4个研究样例的特征变化进行可视化,如下图所示。



至于问题五,就去查资料吧
小结
仅提供一个解题思路,存在一些不合理的地方,具体方案还得各位自己动手。
如有疑问,可咨询企鹅:1244816050
附件
待整理~