python数据分析绘图
ROC-AUC曲线(分类模型)
混淆矩阵
- True negative(TN),称为真阴率,表明实际是负样本预测成负样本的样本数(预测是负样本,预测对了)
- False positive(FP),称为假阳率,表明实际是负样本预测成正样本的样本数(预测是正样本,预测错了)
- False negative(FN),称为假阴率,表明实际是正样本预测成负样本的样本数(预测是负样本,预测错了)
- True positive(TP),称为真阳率,表明实际是正样本预测成正样本的样本数(预测是正样本,预测对了)
ROC曲线示例

可以看到,ROC曲线的纵坐标为真阳率true positive rate(TPR)(也就是recall),横坐标为假阳率false positive rate(FPR)。
TPR即真实正例中对的比例,FPR即真实负例中的错的比例。
- 真正类率(True Postive Rate)TPR:
TPR=TP/(TP+FN)
代表分类器 预测为正类中实际为正实例占所有正实例 的比例。 - 假正类率(False Postive Rate)FPR:
FPR=FP/(FP+TN)
代表分类器 预测为正类中实际为负实例 占 所有负实例 的比例。

可以看到,右上角的阈值最小,对应坐标点(1,1);左下角阈值最大,对应坐标点为(0,0)。从右上角到左下角,随着阈值的逐渐减小,越来越多的实例被划分为正类,但是这些正类中同样也掺杂着真正的负实例,即TPR和FPR会同时增大。
- 横轴FPR: FPR越大,预测正类中实际负类越多。
- 纵轴TPR:TPR越大,预测正类中实际正类越多。
- 理想目标:TPR=1,FPR=0,即图中(0,1)点,此时ROC曲线越靠拢(0,1)点,越偏离45度对角线越好。
AUC值是什么?
AUC(Area Under Curve)被定义为ROC曲线下与坐标轴围成的面积,显然这个面积的数值不会大于1。又由于ROC曲线一般都处于y=x这条直线的上方,所以AUC的取值范围在0.5和1之间。
ROC曲线绘制的代码实现
#导入库
from sklearn.metrics import confusion_matrix,accuracy_score,f1_score,roc_auc_score,recall_score,precision_score,roc_curve
import matplotlib.pyplot as plt
from sklearn.metrics import roc_curve, auc
import matplotlib.pyplot as plt
#绘制roc曲线
def calculate_auc(y_test, pred):
print("auc:",roc_auc_score(y_test, pred))
fpr, tpr, thersholds = roc_curve(y_test, pred)
roc_auc = auc(fpr, tpr)
plt.plot(fpr, tpr, 'k-', label='ROC (area = {0:.2f})'.format(roc_auc),color='blue', lw=2)
plt.xlim([-0.05, 1.05])
plt.ylim([-0.05, 1.05])
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title('ROC Curve')
plt.legend(loc="lower right")
plt.plot([0, 1], [0, 1], 'k--')
plt.show()
相关性热图
表示数据之间的相互依赖关系。但需要注意,数据具有相关性不一定意味着具有因果关系。
相关系数(Pearson)
相关系数是研究变量之间线性相关程度的指标,而相关关系是一种非确定性的关系,数据具有相关性不能推出有因果关系。相关系数的计算公式如下:

其中,公式的分子为X,Y两个变量的协方差,Var(X)和Var(Y)分别是这两个变量的方差。当X,Y的相关程度最高时,即X,Y趋近相同时,很容易发现分子和分母相同,即r=1。
代码实现
相关性计算
import numpy as np
import pandas as pd
# compute correlations
from scipy.stats import spearmanr, pearsonr
from scipy.spatial.distance import cdist
def calc_spearman(df1, df2):
df1 = pd.DataFrame(df1)
df2 = pd.DataFrame(df2)
n1 = df1.shape[1]
n2 = df2.shape[1]
corr0, pval0 = spearmanr(df1.values, df2.values)
# (n1 + n2) x (n1 + n2)
corr = pd.DataFrame(corr0[:n1, -n2:], index=df1.columns, columns=df2.columns)
pval = pd.DataFrame(pval0[:n1, -n2:], index=df1.columns, columns=df2.columns)
return corr, pval
def calc_pearson(df1, df2):
df1 = pd.DataFrame(df1)
df2 = pd.DataFrame(df2)
n1 = df1.shape[1]
n2 = df2.shape[1]
corr0, pval0 = np.zeros((n1, n2)), np.zeros((n1, n2))
for row in range(n1):
for col in range(n2):
_corr, _p = pearsonr(df1.values[:, row], df2.values[:, col])
corr0[row, col] = _corr
pval0[row, col] = _p
# n1 x n2
corr = pd.DataFrame(corr0, index=df1.columns, columns=df2.columns)
pval = pd.DataFrame(pval0, index=df1.columns, columns=df2.columns)
return corr, pval
画出相关性图
import matplotlib.pyplot as plt
import seaborn as sns
def pvalue_marker(pval, corr=None, only_pos=False):
if only_pos: # 只标记正相关
if corr is None:
print('correlations `corr` is not provided, '
'negative correlations cannot be filtered!')
else:
pval = pval + (corr < 0).astype(float)
pval_marker = pval.applymap(lambda x: '**' if x < 0.01 else ('*' if x < 0.05 else ''))
return pval_marker
def plot_heatmap(
mat, cmap='RdBu_r',
xlabel=f'column', ylabel=f'row',
tt='',
fp=None,
**kwds
):
fig, ax = plt.subplots()
sns.heatmap(mat, ax=ax, cmap=cmap, cbar_kws={'shrink': 0.5}, **kwds)
ax.set_title(tt)
ax.set_xlabel(xlabel)
ax.set_ylabel(ylabel)
if fp is not None:
ax.figure.savefig(fp, bbox_inches='tight')
return ax
实例
#构造有一定相关性的随机矩阵
df1 = pd.DataFrame(np.random.randn(40, 9))
df2 = df1.iloc[:, :-1] + df1.iloc[:, 1: ].values * 0.6
df2 += 0.2 * np.random.randn(*df2.shape)
#绘图
corr, pval = calc_pearson(df1, df2)
pval_marker = pvalue_marker(pval, corr, only_pos=only_pos)
tt = 'Spearman correlations'
plot_heatmap(
corr, xlabel='df2', ylabel='df1',
tt=tt, cmap='RdBu_r', #vmax=0.75, vmin=-0.1,
annot=pval_marker, fmt='s',
)

only_pos 这个参数为 False 时, 会同时标记显著的正相关和负相关.
cmap属性调整颜色可选参数:
‘Accent’, ‘Accent_r’, ‘Blues’, ‘Blues_r’, ‘BrBG’, ‘BrBG_r’, ‘BuGn’, ‘BuGn_r’, ‘BuPu’, ‘BuPu_r’, ‘CMRmap’,‘CMRmap_r’, ‘Dark2’, ‘Dark2_r’, ‘GnBu’, ‘GnBu_r’, ‘Greens’, ‘Greens_r’, ‘Greys’, ‘Greys_r’, ‘OrRd’, ‘OrRd_r’, ‘Oranges’, ‘Oranges_r’, ‘PRGn’, ‘PRGn_r’, ‘Paired’, ‘Paired_r’, ‘Pastel1’, ‘Pastel1_r’, ‘Pastel2’, ‘Pastel2_r’, ‘PiYG’, ‘PiYG_r’, ‘PuBu’, ‘PuBuGn’, ‘PuBuGn_r’, ‘PuBu_r’, ‘PuOr’, ‘PuOr_r’, ‘PuRd’, ‘PuRd_r’, ‘Purples’, ‘Purples_r’, ‘RdBu’, ‘RdBu_r’, ‘RdGy’, ‘RdGy_r’, ‘RdPu’, ‘RdPu_r’, ‘RdYlBu’, ‘RdYlBu_r’, ‘RdYlGn’, ‘RdYlGn_r’, ‘Reds’, ‘Reds_r’, ‘Set1’, ‘Set1_r’, ‘Set2’, ‘Set2_r’, ‘Set3’, ‘Set3_r’, ‘Spectral’, ‘Spectral_r’, ‘Wistia’, ‘Wistia_r’, ‘YlGn’, ‘YlGnBu’, ‘YlGnBu_r’, ‘YlGn_r’, ‘YlOrBr’, ‘YlOrBr_r’, ‘YlOrRd’, ‘YlOrRd_r’, ‘afmhot’, ‘afmhot_r’, ‘autumn’, ‘autumn_r’, ‘binary’, ‘binary_r’,‘bone’, ‘bone_r’, ‘brg’, ‘brg_r’, ‘bwr’, ‘bwr_r’, ‘cividis’, ‘cividis_r’, ‘cool’, ‘cool_r’, ‘coolwarm’, ‘coolwarm_r’, ‘copper’, ‘copper_r’, ‘crest’, ‘crest_r’, ‘cubehelix’, ‘cubehelix_r’, ‘flag’, ‘flag_r’, ‘flare’, ‘flare_r’, ‘gist_earth’, ‘gist_earth_r’, ‘gist_gray’, ‘gist_gray_r’, ‘gist_heat’, ‘gist_heat_r’, ‘gist_ncar’, ‘gist_ncar_r’, ‘gist_rainbow’, ‘gist_rainbow_r’, ‘gist_stern’, ‘gist_stern_r’, ‘gist_yarg’, ‘gist_yarg_r’, ‘gnuplot’, ‘gnuplot2’, ‘gnuplot2_r’, ‘gnuplot_r’, ‘gray’, ‘gray_r’, ‘hot’, ‘hot_r’, ‘hsv’, ‘hsv_r’,‘plasma’, ‘plasma_r’, ‘prism’, ‘prism_r’, ‘rainbow’, ‘rainbow_r’, ‘rocket’, ‘rocket_r’, ‘seismic’, ‘seismic_r’, ‘spring’, ‘spring_r’, ‘summer’, ‘summer_r’, ‘tab10’, ‘tab10_r’, ‘tab20’, ‘tab20_r’, ‘tab20b’, ‘tab20b_r’, ‘tab20c’, ‘tab20c_r’, ‘terrain’, ‘terrain_r’, ‘turbo’, ‘turbo_r’, ‘twilight’, ‘twilight_r’, ‘twilight_shifted’, ‘twilight_shifted_r’, ‘viridis’, ‘viridis_r’, ‘vlag’, ‘vlag_r’, ‘winter’, ‘winter_r’
棒棒糖图
条形图在数据可视化里,是一个经常被使用到的图表。虽然很好用,也还是存在着缺陷呢。比如条形图条目太多时,会显得臃肿,不够直观。
棒棒糖图表则是对条形图的改进,以一种小清新的设计,清晰明了表达了我们的数据。
代码实现
# 导包
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
# 创建数据
x=range(1,41)
values=np.random.uniform(size=40)
# 绘制
plt.stem(x, values)
plt.ylim(0, 1.2)
plt.show()
# stem function: If x is not provided, a sequence of numbers is created by python:
plt.stem(values)
plt.show()
# Create a dataframe
df = pd.DataFrame({'group':list(map(chr, range(65, 85))), 'values':np.random.uniform(size=20) })
# Reorder it based on the values:
ordered_df = df.sort_values(by='values')
my_range=range(1,len(df.index)+1)
ordered_df.head()
# Make the plot
plt.stem(ordered_df['values'])
plt.xticks( my_range, ordered_df['group'])
plt.show()
# Horizontal version
plt.hlines(y=my_range, xmin=0, xmax=ordered_df['values'], color='skyblue')
plt.plot(ordered_df['values'], my_range, "D")
plt.yticks(my_range, ordered_df['group'])
plt.show()
# change color and shape and size and edges
(markers, stemlines, baseline) = plt.stem(values)
plt.setp(markers, marker='D', markersize=10, markeredgecolor="orange", markeredgewidth=2)
plt.show()
# custom the stem lines
(markers, stemlines, baseline) = plt.stem(values)
plt.setp(stemlines, linestyle="-", color="olive", linewidth=0.5 )
plt.show()
# Create a dataframe
value1=np.random.uniform(size=20)
value2=value1+np.random.uniform(size=20)/4
df = pd.DataFrame({'group':list(map(chr, range(65, 85))), 'value1':value1 , 'value2':value2 })
# Reorder it following the values of the first value:
ordered_df = df.sort_values(by='value1')
my_range=range(1,len(df.index)+1)
# The horizontal plot is made using the hline function
plt.hlines(y=my_range, xmin=ordered_df['value1'], xmax=ordered_df['value2'], color='grey', alpha=0.4)
plt.scatter(ordered_df['value1'], my_range, color='skyblue', alpha=1, label='value1')
plt.scatter(ordered_df['value2'], my_range, color='green', alpha=0.4 , label='value2')
plt.legend()
# Add title and axis names
plt.yticks(my_range, ordered_df['group'])
plt.title("Comparison of the value 1 and the value 2", loc='left')
plt.xlabel('Value of the variables')
plt.ylabel('Group')
# Show the graph
plt.show()
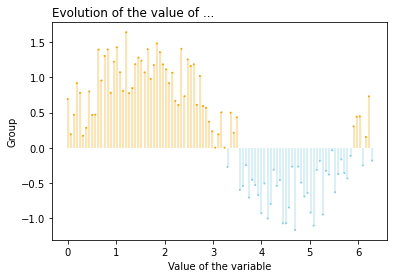
# Data
x = np.linspace(0, 2*np.pi, 100)
y = np.sin(x) + np.random.uniform(size=len(x)) - 0.2
# Create a color if the y axis value is equal or greater than 0
my_color = np.where(y>=0, 'orange', 'skyblue')
# The vertical plot is made using the vline function
plt.vlines(x=x, ymin=0, ymax=y, color=my_color, alpha=0.4)
plt.scatter(x, y, color=my_color, s=1, alpha=1)
# Add title and axis names
plt.title("Evolution of the value of ...", loc='left')
plt.xlabel('Value of the variable')
plt.ylabel('Group')
# Show the graph
plt.show()
火山图
火山图(Volcano plots)是散点图的一种,根据变化幅度(FC,Fold Change)和变化幅度的显著性(P value)进行绘制,其中标准化后的FC值作为横坐标,P值作为纵坐标,可直观的反应高变的数据点,常用于基因组学分析(转录组学、代谢组学等)。
绘制
制作差异分析结果数据框
genearray = np.asarray(pvalue)
result = pd.DataFrame({'pvalue':genearray,'FoldChange':fold})
result['log(pvalue)'] = -np.log10(result['pvalue'])
制作火山图的准备工作
result['sig'] = 'normal'
result['size'] =np.abs(result['FoldChange'])/10
result.loc[(result.FoldChange> 1 )&(result.pvalue < 0.05),'sig'] = 'up'
result.loc[(result.FoldChange< -1 )&(result.pvalue < 0.05),'sig'] = 'down'
ax = sns.scatterplot(x="FoldChange", y="log(pvalue)",
hue='sig',
hue_order = ('down','normal','up'),
palette=("#377EB8","grey","#E41A1C"),
data=result)
ax.set_ylabel('-log(pvalue)',fontweight='bold')
ax.set_xlabel('FoldChange',fontweight='bold')