基于聚类的离群点检测(sklearn实现)
目录
- 前言
- 一、对Iris数据集应用kmeans聚类方法进行离群点检测,并分别采用tsne、MDS、Isomap和PCA降维将原数据降到2维并在新数据中标出离群点
-
- 1.1 数据准备
- 1.2 离群点检测
- 1.3 在降维后的数据上显示离群点
- 二、使用Kmeans聚类、DBCAN聚类和BIRCH聚类方法分别对去除离群点前后的数据集进行聚类,最后通过比较他们的NMI值确定聚类效果的好坏
-
- 2.1 设置Kmeans聚类、DBCAN聚类和BIRCH聚类的参数
- 2.2 对原始数据进行三种聚类并计算其NMI
- 2.3 得到去除离群点之后的新数据
- 2.4 对新数据进行三种聚类并计算其NMI值
- 2.5 统计不同聚类方法在去除离群点前后的NMI值
- 三、对Iris数据集先分别采用tsne、MDS、Isomap和PCA降维,然后对降维后的数据进行离群点的检测。
-
- 3.1 定义kmean检测离群点函数
- 3.2 对进行tsne、MDS、Isomap和PCA降维后的数据进行离群点检测
- 总结
前言
离群点的定义:离群点是一个数据对象,它显著不同于其他数据对象,好像它是被不同的机制产生一样。
生活中的数据往往会受到各种因素的影响而呈现异常的状态,为了对数据进行分析和处理,进行离群点检测便变得十分重要。
本文主要包括以下三个方面:
- 对Iris数据集应用kmeans聚类方法进行离群点检测,并分别采用tsne、MDS、Isomap和PCA降维将原数据降到2维并在新数据中标出离群点。
- 使用Kmeans聚类、DBCAN聚类和BIRCH聚类方法分别对去除离群点前后的数据集进行聚类,最后通过比较他们的NMI值确定聚类效果的好坏
- 对Iris数据集先分别采用sne、MDS、Isomap和PCA降维,然后对降维后的数据进行离群点的检测。
一、对Iris数据集应用kmeans聚类方法进行离群点检测,并分别采用tsne、MDS、Isomap和PCA降维将原数据降到2维并在新数据中标出离群点
1.1 数据准备
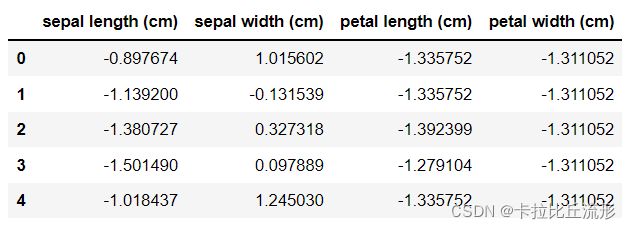
将Iris数据集转化为数据框,以便后续的处理。
import warnings
warnings.filterwarnings("ignore")
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from sklearn import datasets #数据集包
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
threshold = 2 #离群点的阈值
Iris_df = datasets.load_iris()
df = pd.DataFrame(Iris_df["data"],columns=Iris_df.feature_names)#数据
df1 = pd.DataFrame(Iris_df["target"],columns = ["target"]) #真实数据标签
#df = pd.concat([df1, df2], axis = 1)
data = 1.0*(df - df.mean())/df.std()#对数据进行标准化
data.head()#显示部分数据
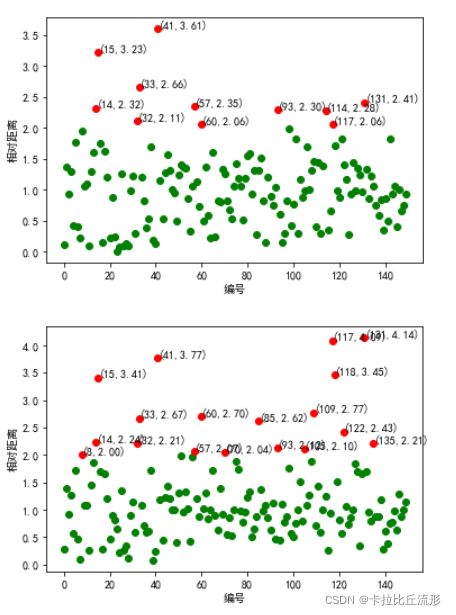
1.2 离群点检测
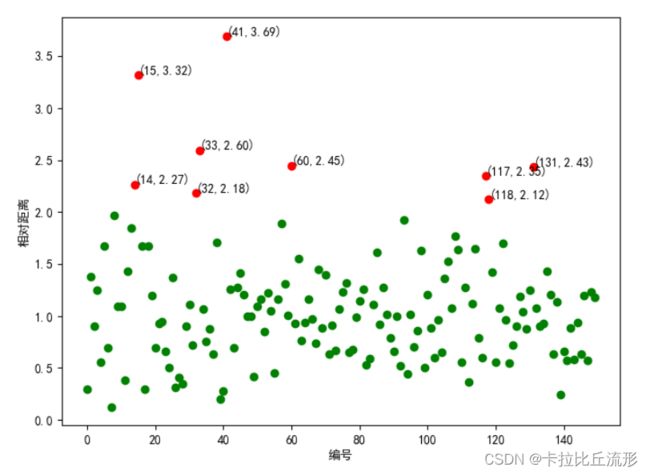
对原始的Iris数据集进行基于聚类的离群点检测,并标出离群点。
k = 3 #聚类类别
iteration = 500 #聚类最大循环次数
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=3, n_jobs=4,max_iter=500, random_state=1314)
kmeans.fit(data)#训练kmeans模型
y_pred0 = kmeans.predict(data)
r = pd.concat([data, pd.Series(kmeans.labels_, index = data.index)], axis = 1)
r.columns = list(data.columns) + ["聚类类别"] #重置新的表头
y_pre1 = r['聚类类别']
norm = []
for i in range(k): #逐一处理
norm_tmp = r[data.columns][r["聚类类别"]==i] - kmeans.cluster_centers_[i]
norm_tmp = norm_tmp.apply(np.linalg.norm, axis = 1)#求出绝对距离
norm.append(norm_tmp/norm_tmp.median())#求出相对距离并添加
norm = pd.concat(norm) #将每个点与各自聚类中心点的距离
ax = plt.figure(figsize=(8,6),dpi=100)
norm[norm<=threshold].plot(style="go")#小于阈值即为正常点并绘制图像
discreste_points = norm[norm > threshold]#大于阈值即为离群点
discreste_points.plot(style="ro")#绘制离群点
for i in range(len(discreste_points)):#为离群点添加标签
id = discreste_points.index[i] #离群点的索引编号
n = discreste_points.iloc[i] #离群点与各自聚类中心的相对距离
plt.annotate("(%s,%0.2f)"%(id,n),xy=(id,n),xytext=(id,n))
plt.xlabel("编号")
plt.ylabel("相对距离")
1.3 在降维后的数据上显示离群点
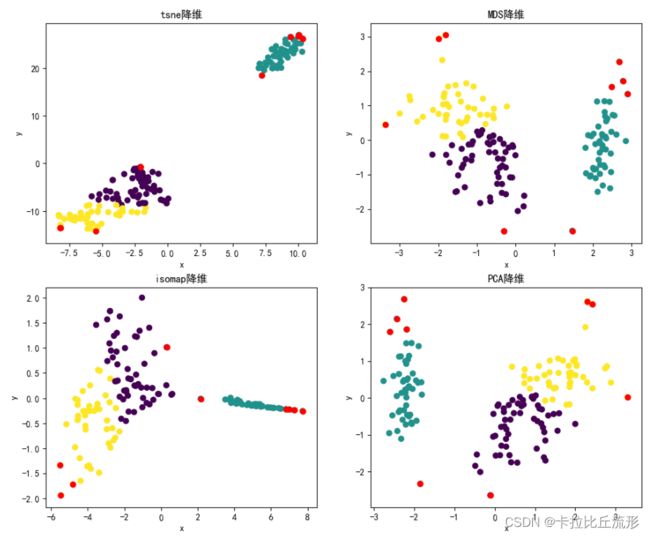
分别采用tsne、MDS、Isomap和PCA降维将原数据降到2维并在新数据中标出离群点
plt.figure(figsize=(12,10),dpi=100)
from sklearn.manifold import TSNE
tsne = TSNE(n_components=2, random_state=105)
tsne_result = tsne.fit_transform(data)#降维后的数据
plt.subplot(2,2,1)
plt.scatter(tsne_result[:,0],tsne_result[:,1],c=r["聚类类别"])
plt.scatter(tsne_result[discreste_points.index.values,0],tsne_result[discreste_points.index.values,1],c="red")
plt.title("tsne降维")
plt.xlabel("x")
plt.ylabel("y")
from sklearn.manifold import MDS
mds = MDS(n_components=2)
mds_result = mds.fit_transform(data)
plt.subplot(2,2,2)
plt.scatter(mds_result[:,0],mds_result[:,1],c=r["聚类类别"])
plt.scatter(mds_result[discreste_points.index.values,0],mds_result[discreste_points.index.values,1],c="red")
plt.title("MDS降维")
plt.xlabel("x")
plt.ylabel("y")
from sklearn.manifold import Isomap
isomap = Isomap(n_components=2)
isomap_result = isomap.fit_transform(data)
plt.subplot(2,2,3)
plt.scatter(isomap_result[:,0],isomap_result[:,1],c=r["聚类类别"])
plt.scatter(isomap_result[discreste_points.index.values,0],isomap_result[discreste_points.index.values,1],c="red")
plt.title("isomap降维")
plt.xlabel("x")
plt.ylabel("y")
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca_result = pca.fit_transform(data)
plt.subplot(2,2,4)
plt.scatter(pca_result[:,0],pca_result[:,1],c=r["聚类类别"])
plt.scatter(pca_result[discreste_points.index.values,0],pca_result[discreste_points.index.values,1],c="red")
plt.title("PCA降维")
plt.xlabel("x")
plt.ylabel("y")
二、使用Kmeans聚类、DBCAN聚类和BIRCH聚类方法分别对去除离群点前后的数据集进行聚类,最后通过比较他们的NMI值确定聚类效果的好坏
2.1 设置Kmeans聚类、DBCAN聚类和BIRCH聚类的参数
from sklearn import metrics
from sklearn.cluster import DBSCAN
from sklearn.cluster import KMeans
from sklearn.cluster import Birch
y_true = df1["target"]#真实标签
kmeans = KMeans(n_clusters=3, n_jobs=4,max_iter=500, random_state=1314)
dbcan = DBSCAN(eps=0.6, min_samples=9)
birch = Birch(n_clusters=None)
2.2 对原始数据进行三种聚类并计算其NMI
y_pred11 = kmeans.fit_predict(data)#kmeans预测的标签
nmi11 = metrics.normalized_mutual_info_score(y_true, y_pred11)
y_pred12 = dbcan.fit_predict(data)#dbcan预测的标签
nmi12 = metrics.normalized_mutual_info_score(y_true, y_pred12)
y_pred13 = birch.fit_predict(data)#birch预测的标签
nmi13 = metrics.normalized_mutual_info_score(y_true, y_pred13)
2.3 得到去除离群点之后的新数据
#去除离群点之后的数据
data1 = data
data_new = data.drop(index=discreste_points.index.values)
df2 = df1
df2 = df2.drop(index=discreste_points.index.values)
y_true = df2["target"]
2.4 对新数据进行三种聚类并计算其NMI值
y_pred21 = kmeans.fit_predict(data_new)#kmeans预测的标签
nmi21 = metrics.normalized_mutual_info_score(y_true, y_pred21)
y_pred22 = dbcan.fit_predict(data_new)#dbcan预测的标签
nmi22 = metrics.normalized_mutual_info_score(y_true, y_pred22)
y_pred23 = birch.fit_predict(data_new)#birch预测的标签
nmi23 = metrics.normalized_mutual_info_score(y_true, y_pred23)
2.5 统计不同聚类方法在去除离群点前后的NMI值
dists = {
"剔除离群点前的NMI":[nmi11,nmi12,nmi13],
"剔除离群点后的NMI":[nmi21,nmi22,nmi23],
}
result = pd.DataFrame(dists,index=["Kmeans聚类","DBSCAN聚类","BIRCH聚类"])
result
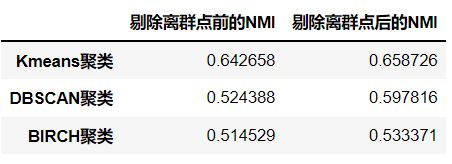
NMI值越大越好,从这里可以看出在剔除离群点前后Kmeans的聚类效果最好。
**注意:**这里的聚类效果好坏是相对的,我们还要综合其它的聚类指标来进行综合考量,而且对于不同模型设置的参数不同,最终得到的NMI值也不相同。
三、对Iris数据集先分别采用tsne、MDS、Isomap和PCA降维,然后对降维后的数据进行离群点的检测。
3.1 定义kmean检测离群点函数
def kmean_outlier_detection(data): #kmean检测离群点
#输入:数据框
k = 3 #聚类类别
iteration = 500 #聚类最大循环次数
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=3, n_jobs=4,max_iter=500, random_state=1)
kmeans.fit(data)#训练kmeans模型
y_pred0 = kmeans.predict(data)
r = pd.concat([data, pd.Series(kmeans.labels_, index = data.index)], axis = 1)
r.columns = list(data.columns) + ["聚类类别"] #重置新的表头
y_pre1 = r['聚类类别']
norm = []
for i in range(k): #逐一处理
norm_tmp = r[data.columns][r["聚类类别"]==i] - kmeans.cluster_centers_[i]
norm_tmp = norm_tmp.apply(np.linalg.norm, axis = 1)#求出绝对距离
norm.append(norm_tmp/norm_tmp.median())#求出相对距离并添加
norm = pd.concat(norm) #将每个点与各自聚类中心点的距离
ax = plt.figure()
norm[norm<=threshold].plot(style="go")#小于阈值即为正常点并绘制图像
discreste_points = norm[norm > threshold]#大于阈值即为离群点
discreste_points.plot(style="ro")#绘制离群点
for i in range(len(discreste_points)):#为离群点添加标签
id = discreste_points.index[i] #离群点的索引编号
n = discreste_points.iloc[i] #离群点与各自聚类中心的相对距离
plt.annotate("(%s,%0.2f)"%(id,n),xy=(id,n),xytext=(id,n))
plt.xlabel("编号")
plt.ylabel("相对距离")
return plt
3.2 对进行tsne、MDS、Isomap和PCA降维后的数据进行离群点检测
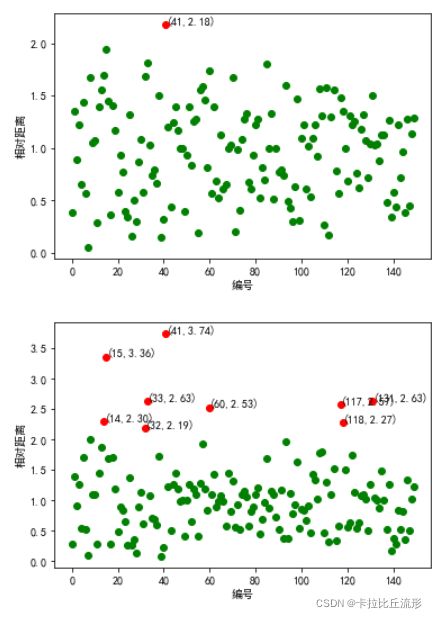
对进行tsne、MDS、Isomap和PCA降维后的数据分别调用kmean_outlier_detection进行离群点检测并绘制图形。
from sklearn.manifold import TSNE
tsne = TSNE(n_components=2, random_state=105)
tsne_result = tsne.fit_transform(data)#降维后的数据
tsne_result = pd.DataFrame(tsne_result,columns=["特性一","特性二"])#将数据转换为数据框
kmean_outlier_detection(tsne_result)
from sklearn.manifold import MDS
mds = MDS(n_components=2)
mds_result = mds.fit_transform(data)
mds_result = pd.DataFrame(mds_result,columns=["特性一","特性二"])
kmean_outlier_detection(mds_result)
from sklearn.manifold import Isomap
isomap = Isomap(n_components=2)
isomap_result = isomap.fit_transform(data)
isomap_result = pd.DataFrame(isomap_result,columns=["特性一","特性二"])
kmean_outlier_detection(isomap_result)
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca_result = pca.fit_transform(data)
pca_result = pd.DataFrame(pca_result,columns=["特性一","特性二"])
kmean_outlier_detection(pca_result)#对原始数据进行离群点检测
总结
我们遇到的数据通常都会存在离群点,离群点的检测对我们后期的模型的训练有着重要的意义,在高质量的数据集上进行训练往往比在更多数量的低质量数据集上进行训练的效果更好。