机器学习入门(九):非监督学习:5种聚类算法+2种评估模型
机器学习入门专栏其他章节:
机器学习入门(一)线性回归
机器学习入门(二)KNN
机器学习入门(三)朴素贝叶斯
机器学习入门(四)决策树
机器学习入门(五)集成学习
机器学习入门(六)支持向量机
机器学习入门(七)多项式回归
机器学习入门(八)主成分分析
文章目录
-
-
- 聚类算法
-
- 距离公式
- K-means算法
-
- 直接调用实现
- 基于EM的实现
- 局限性
- Hierarchical Clustering算法
-
- 代码实现
- 局限性
- GMM(Gaussian Mixture Model高斯混合模型)
-
- 代码实现
- 局限性
- DBSCAN
-
- 算法详解
- 代码实现
- 基于网格的聚类算法
-
- 代码实现
- 局限性
- 模型评估evaluation
-
- elbow method
-
- 代码实现
- silhouette analysis
-
聚类算法(Clustering Algorithms)常用于进行非监督学习(unsupervised learning),即它处理的是没有事先标记分类的数据。一共介绍五种常见聚类算法:
K-means
Hierarchical
GMM
DBSCAN(基于密度的聚类算法)
基于网格Grid的聚类算法
聚类算法
距离公式
在了解聚类算法如何实现之前,需要先了解几种常见的距离计算公式,因为聚类算法会通过距离判断两个点是否属于同一类。
欧式距离:假设有两个点 ( x 1 , y 1 ) , ( x 2 , y 2 ) (x_1,y_1),(x_2,y_2) (x1,y1),(x2,y2)则距离d为:
d = ( x 1 − x 2 ) 2 + ( y 1 − y 2 ) 2 d=\sqrt{(x_1-x_2)^2+(y_1-y_2)^2} d=(x1−x2)2+(y1−y2)2
扩展到更加一般的情况则是:
d = ( Σ i = 1 n ( x 1 ( i ) − x 2 ( i ) ) 2 ) 1 / 2 d=(\Sigma_{i=1}^n (x_1^{(i)}-x_2^{(i)})^{2})^{1/2} d=(Σi=1n(x1(i)−x2(i))2)1/2
曼哈顿距离:
对于两个点则是:
d = ∣ x 1 − x 2 ∣ + ∣ y 1 − y 2 ∣ d=|x_1-x_2|+|y_1-y_2| d=∣x1−x2∣+∣y1−y2∣
其他比较出名的还有Minkowski明氏距离, chebyshev切比雪夫距离,cosine余弦距离等,再次不过多展开。
K-means算法
算法过程如下:
(1)初始化聚类点centroid
(2)计算距离,将每个点分配到最近的聚类
(3)取每个聚类的平均值,获得新的聚类点centroid
(4)重复(2),(3)直到不再变化
用数学公式表达就是SSE(sum of squared error) 达到最小的时候:
S S E = Σ j = 1 k Σ x ∈ C j d ( x , m j ) 2 SSE=\Sigma_{j=1}^k \Sigma_{x\in {C_j}}d(x,m_j)^2 SSE=Σj=1kΣx∈Cjd(x,mj)2第一个求和是所有的聚类,第二个求和是每个聚类的中心点到聚类中所有点的距离平方和。
直接调用实现
基于sklearn,可以直接调用现有的kmeans算法。
首先导入包:
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets.samples_generator import make_blobs
X, y_true = make_blobs(n_samples=300, centers=4,
cluster_std=0.60, random_state=0)
from sklearn.cluster import KMeans,MiniBatchKMeans
#训练模型
k=4
kmeans = KMeans(n_clusters=k)
minimeans=MiniBatchKMeans(n_clusters=k)
kmeans.fit(X)
y_kmeans = kmeans.predict(X)
y_kmeans2 = minimeans.fit_predict(X)
#绘制图像
plt.scatter(X[:, 0], X[:, 1], c=y_kmeans, s=50, cmap='viridis')
centers = kmeans.cluster_centers_
plt.scatter(centers[:, 0], centers[:, 1], c='black', s=200, alpha=0.5);
plt.show()
plt.scatter(X[:, 0], X[:, 1], c=y_kmeans2, s=50, cmap='viridis')
centers2 = minimeans.cluster_centers_
plt.scatter(centers2[:, 0], centers2[:, 1], c='black', s=200, alpha=0.5);
plt.show()
查看结果:
kmeans模型的训练结果
MiniBatchMeans的训练结果:

MiniBatchKmeans是另一种优化实现,它比Kmeans更快,但结果可能不同
基于EM的实现
EM算法分为E和M两个步骤,具体步骤和之前类似:
(1)随机初始化聚类中心点
(2)计算距离,分配点
(3)重新获得中心点
(4)重复(2),(3)直到停止
代码实现
from sklearn.metrics import pairwise_distances_argmin
def find_clusters(X, n_clusters, rseed=2):
rng = np.random.RandomState(rseed)
#随机初始化
i = rng.permutation(X.shape[0])[:n_clusters]
centers = X[i]
while True:
#计算距离
labels = pairwise_distances_argmin(X, centers, metric='euclidean')
#重新计算中心点
new_centers = np.array([X[labels == i].mean(0)
for i in range(n_clusters)])
#判断是否停止
if np.all(centers == new_centers):
break
centers = new_centers
return centers, labels
#获得图像
centers, labels = find_clusters(X, 4)
plt.scatter(X[:, 0], X[:, 1], c=labels,
s=50, cmap='viridis');
plt.scatter(centers[:,0],centers[:,1],c='red',s=100,alpha=0.5)
plt.show()
结果跟上面的图类似,不再展示。
局限性
kmeans 算法对于不同密度,不同大小的,或者非球形形状的数据,有一定限制
from sklearn.datasets import make_moons
X, y = make_moons(200, noise=.05, random_state=0)
labels = KMeans(2, random_state=0).fit_predict(X)
plt.scatter(X[:, 0], X[:, 1], c=labels,s=50, cmap='viridis')
plt.show()
传统的kmeans表现非常不好:

这个时候有一种优化方式,类似SVM算法中的核函数,具体过程不展开,直接调用现有算法实现:
from sklearn.cluster import SpectralClustering
model = SpectralClustering(n_clusters=2, affinity='nearest_neighbors',assign_labels='kmeans')
labels = model.fit_predict(X)
plt.scatter(X[:, 0], X[:, 1],c=labels,s=50,cmap='viridis')
plt.show()

kmeans算法还有个缺点是,需要提前设置好k,即聚类cluster的个数,这种设置在某种程度上不太像非监督学习,接下来的有些算法则可以避免这个问题。
Hierarchical Clustering算法
Hierarchical 层次聚类这种算法不需要预设K值,它通过将距离较近的点或者聚类融入到一起,最后会实现一种树形结构的等级关系图,这也是hierarchical(层级,等级制度)这个名字的由来。
这个算法分为自顶向下(top down, Divisive)和自下而上(bottom up, Agglomerative)两种生成方式
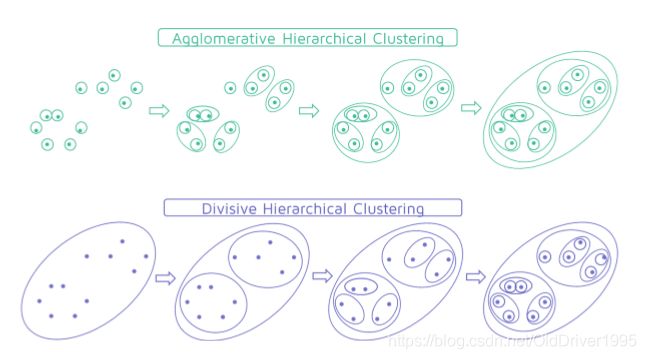 由于涉及到计算聚类之间的距离,这里参考的不是距离的计算方式,而是考虑聚类的哪一个点用来计算距离(在kmeans中是每个点和平均值点计算距离),一共三种方式:
由于涉及到计算聚类之间的距离,这里参考的不是距离的计算方式,而是考虑聚类的哪一个点用来计算距离(在kmeans中是每个点和平均值点计算距离),一共三种方式:

single-link: 距离最短的点
complete-link:距离最大的点
average-link:两个类中每个点的距离的平均值
代码实现
导入包
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets.samples_generator import make_blobs
X
from scipy.cluster.hierarchy import dendrogram, linkage
用现有的函数可以很快生成
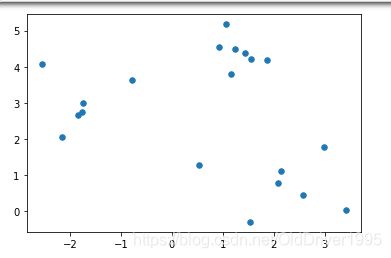
X, y_true = make_blobs(n_samples=20, centers=3,cluster_std=0.60, random_state=0)
plt.scatter(X[:, 0], X[:, 1], s=30)
plt.show()
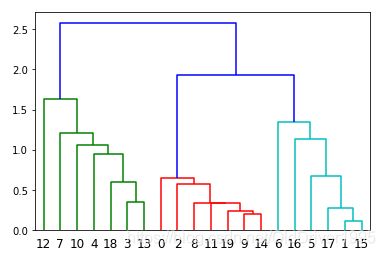
link_matrix=linkage(X,'single')
dendrogram(link_matrix)
plt.show()
局限性
对噪音敏感
对非球形数据处理效果不佳
GMM(Gaussian Mixture Model高斯混合模型)
先看这么一个例子,将数据进行一定的处理,使得它们非常紧密:
rng = np.random.RandomState(13)
X, y_true = make_blobs(n_samples=400, centers=4,cluster_std=0.60, random_state=0)
X = X[:, ::-1]
X2 = np.dot(X, rng.randn(2, 2))
kmeans=KMeans(4, random_state=0)
labels = kmeans.fit_predict(X2)
plt.scatter(X2[:, 0], X2[:, 1], c=labels,s=50, cmap='viridis')
plt.show()
 因为kmeans是取均值求中心点,但有的时候均值并不是最好的方式,如上图这种形式就不适合kmeans,使用高斯混合模型可以避免这种情况,在某种程度上,kmeans是GMM的一种特例,他们本身都是基于EM算法的思想实现的。
因为kmeans是取均值求中心点,但有的时候均值并不是最好的方式,如上图这种形式就不适合kmeans,使用高斯混合模型可以避免这种情况,在某种程度上,kmeans是GMM的一种特例,他们本身都是基于EM算法的思想实现的。
代码实现
首先有一个画椭圆范围的函数,如果看不懂不用深入,直接调用
from matplotlib.patches import Ellipse
def draw_ellipse(position, covariance, ax=None, **kwargs):
"""Draw an ellipse with a given position and covariance"""
ax = ax or plt.gca()
# Convert covariance to principal axes
if covariance.shape == (2, 2):
U, s, Vt = np.linalg.svd(covariance)
angle = np.degrees(np.arctan2(U[1, 0], U[0, 0]))
width, height = 2 * np.sqrt(s)
else:
angle = 0
width, height = 2 * np.sqrt(covariance)
# Draw the Ellipse
for nsig in range(1, 4):
ax.add_patch(Ellipse(position, nsig * width, nsig * height,angle, **kwargs))
然后是gmm的实现:
from sklearn.mixture import GaussianMixture as GMM
gmm = GMM(n_components=4, random_state=42, max_iter=1, warm_start=True)
labels2 = gmm.fit_predict(X2)
plt.scatter(X2[:, 0], X2[:, 1], c=labels2, s=40, cmap='viridis', zorder=2)
w_factor = 0.2 / gmm.weights_.max()
for pos, covar, w in zip(gmm.means_, gmm.covariances_, gmm.weights_):
draw_ellipse(pos, covar, alpha=w * w_factor)
局限性
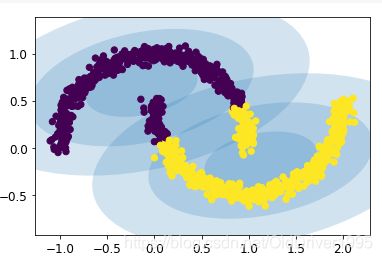
在这个例子中,gmm发挥良好,但是在他和kmeans一样,对于非球形数据的表现依旧不是很好,比如之前的月牙形数据,他就不能很好发挥。对于形状比较诡异,密度不均的数据,就要用到后面的算法。
DBSCAN
DBSCAN的全称是(Density Based Spatial Clustering of Applications with Noise) 从名字上来看就得知它是基于密度的,并且可以应付噪音。
算法详解
DBSCAN首先要直到两个参数:
Epsilon ϵ \epsilon ϵ, 它决定了一个点的“势力范围”,如果某个点的邻居想要和他属于同一类,则需要距离小于这个半径

MinPts: Minimum Points,限定了最少在规定的 ϵ \epsilon ϵ 半径内,有多少个点

看到这里不是很懂,没有关系,继续看规定了三种点的类型,分别是:
core point 核心点:一个点在规定半径 ϵ \epsilon ϵ 内,含有至少MinPts这么多个点,那这个点即为核心点
border point 边界点: 在核心点旁边,但半径内个数不足MinPts
Noise/Outlier point 噪音/外部点:既不是核心点也不是边界点。
三种点如下图:
关于半径 ϵ \epsilon ϵ的选择,如果太大,则所有点都是core点,反之,所有点都是noise了。
三种点之间的关系:

图中,所有红点都是core point,黄点是border point,蓝点是noise
Directly Density Reachable:
距离小于 ϵ \epsilon ϵ,并且直接和core point相连。比如,所有红点和它旁边紧挨着的红点都是directly reachable,C点和它旁边的core红点也属于这样的关系。但需要注意的是reachable关系都不是对称的,只能说core point reaches border point(图中指向黄点的箭头是有方向的,而红点之间的箭头是双向的),而一个边界点是不能被reach的(维基解释:Reachability is not a symmetric relation: by definition, only core points can reach non-core points. The opposite is not true, so a non-core point may be reachable, but nothing can be reached from it)
Density Reachable:
两个点,没有直接相连,但他们中间存在着至少一个点,每个点相互之前都是directly reachable的,有点类似于传递。比如图中左边的红点和右边的红点就是这种关系。同样地,B点和A点也是这样地关系,但BC不是,因为reachable关系必须满足其中某一方是core point。如果一个点是和一个cluster中的某个点是reachable关系,则这个点也属于这个cluster,所以BC也属于这个cluster。
Density Connected:
存在两个点p,q,如果有另一个点o和他们依次都是reachable的,那么p和q就是connected状态,这种关系适用于B和C,他不要求其中某一方是core。更广泛的情况,一个聚类中,任意两个点都是connected的。
当清楚了这几种关系后,DBSCAN算法的执行过程就可以理解了。在遍历点的时候,对与其中一个点,它会找到所有和这个点density-connected的点,把他们全部都放到同一个cluster中,直到没有点和它connected,进而前往下一个没有遍历过的点(所有connected的点都属于遍历过的)。
代码实现
生成数据
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=1000, noise=0.05, random_state=42)
plt.scatter(X[:,0], X[:,1], s=10)
from sklearn.cluster import DBSCAN
dbscan = DBSCAN(eps=0.05, min_samples=5)
dbscan.fit(X)
###查看一共生成了多少个cluster
np.unique(dbscan.labels_)
生成如下结果, 一共7个类+noise:
array([-1, 0, 1, 2, 3, 4, 5, 6])
如果只想看最简单的图像,可以直接这样:
plt.scatter(X[:,0], X[:,1],c=dbscan.labels_, s=40, cmap='viridis')
如果想看的更加仔细,可以像下面这样设置,把三种不同的点分别取出来:
首先弄一个全False数组,维度跟dbscan.labels一样。
由于dbscan自动记录core point,所以直接用core_sample_indices把对应的点先设成True
然后noise是所有labels==-1的点,这个方式同样可以获得一个True/False数组。
然后对于border point,用~这个操作取反。
具体代码如下:
#retrieve the index of 3 kinds of points
core_index=np.zeros_like(dbscan.labels_, dtype=bool) #创建一个跟labels长度一样,但全为T/F的数组
core_index[dbscan.core_sample_indices_]=True #dbscan自动已经存储了哪些是core,所以直接将对应index的设成True
noise_index=dbscan.labels_==-1
border_index=~(core_index | noise_index) #neither core, nor noise
#get correspond point
core=X[core_index]
border=X[border_index]
noise=X[noise_index]
#plot core point
plt.scatter(core[:,0], core[:,1],c=dbscan.labels_[core_index], s=40, cmap='viridis')
#plot border point
plt.scatter(border[:,0], border[:,1],c='grey', marker='^')
#plot noise
plt.plot(noise[:,0],noise[:,1],'rx')
结果:

灰色是border点,红x是噪音,剩余的是core点。注意所有的border其实也属于它附近的cluster,如果直接scatter所有的点,那他们border会跟对应的core是一种颜色。
可以发现,一共分出来7个cluster,效果不是很好
于epsilon设置的有点小,导致生成了很多类,现在适当修改一下参数,进行对比,将eps设置为0.08,获得如下图象,训练效果有一定提升。

eps设置为0.2:

可以看出,不同的参数,对结果的影响非常明显。所以参数的选择也应该仔细考虑
基于网格的聚类算法
代码实现
这里需要用到pyclustering包
!pip install pyclustering
from pyclustering.cluster.clique import clique,clique_visualizer
intervals = 30 #决定了网格的个数,interval越大,网格越多
threshold = 0 #每个点都会被考虑,网格中的点小于这个数值,该网格会被当成outlier
#数据还是之前的半月形数据X
clique_instance = clique(X, intervals, threshold)
clique_instance.process()
clusters = clique_instance.get_clusters()
noise = clique_instance.get_noise()
cells = clique_instance.get_cells()
clique_visualizer.show_grid(cells, X) #show formed grid
clique_visualizer.show_clusters(X, clusters, noise)
可以得到如下结果:

注意第二个图中有个小绿点,所以实际上有三个cluster
将intervals设成20,threshold设成4,由于阈值提高,很多网格不满足要求,会产生很多cluster
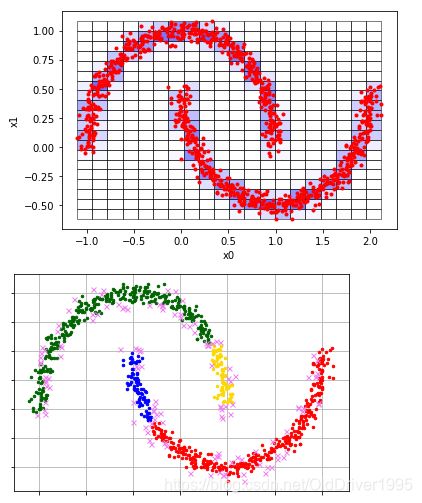
其中粉色点是噪音
局限性
不好处理高度不规则数据
受限于提前设置好的网格数量,边界,还有密度阈值(threshold)
不好处理高维数据。
模型评估evaluation
由于是非监督学习,没有y标签值判断是否分类正确,所以需要其他的方式来判断其算法模型的优劣。evaluation的方式有很多种,实际算法中并没有最,具体模型具体对待。评估方式主要说以下两种。
这里并不进行严格的数学证明,只是提供方式和思路。
elbow method
通过该算法可以选择最优的K数量(Kmeans中的K),即多少个聚类是最好的。该方法比较直观的理解就是,它可以找到一个K使得这个模型已经很好了,添加更多的聚类对结果影响不大,那么当前这个K就是一个比较合适的值.
首先对比一下两种模型
#设置数据
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
blob_centers = np.array(
[[ 0.2, 2.3],
[-1.5 , 2.3],
[-2.8, 1.8],
[-2.8, 2.8],
[-2.8, 1.3]])
blob_std = np.array([0.4, 0.3, 0.1, 0.1, 0.1])
X, y = make_blobs(n_samples=2000, centers=blob_centers,cluster_std=blob_std, random_state=7)
然后要写一个可以画决策边界的函数,这里还不是elbow method,只是简单对比一下不同K值的两个模型:
def plot_decision_boundary(clf , X):
mins = X.min(axis=0) - 0.1
maxs = X.max(axis=0) + 0.1
xp=np.linspace(mins[0], maxs[0], 500)
yp=np.linspace(mins[1], maxs[1], 500)
x1, y1=np.meshgrid(xp, yp)
xy=np.c_[x1.ravel(), y1.ravel()]
labels=clf.fit_predict(X)
y_pred = clf.predict(xy).reshape(x1.shape)
plt.contourf(x1, y1, y_pred, alpha=0.3, cmap=plt.cm.brg)
#画中心点
centers=clf.cluster_centers_
plt.scatter(centers[:,0], centers[:,1], s=200,c='red',alpha=0.5,zorder=2)
#zorder数值大的会显示在上面
plt.scatter(X[:,0], X[:,1], c=labels, cmap='viridis',s=20,zorder=1)
plt.show()
然后对比两个模型
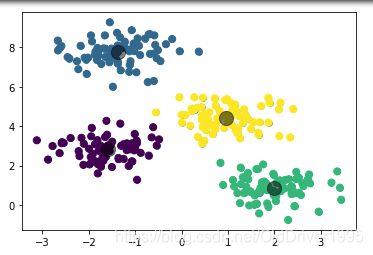
kmeans_k3 = KMeans(n_clusters=3, random_state=42)
kmeans_k8 = KMeans(n_clusters=8, random_state=42)
plot_decision_boundary(kmeans_k3, X)
plot_decision_boundary(kmeans_k8, X)
结果如下:

我们虽然可以调用模型的inertia参数,但随着K增大,inertia肯定会减少,但由于它是连续递减的,我们需要找到一个递减的”差不多“的位置。
代码实现
kmeans_per_k = [KMeans(n_clusters=k, random_state=42).fit(X)
for k in range(1, 10)]
inertias = [model.inertia_ for model in kmeans_per_k]
plt.plot(range(1,10), inertias, 'bo-')
plt.xlabel("$k$", fontsize=14)
plt.ylabel("Inertia", fontsize=14)
生成图像,有点类似于梯度下降:
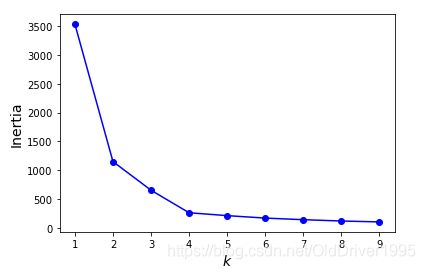
发现k=4的时候就已经下降的很缓慢了。看一下k=4的分类效果:
 这里只是比较直观的选择了K,实际上选择的时候需要一定的计算,不过这里省略了这一过程。
这里只是比较直观的选择了K,实际上选择的时候需要一定的计算,不过这里省略了这一过程。
silhouette analysis
侧影分析,他的原理是基于相似度计算实现的,计算某一个点和它同一个cluster的其他点的相似度,以及和其他cluster中每个点的相似度,然后会获得一个介于-1到1之间的silhouette score。这个数值越接近1,则说明这个点和同cluster的点越接近,和其他cluster中的点越远(距离上的远近)
然后每个点的score取平均,获得整个模型的score,越接近与1,说明模型越好。
首先plot一下图像:
from sklearn.metrics import silhouette_score
silhouette_scores = [silhouette_score(X, model.labels_)for model in kmeans_per_k[1:]]
plt.plot(range(2, 10), silhouette_scores, "bo-")
plt.xlabel("$k$", fontsize=14)
plt.ylabel("Silhouette score", fontsize=14)
import matplotlib as mpl
from sklearn.metrics import silhouette_samples
from matplotlib.ticker import FixedLocator, FixedFormatter
plt.figure(figsize=(10, 8))
for k in (3, 4, 5, 6):
plt.subplot(2, 2, k - 2)
plt.subplots_adjust(hspace=0.5)
y_pred = kmeans_per_k[k - 1].labels_
silhouette_coefficients = silhouette_samples(X, y_pred)
padding = len(X) // 30
pos = padding
ticks = []
for i in range(k):
coeffs = silhouette_coefficients[y_pred == i]
coeffs.sort()
color = mpl.cm.Spectral(i / k)
plt.fill_betweenx(np.arange(pos, pos + len(coeffs)), 0, coeffs, facecolor=color, edgecolor=color, alpha=0.7)
ticks.append(pos + len(coeffs) // 2)
pos += len(coeffs) + padding
plt.gca().yaxis.set_major_locator(FixedLocator(ticks))
plt.gca().yaxis.set_major_formatter(FixedFormatter(range(k)))
if k in (3, 5):
plt.ylabel("Cluster")
if k in (3, 4, 5, 6):
plt.gca().set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
plt.xlabel("Silhouette Coefficient")
else:
plt.tick_params(labelbottom=False)
plt.axvline(x=silhouette_scores[k - 2], color="red", linestyle="--")
plt.title("$k={}$".format(k), fontsize=16)
plt.show()
结果如下:
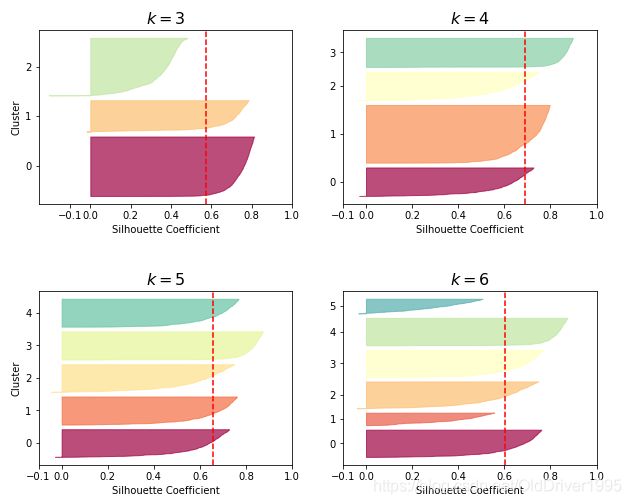
可以看出K=4的时候,silhouette score是最高的,尽管K=5中每个cluster更加均匀。和之前的elbow method的方法所得到的结果相符。