轻量级肝脏与肝脏瘤2.5D分割网络阅读笔记
点击上方“计算机视觉工坊”,选择“星标”
干货第一时间送达
作者丨李响
来源丨GiantPandaCV
文章目录
1 前言
2 方法概述
2.1 InceptionV1-V3 and convolution conversion
2.2 Residual block
2.3 2.5D 网络
3 RIU-Net 的整体结构
4 实验和可视化
5 总结
1 前言
最近一直在读医学图像的论文,于是我打算写一个系列的阅读笔记,语言比较精简。在上一篇阅读笔记(https://zhuanlan.zhihu.com/p/505483978)中,分析了医学图像分割的混合 Transformer 网络:UTNet,在这一篇笔记中介绍的网络与 UTNet 思路完全不同,追求的是比 U 型网络更轻量级,但准确率更高的设计。还是先给下论文地址(刚刚接收):https://www.sciencedirect.com/science/article/abs/pii/S1746809422000891 。
首先分析下这篇论文的动机,对于 LITS17 和 3DIRCADb 这类肝脏与肝肿瘤数据集,3D 卷积网络具有学习上下文特征的能力,有效利用 CT 图像的空间信息,但是训练和部署也会消耗大量计算资源。所以,既能利用切片间的空间信息,并且保证分割精准度,就是论文需要解决的问题。本篇阅读笔记首先对论文中涉及的方法进行概述;再详细介绍论文提出的 RIU-Net 结构;最后分析了实验和分割可视化部分,并简单的总结。
2 方法概述
这一部分作为前置依赖,类似于 Related Work,对涉及网络结构熟悉的读者可以略过相应的介绍。
2.1 InceptionV1-V3 and convolution conversion
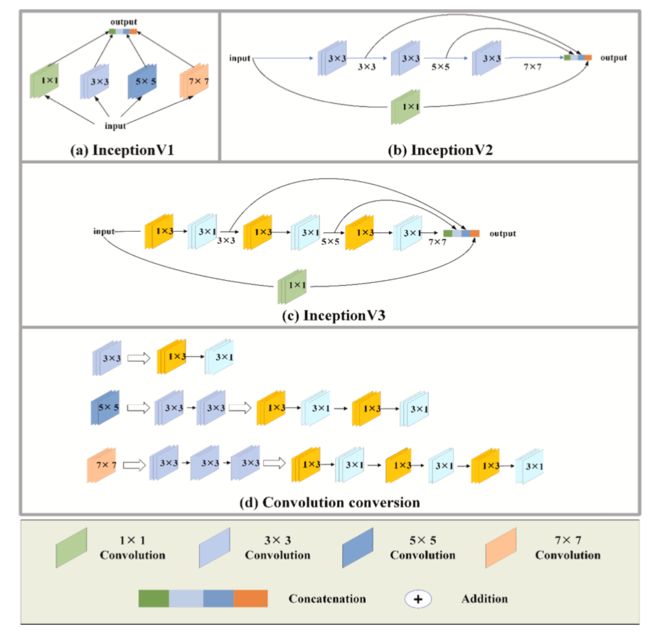
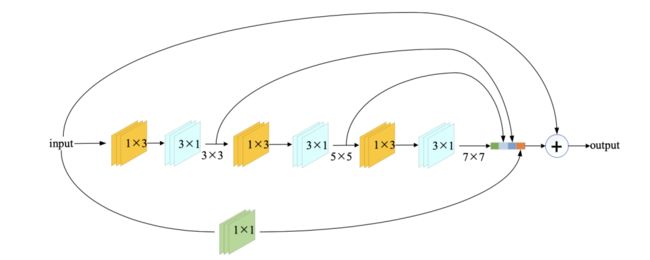
如下图,先看 InceptionV1 模块,它并行包含了 1×1、3×3、5×5 和 7×7 大小的卷积核。虽然该模块提高了网络的可行性,但是却增大了网络参数量。InceptionV2 使用两个 3×3 卷积核来代替一个 5×5 卷积序列,三个 3×3 卷积核来代替一个 7×7 卷积序列。InceptionV3 实现了将 N×N 的卷积序列替换 为 1×N 和 N×1 的卷积序列。因此,3×3 卷积序列可以替换为 1×3 和 3×1 的卷积组合, 同理,5×5 卷积序列可以替换为两组 1×3 和 3×1 的卷积组合,7×7 卷积序列可以替换为三组 1×3 和 3×1 的卷积组合,可以看下图(d),这里是很清晰的,这种处理让网络的参数量更理想化。
2.2 Residual block
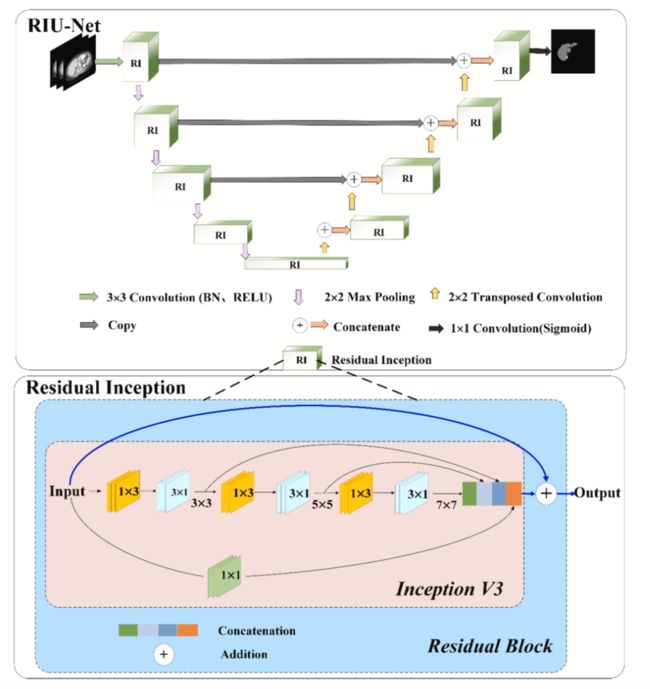
关于 Res-Net 的思想,我就不多介绍了。在这篇文章中,Residual block 也被嵌入到 InceptionV3 中,如下图所示。这样做,把 U-Net 的基础 block 替换为 RI(Res- Inception) 结构,就是更薄且更宽的 Inception 卷积结构为基础卷积序列带有残余连接,网络整体参数量比传统 U-Net 降低了 70%,节省了计算资源。
2.3 2.5D 网络
在医学图像分割中,充分医用好医学图像切片间的空间信息是至关重要的。直接将 3D 图像输入到网络中,3D 图像会占据巨大的内存,或者直接将 3D 图像转换为 2D 图像,这样也是不可取的,直接抛弃了医学图像切片间的空间信息。所以出现了 2.5D 的思想,即将一叠相邻切片作为网络的输入,并生成与中心切片的分割图,这样既能节省计算资源也能利用好空间信息。
3 RIU-Net 的整体结构
在上一部分,我们重点介绍了论文中涉及到的技术,基于此,我们来看下这个网络的整体设计。网络架构是以 U-Net 为原型设计的,模型依旧呈现出对称结构,左半部分是编码器,用于特征提取,右半部分是解码器,用于定位感兴趣区域,编码器与解码器之间普通的跳跃连接,实现低级语义信息与高级语义特征之间的组合。整体框架由 9 个 RI(Res- Inception) 模块、4 个下采样层、4 个上采样层和一个 1×1 的卷积层组成。如下图所示。
因为在肝脏与肝肿瘤分割中,尤其是肿区域大小不一,如果使用 U-Net 中固定的卷积序列,势必会导致感受野受到限制,降低分割精度。这样的设计可以用不同的卷积核来获得图像不同尺度的感受野,来保障分割精度。
4 实验和可视化
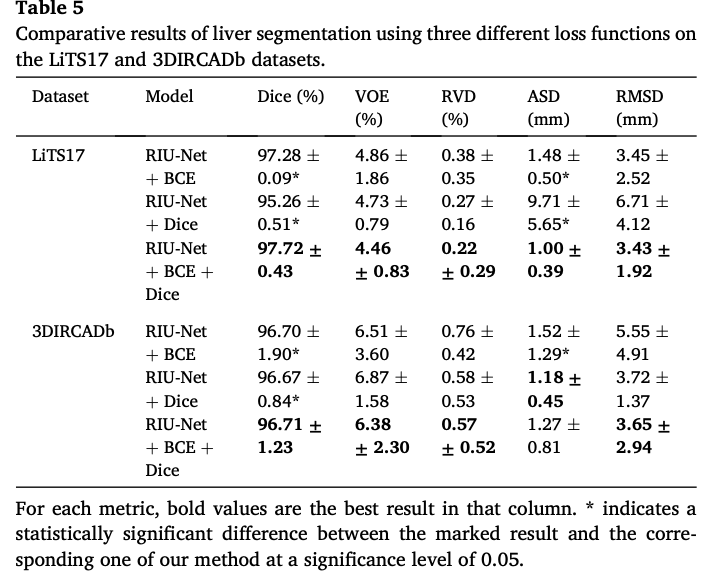
在这这篇论文中,实验和可视化的部分是可以重点借鉴的,使用 LiTS17 和 3DIRCADb 两个数据集就行训练、验证与测试。首先,我们来看下消融实验的部分,分别在两个数据集上就行消融实验分析。评价指标包括 Dice 系数、体积重叠误差(VOE)、相对体积误差(RVD)、平均对称表面距离(ASD)、均方根对称面距离(RMSD)。
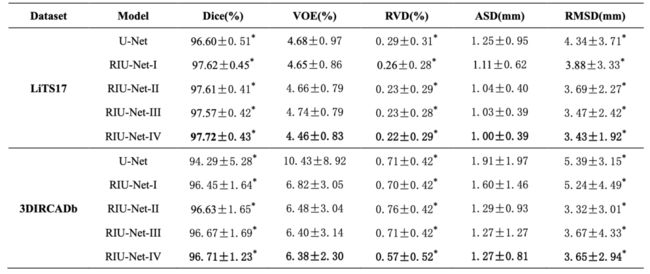
下表是消融分析在 LiTS17/3DIRCADb 数据集上的肝脏定量分割结果,其中,RIU-Net-I 到 RIU-Net-IV 为不同的Inception,这个对应在第二部分的介绍中。
 下表是消融分析在 LiTS17/3DIRCADb 数据集上的肝肿瘤定量分割结果。
下表是消融分析在 LiTS17/3DIRCADb 数据集上的肝肿瘤定量分割结果。 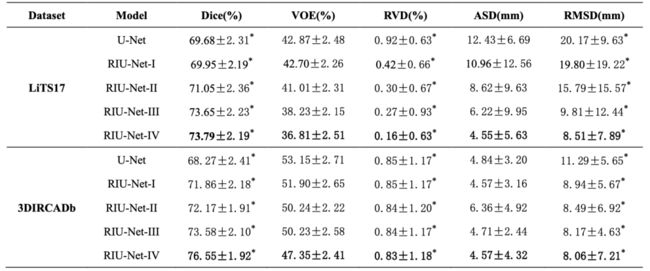
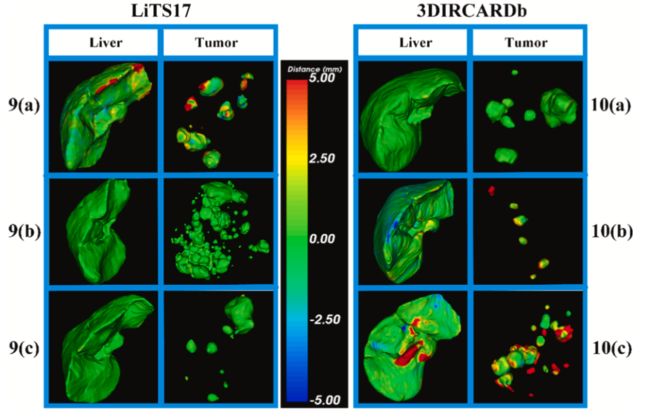
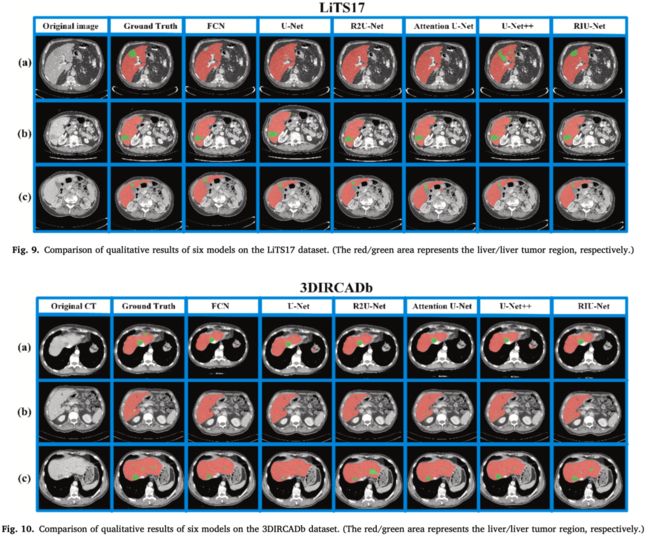
上面两个表对应的可视化对应下图,红色代表肝脏区域,绿色代表肿瘤区域。
对比实验基于 VGGnet 框架的 FCN 网络、U-Net、R2U-Net、Attention U-Net 和 U-Net++ 网络,如下,分别是肝脏定量分割结果和肝肿瘤定量分割结果。RIU-Net 不但精度更高,而且波动范围较小。
对应的,下图表示 6 种模型在 LiTS17 和 3DIRCADb 数据集上的可视化分割比较结果,在处理肝脏边界含有肿瘤与小肿瘤难分割这两种情况时,论文中提出的网络效果更好。
最后,在下图中,分别在 LiTS17 和 3DIRCAb 数据集上对肝脏和肝脏肿瘤分割进行 3D 错误可视化(红色和蓝色区域分别表示明显的过分割和欠分割错误,而绿色区域表示与金标准的一致性)。
5 总结
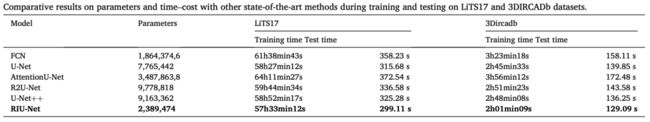
这篇论文在保证精度的同时,大幅减少了医学图像分割网络的参数量。所以关于训练和推理时间,是需要我们重点关注的。如下表,对比时间成本达到了最低。
总结一下,这篇论文提出了一种医学图像肝脏与肝肿瘤分割网络,核心思想是建立一个提取图像多尺度信息又可以充分节省计算资源的轻量级网络。引入了残差,结合不同分辨率的特征图,可以避免过拟合。为了节省计算资源,将 U-Net 中的所有卷积序列替换成 Inception 模块,不仅减少了参数量也能够提取更多图像特征。另外,为了解决输入网络结构中的 2D 图像无法利用医学图像切片间的空间信息以及 3D 图像又会占据巨大的内存的问题,论文中采用了 2.5D 的输入形式,即将一叠相邻切片作为网络的输入,并生成与中心切片的分割图。最后,做了丰富的可视化工作。
本文仅做学术分享,如有侵权,请联系删文。
干货下载与学习
后台回复:巴塞罗那自治大学课件,即可下载国外大学沉淀数年3D Vison精品课件
后台回复:计算机视觉书籍,即可下载3D视觉领域经典书籍pdf
后台回复:3D视觉课程,即可学习3D视觉领域精品课程
计算机视觉工坊精品课程官网:3dcver.com
1.面向自动驾驶领域的多传感器数据融合技术
2.面向自动驾驶领域的3D点云目标检测全栈学习路线!(单模态+多模态/数据+代码)
3.彻底搞透视觉三维重建:原理剖析、代码讲解、及优化改进
4.国内首个面向工业级实战的点云处理课程
5.激光-视觉-IMU-GPS融合SLAM算法梳理和代码讲解
6.彻底搞懂视觉-惯性SLAM:基于VINS-Fusion正式开课啦
7.彻底搞懂基于LOAM框架的3D激光SLAM: 源码剖析到算法优化
8.彻底剖析室内、室外激光SLAM关键算法原理、代码和实战(cartographer+LOAM +LIO-SAM)
9.从零搭建一套结构光3D重建系统[理论+源码+实践]
10.单目深度估计方法:算法梳理与代码实现
11.自动驾驶中的深度学习模型部署实战
12.相机模型与标定(单目+双目+鱼眼)
13.重磅!四旋翼飞行器:算法与实战
14.ROS2从入门到精通:理论与实战
15.国内首个3D缺陷检测教程:理论、源码与实战
重磅!计算机视觉工坊-学习交流群已成立
扫码添加小助手微信,可申请加入3D视觉工坊-学术论文写作与投稿 微信交流群,旨在交流顶会、顶刊、SCI、EI等写作与投稿事宜。
同时也可申请加入我们的细分方向交流群,目前主要有ORB-SLAM系列源码学习、3D视觉、CV&深度学习、SLAM、三维重建、点云后处理、自动驾驶、CV入门、三维测量、VR/AR、3D人脸识别、医疗影像、缺陷检测、行人重识别、目标跟踪、视觉产品落地、视觉竞赛、车牌识别、硬件选型、深度估计、学术交流、求职交流等微信群,请扫描下面微信号加群,备注:”研究方向+学校/公司+昵称“,例如:”3D视觉 + 上海交大 + 静静“。请按照格式备注,否则不予通过。添加成功后会根据研究方向邀请进去相关微信群。原创投稿也请联系。
▲长按加微信群或投稿
▲长按关注公众号
3D视觉从入门到精通知识星球:针对3D视觉领域的视频课程(三维重建系列、三维点云系列、结构光系列、手眼标定、相机标定、激光/视觉SLAM、自动驾驶等)、知识点汇总、入门进阶学习路线、最新paper分享、疑问解答五个方面进行深耕,更有各类大厂的算法工程人员进行技术指导。与此同时,星球将联合知名企业发布3D视觉相关算法开发岗位以及项目对接信息,打造成集技术与就业为一体的铁杆粉丝聚集区,近4000星球成员为创造更好的AI世界共同进步,知识星球入口:
学习3D视觉核心技术,扫描查看介绍,3天内无条件退款
圈里有高质量教程资料、可答疑解惑、助你高效解决问题
觉得有用,麻烦给个赞和在看~