基于随机森林的植物环状RNA识别软件
基于随机森林的植物环状RNA识别软件
摘要
环状RNA是一种新型的闭合RNA。越来越多的环状RNA在植物中被发现,研究表明环状RNA具有很重要的调控作用。
因此,从RNA序列中识别环状RNA非常重要。但是传统的环状RNA识别存在局限性。
近年来机器学习的提出,给环状RNA识别提供了很多方法。但是植物序列RNA特征与动物序列RNA特征不同,利用这些方法来识别植物RNA是不可行的。
因此迫切需要开发一款基于植物环状RNA的特征识别软件。
结果
本文构建了一个名为PCirc的软件程序,使用机器学习的方法通过RNA序列数据预测植物环状RNA。
过程
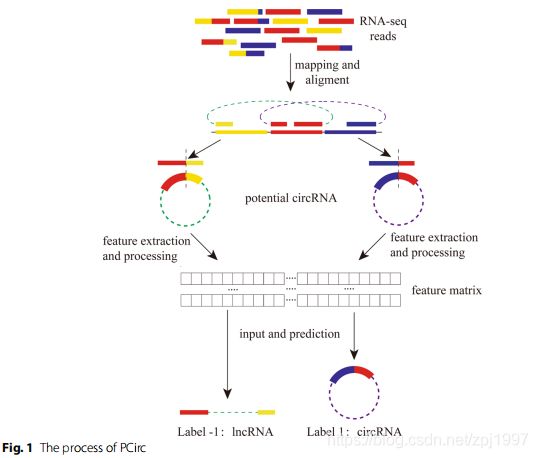
首先:从水稻环状RNA和lncRNA数据中提取了不同的特征,包括开放阅读框、k-mers数量和剪接连接序列编码。
第二:使用随机森林模型进行训练,训练集采用十倍交叉验证。
第三:根据 准确率、精度 和 F1得分 来评估我们的分类,模型测试数据的所有得分都在0.99以上。
第四:通过其它植物数据进行验证。得到了良好的结果,准确率达到了0.8以上。
最后,将构建的机器学习模型和使用的编程脚本打包到本地运行的循环RNA预测软件Pcirc https://github.com/Lilab -SNNU/Pcirc
总结
本研究基于水稻circRNA和lncRNA数据,利用随机森林算法构建了植物circRNA识别的机器学习模型,该模型也可以应用于植物circRNA识别。同时,将构建的机器学习模型和本研究使用的编程脚本打包成本地化的circRNA预测软件Pcirc,方便植物circRNA研究人员使用。
背景
环状RNA是一种新发现的非编码RNA。与典型的线性RNA不同,它没有5 '端帽结构,也没有3 '端poly-A尾巴结构,而是5 '端与3 '端,端对端连接形成的闭环结构。
CircRNA于20世纪70年代首次在植物类病毒中被发现,但由于其低水平表达,被认为是转录的副产物。
随之技术的进展,在原核生物与真核生物钟发现大量环状RNA,其中一些环状RNA被证实可以编码蛋白。虽然目前对环状RNA研究很多,但大多集中在哺乳动物和人类,对植物环状RNA研究较少。
目前,CIRI、CIRCexplorer2和find_circ是比较流行的circRNA识别软件。这三种软件程序能够从转录组数据中预测环状RNA的重要共同基础之一是覆盖环状RNA反剪接连接(BSJs)的支持读数。但是,每个预测软件支持reads数的经验标准不同,导致预测的circRNAs数量差异很大,不同软件程序得到的重叠circRNAs数量较少。同时,由于不同阶段和不同组织中环状RNA的表达不尽相同,因此将连接读数作为预测环状RNA的重要标准,很容易丢失一些环状RNA。为了克服上述缺点,我们开发了一种基于机器学习方法的动物circRNA识别方法DeepCirCode,并在哺乳动物上取得了良好的效果。
DeepCirCode使用的特征包括GT-AG剪接位点、反向剪接位点上下游的Alu重复序列,以及circRNA剪接位点两端与基因组相反的序列方向。然而,利用这些特征来识别植物环状rna是不可行的,因为植物环状rna序列的特征与动物环状rna不同。
为此,迫切需要基于植物环状rna的特征开发基于机器学习的环状rna识别软件。
在本研究中,我们首先将环状RNA和lncRNAs分别作为正集和负集,根据k-mers、ORFs的主要特征以及覆盖反剪接位点的序列编码信息,构建了机器学习模型。Pcirc的源代码和安装说明可以在https上://githu b.com/Lilab -SNNU/Pcirc找到。
Dataset
circRNA数据下载自PlantCircBasehttp://ibi.zju.edu.cn/plantcircbase。incRNA下载自GreeNC http://green.sciencedesigners.com
为了使数据集更可信,我们首先比较了circRNAs数据与lncRNA数据,筛选的序列与序列相似性高于95%的两个数据集,然后比较了序列在每个数据集和删除那些相似度高于95%,保持最长的一个。
最后,我们使用盒须图方法从两个数据集中删除极端数据值(长度过长或过短)。综上所述,我们分别获得33101个环状rna和4656个lncrna阳性数据和阴性数据。我们随机选择4000从积极和消极的数据序列,然后有8000序列作为训练集,剩下的数据作为一个独立的测试集。circRNAs和lncRNAs拟南芥和玉米也从PlantCircBase下载和GreeNC分别,然后用来测试Pcirc的普遍性。
Random forest
随机森林(Random forest, RF)是由训练数据的独立bootstrap样本和独立于原始特征空间[23]采样的每个特征子集组成的多个未修剪决策树的集合。它可以从训练数据集的原始特征构造多个独立的决策树,然后通过投票的方式对所有树进行融合,得到最优的分类模型。该方法在包括生物信息学在内的数据处理领域得到了广泛的应用。K-nearest neighbors (KNN)是ML领域另一种流行的算法;虽然KNN也能很好地处理分类问题,但算法中的许多参数需要调整。随着KNN中关键参数数量的增加,计算量会迅速扩大,而RF只需要调整少量的模型参数就可以得到一个很好的预测分类模型。
Pcirc软件的实验设置
本研究用的是 sciki-learn模块,该模块包含很多机器学习算法。然后根据以下流程基础来开发了Pcirc软件。
随机森林的参数为:树的数目为n_estimators=100,其他参数为默认值。
GNB:为默认参数
SVC(支持向量机):哈希值为{C:10, gammer:0.001, kernel: ‘rbf ’}
KNN:{n_neighbors:7, weights: ‘uniform’ , p:2} 其他参数为默认值。
特征提取
从训练集中提取具有识别能力的特征是构建ML模型的关键步骤。一些简单的单一特征,如GC含量和序列长度,并不能轻易直接区分阳性数据的环状rna和阴性数据的lncRNAs。
本研究选择k-mers、开放阅读框(open reading frames, ORFs)和覆盖反剪接位点的结序列作为Pcirc的主要特征。
K-mer:
其中k的取值为{1,2,3,4} ,构成一个340维的向量,其中每一个成员Fi是其出现的频率,即每种元素出现的次数除以序列长度。向量最后再引入GC含量,构成341维特征向量。
ORF(open reading frames)
开放阅读框(ORF)是序列中的一个重要特征。通常,它是序列中具有蛋白质编码功能的部分。许多研究表明,环状RNA的ORF与编码序列有显著差异。具体来说,circRNA序列的ORF越短,整个序列的orf覆盖范围越小。因此,我们在本研究中使用ORF-length和ORF-coverage作为一组特征(3)。ORF-length是指序列中ORF的长度,ORF-coverage是指ORF的长度与序列总长度之比。
首先我们使用UGENE http://ugene .unipr o.ru/downl oad.html去预测序列中的ORF,然后使用Python脚本从结果文件中提取最佳ORF,并计算它所占序列的长度比例。对于ORF-coverage,我们在提取特征时对值进行了* 10的标准化。
![]()
Splicing junction sequence coding (SJSC)拼接序列编码
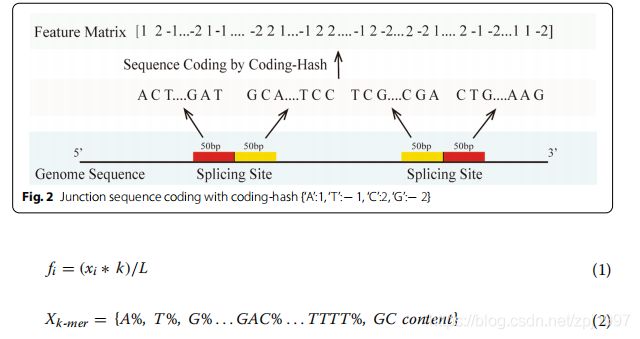
基因转录过程涉及许多可变的剪接事件,不同的剪接位点导致不同的转录本,尤其是环状rna。目前,环状rna中的拼接信号GT/AG可被RBP识别形成环状rna。由于RBP的结合区域通常位于反剪接位点的上游和下游,因此反剪接位点上游和下游的序列信息可能是区分环状rna与其他序列的有用特征。
对于back-splicing网站,我们位于基因组拼接两个地方因为没有back-splicing lncRNAs网站,然后提取序列50个基点上游和下游50个基点的基因组序列中的每个拼接形成一个数据集,记录100个基点的长度连接序列{“a”:1、“T”:−1,“C”:2,“G”:−2}(图2),并得到一个一维数组的矩阵组成的一组{1 2−1−2},可以认识到一台电脑。将所有数据集重新编码后,生成一组相应的高维数组矩阵,用于训练和测试。
模型测量
评估指标采用了:召回率,精确率,正确率,F1分数,MCC

结果
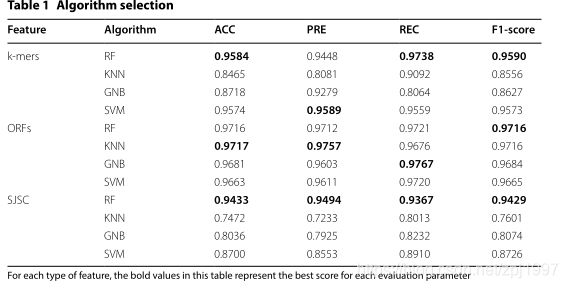
1、算法比较
本文采用了四种流行算法:随机森林、K近邻算法、支持向量机、高斯朴素贝叶斯。
为了选择合适算法,在三种特征集上分别重复十次十倍交叉验证对比。
结果表明,随机森林算法成绩最棒,最低分数为0.9433和它们之间的最大差值小于0.05,因此被选为最稳定的模型构建算法。
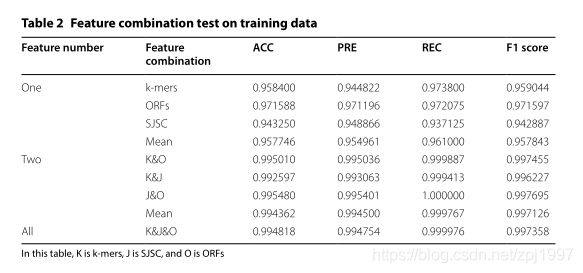
2、特征联合分析
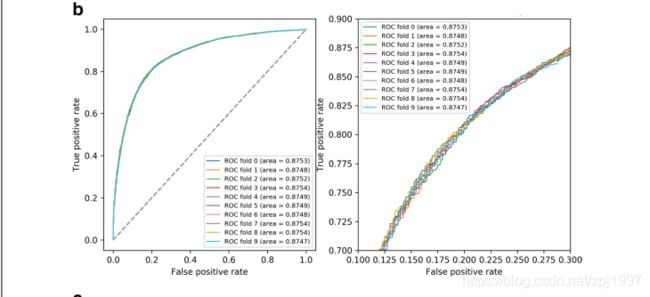
在本研究中,我们选择了三种特征作为ML在软件中的主要特征来构建模型。为了检验所选择的特征是否能够准确区分阳性和阴性类别,我们在训练集上分别对三类特征进行了测试(表2,图4)。可以看出,我们选择的三类特征都有很好的分类效果;各特征组合得分均在0.9以上,各特征组合得分均在0.99以上,在ROC曲线上,组合结果的参数评价优于单特征分析。分数也随着特征数量的增加而增加。为了避免模型过拟合,我们还在测试集上进行了相应的测试(图5)。可以看出,在测试集上,多特征组合的建模结果比任何单一类型特征的建模结果更好,更稳定。
3、模型评价与应用
为了得到最佳的预测结果,我们根据特征组合的结果建立模型。为了避免过度拟合或欠拟合,模型的拟合效果是由十倍交叉验证的方法评估和precision-recall曲线(额外的文件1:图S1,额外的文件2:文件1),和最终的模型评估测试集(表3,图6)。结果表明,我们的模型实现了精度0.9936,这表明,我们的模型可以分类circRNAs lncRNAs好。除了仅使用lncRNA作为阴性测试数据集外,我们还在我们的阴性数据集中加入了其他三种类型的非编码RNA miRNA、snRNA和snoRNA来测试我们的模型。结果表明,该方法的正确率为0.8935,正确率和召回率均达到90%以上(附加文件3:文件2.)。为了检验我们的模型在其他植物中是否也可靠,我们选取了双子叶植物拟南芥和单子叶植物玉米的circRNA和lncRNA数据来检验我们的模型(表3),分别获得了0.8980和0.8130的精度。综上所述,我们的结果表明,我们的模型不仅在模型构建的测试集上获得了准确的结果,而且可以有效地预测其他植物的环状rna。


讨论
环状rna是一类环状非编码rna,大部分长度大于200nt,而lncRNAs是一类长度大于200nt的线性非编码rna。因为circRNA和lncrna的长度分布几乎相同,所以单凭序列长度很难区分。将序列特征与机器学习相结合已被报道为一种有效的分类长链非编码rna[27]的方法。因此,我们从circRNA的生物学特性出发,构建了一个名为Pcirc的软件,利用机器学习方法对植物circRNA进行预测。在构建机器模型的过程中,我们选择了三种特征,其中k-mers和ORFs在lncrna和circrna的识别和预测中得到了广泛的应用。由于反向剪接位点在环状rna的形成中起着重要的作用,反向剪接位点的上下游序列引起了科学家们的关注。现在研究者开始研究通过剪接连接序列编码(SJSC)来研究环状rna的形成,但是我们的方法中SJSC的策略与之前的方法不同。
除了剪接位点相关的特征,我们结合广泛使用的k-mer和ORF特征来构建我们的ML模型,因为它们代表了序列的基础。k-mer是序列多样性的基础。随着k值的增加,在同一序列甚至同一基因组中越来越难以找到具有相同高k值的k-mer片段。同时,在一定范围内,k值越大,k-mer片段越具有代表性;但是较大的K值所需要的额外计算量呈指数级增加,所以我们需要更好地平衡特征优化和计算成本这两个问题。经过多次尝试,我们选择kmax = 4作为k-mers的代表值。四种核苷酸的顺序包含着重要的生物信息,ORF是一种具有代表性的信息类型。在我们的研究中,虽然这类特征是由二维向量组成的,但是ORFs经过处理后也包含了丰富的信息。从三种主要算法测试和单特征类型测试的结果可以看出,这种特征具有很好的分类能力(表1,2)。最初,由于lncrna与mrna在于编码能力差异较大,这种特征被广泛应用于识别lncRNAs。目前,circrna大多被认为是ncrna; 然而,由于一些环状rna已经被证明具有编码能力,我们使用这一特性进行测试,结果远远超出了我们的预期。可能是由于选择性剪接的问题,环状rna中包含的外显子比lncrna中包含的外显子更多,从而有可能获得更多的ORFs。也有可能是没有明确起始和终止位点的环状结构比具有明确起始和终止位点的线性序列具有更强的可翻译性,因此ORF特征在区分环状rna和lncRNAs方面具有很强的分类能力。在本研究中,在算法选择过程和特征组合测试中,ORF特征获得了最好的分数(表1、2)。在未来的研究中,我们将对预测的环状rna进行进一步的特征测试、编码分析和实验验证。