单细胞分析Scanpy(一):Anndata数据结构
Scanpy是一个分析单细胞转录组数据的python库,AnnData是scanpy的数据存储格式。
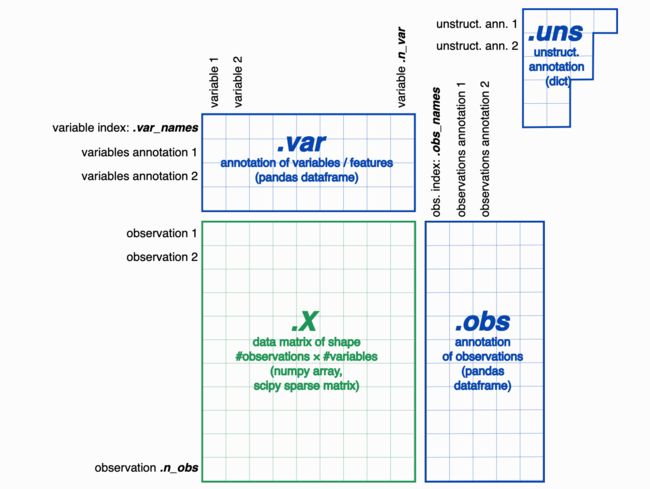
一、AnnData数据结构
1、功能介绍
| 结构 | 功能 | 数据类型 |
|---|---|---|
| adata.X | 矩阵数据 | unmpy数组、scipy sparse矩阵 |
| adata.obs | 观测值数据 | pandas Dataframe |
| adata.var | 特征和高可变基因数据 | pandas Dataframe |
| adata.uns | 非结构化数据 | 字典dict |
adata.X存储的是矩阵信息,它的结构虽然是一个数组,但是没有行名和列名信息。矩阵的行名信息存储在adata.obs中,列名信息存储在adata.var中。
一般情况,在单细胞处理中adata.obs存储的是细胞的数据(对应观测),adata.var存储的是基因的数据(对应特征)。
2、adata常用函数
# 数据数目统计
adata.n_obs # 返回细胞数 2695
adata.n_vars # 返回基因数 18270
adata.shape # (2695, 18270)
# 数据键值提取
adata.obs_keys() # 细胞注释信息的keys,比如 ['ClusterID', 'ClusterName', 'SCT_snn_res_0_8', 'nCount_SCT', 'nCount_Spatial', 'nFeature_SCT', 'nFeature_Spatial', 'orig_ident', 'seurat_clusters', 'imagecol', 'imagerow'']
adata.obs_names # 返回细胞ID 数据类型是object
adata.var.index # 返回基因 数据类型是object
adata.var_names.to_list() # 返回基因 数据类型是list
adata.obs.head() # 查看前5行的数据
# 其他的数据组成也可以使用
二、构造anndata数据
通过代码,手动创建一个Anndata数据:
import numpy as np
import pandas as pd
import anndata as ad
from string import ascii_uppercase
n_obs = 1000 # 设置观测样本的数量
obs = pd.DataFrame() # obs用于保存观测量的信息
# 生成观察时间
obs['time'] = np.random.choice(['day 1', 'day 2', 'day 4', 'day 8'], n_obs)
# 函数解释:numpy.random.choice(a, size = None)
# a是一维的数组,从a中随机抽取元素,并组成指定大小的数组
# 设置特征名
var_names = [i*letter for i in range(1, 10) for letter in ascii_uppercase]
# print(var_names)
# 输出为['A'~'z',...'AAAAAAAAA'~'ZZZZZZZZZ'] 从一个字母到九个重复的字母,共26*9=234
n_vars = len(var_names) # 输出大小为234个
var = pd.DataFrame(index=var_names)
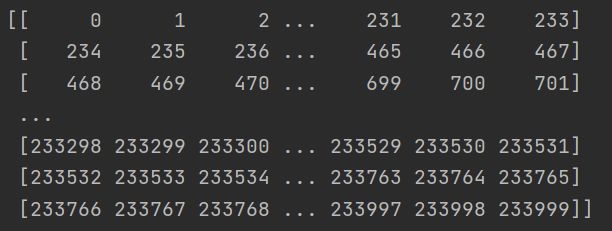
X = np.arange(n_obs * n_vars).reshape(n_obs, n_vars) X的输出为:
三、Anndata初始化
AnnData 对象默认采用数据类型 float32,可以更精确的存储数据,我们为了便于后期观察打印结果,设置数据类型为 int32:
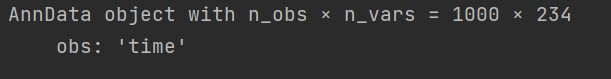
adata=ad.AnnData(X,obs=obs,var=var,dtype='int32')输出adata,得到: (列*行=1000*234)
四、函数学习
1、Anndata的切片特性
查看观测值和变量,也就是obs(列)的名字和var(行)的名字。
print(adata.obs_names[:10].tolist())
print(adata.obs_names[-10:].tolist())
print(adata.var_names[:10].tolist())
print(adata.var_names[-3:].tolist())
输出如下:
2、Anndata的view特性
我们每次操作 AnnData 时,并不是再新建一个 AnnData 来存储数据,而是直接找到已经在之前初始化好的 AnnData 的内存地址,通过内存地址来直接改变 AnnData 的值。这样做的好处是:无需分配多余的内存,可以直接修改已经初始后的Anndata对象。
获取与设置Anndata对象
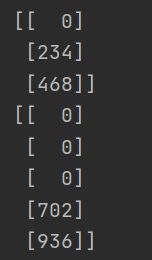
print(adata[:3, 'A'].X) # 查看 'A' 列的头三个元素
adata[:3, 'A'].X = [0, 0, 0] # 设置 'A' 列的头三个元素
print(adata[:5, 'A'].X) # 查看 'A' 列的头五个元素输出结果:
(因为在第二行代码的时候修改了对象的值,所以再次查看的时候,第A列的前三行都为0)
五、参考文章
1、http://t.csdn.cn/V9fZY
2、http://t.csdn.cn/WJvI4
3、Scanpy数据结构:AnnData - 何帅 - 博客园