nnUNet一个医学图像分割神仙算法
很多年之前接触过UNet的细胞分割,这次突然拿出来做一个三维的腿骨分割,突然有点不知所措了,可能我的技术只局限于二维的吧,经过一个月的研究差不多鼓捣明白了一点,欢迎有经验的大佬赐教一些知识,我这就写写我自己做的过程吧。
一 训练前的准备工作
- 算法框架搭建以及环境配置
必要程序,环境就搞了我好多天,以为虚拟机就够了,结果配置还是不行,果断换成双系统,这个就不写了,网上太多了而且也不难,我以前都是用的虚拟机或者纯ubuntu系统没有装过,网上找了找一天也搞完了。
a. 操作系统:ubuntu18.04
Ubuntu 18.04.6 LTS (Bionic Beaver)
b. NVIDIA驱动:nvidia465.27(对应自己电脑配置在官网查看对应版本下载安装)
NVIDIA 驱动程序下载
c. pytorch:1.8.0 Previous PyTorch Versions | PyTorch
CUDA:11.1
CUDNN:11.1
# CUDA 11.1
conda install pytorch==1.8.0 torchvision==0.9.0 torchaudio==0.8.0 cudatoolkit=11.1 -c pytorch -c conda-forge
d. Annaconda3:python3.6.9
这个在官网下载很慢,下面链接差不多可以达到1MB/s,还是不错的,找到对应的python版本即可,没有非要跟我一样,看你自己用哪个版本但是不要太高也不要太低。
Index of /anaconda/archive/ | 清华大学开源软件镜像站 | Tsinghua Open Source Mirror
- 整理数据:安装nnUNet
第一步:在home下创建nnUNetFrame文件夹,在此文件夹打开终端输入
git clone https://github.com/MIC-DKFZ/nnUNet.git
第二步:进入 nnUNet 文件夹
第三步:终端输入 pip install -e .(如果报错那一定是环境配置不对,版本没有对应上)
第四步:回到上一级,创建一个名为DATASET 的文件夹,保证你的nnUNetFrame文件夹下有两个文件夹,nnUNet 是源码,DATASET就是用来放数据的地方.
第五步:终端进入DATASET,之后创建三个文件夹,nnUNet_raw 放要训练的数据,nnUNet_preprocessed放预处理完的数据,nnUNet_trained_models放训练结果。
第六步:终端进入nnUNet_raw,再创建两个文件夹,nnUNet_raw_data 为原始数据,nnUNet_cropped_data为crop以后的数据。
第七步:进入nnUNet_raw_data,创建一个名为 Task08_HepaticVessel的文件夹前面只是个ID编号,后面是分割项目名字,格式就是这样的,名字和编号可以根据自己项目修改。
第八步:自己的数据集放在上面创建好的文件夹下(也可以是下载好的)。
打开下载好的数据集你会看到,json文件,imagesTr是你的训练数据集,而labelsTr里时对应这个imagesTr的标签文件,同样为nii.gz。训练的时候imageTs文件夹可以先不管
- 设置一下nnUNet读取文件的路径:
a:在home目录下ctrl + h,显示隐藏中的文件,
b:找到.bashrc文件,vim打开,
c:在末尾添加下面三行,保存文件。
export nnUNet_raw_data_base="/home/czc/nnUNetFrame/DATASET/nnUNet_raw"
export nnUNet_preprocessed="/home/czc/nnUNetFrame/DATASET/nnUNet_preprocessed"
export RESULTS_FOLDER="/home/czc/nnUNetFrame/DATASET/nnUNet_trained_models"
d:终端source .bashrc更新一下下。
二,准备开始训练
1.先转换一下数据集
这里我说一下我用自己的数据集训练的顺序,我用的是大腿骨数据,拿到之后有些不是dcm格式结尾的,所以要统一一下。
第一步:先统一改文件后缀为“.dcm”。
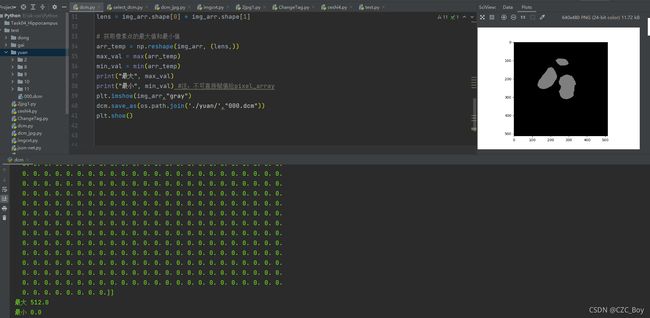
因为我是大腿股骨和胫骨的分割,之后我想修改一下两部分的色差,这样训练完我就可以直接看出哪部分是股骨,哪部分是胫骨,因为原dcm文件内就是二值化的图片,里面只有0个1的值,所以我把dcm中512*512矩阵中的1改小了,颜色确实变成了灰色,和其他没改的0白色可以看出色差,但是保存后依然没有变化,鼓捣了很久没办法,所以……
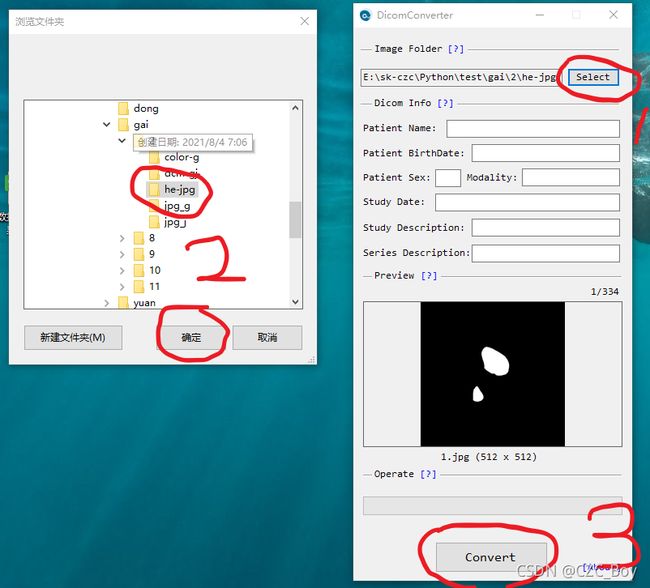
第二步:把dcm格式文件转换为jpg格式。
这样修改会导致原dcm格式中丢失很多信息,不过我就是训练图片,信息没有什么太必要。
第三步:修改股骨或者胫骨颜色另存。
这个修改颜色也就是个修改RBG三通道的值而已。
第四步:把修改好的jpg和未修改的合并。
因为要区分,而且我的股骨和胫骨是两个文件夹,所以就,修改其中一个就行。
第五步:把合并好的jpg转换回dcm。
把两部分文件合并起来,并转换为dcm,不知道怎么转换的,我之前的资源里有。
第六步:添加tag信息到转换回的dcm文件内。
因为之前转换为jpg时候丢失的一些信息,所以我想把一些信息再加进去。
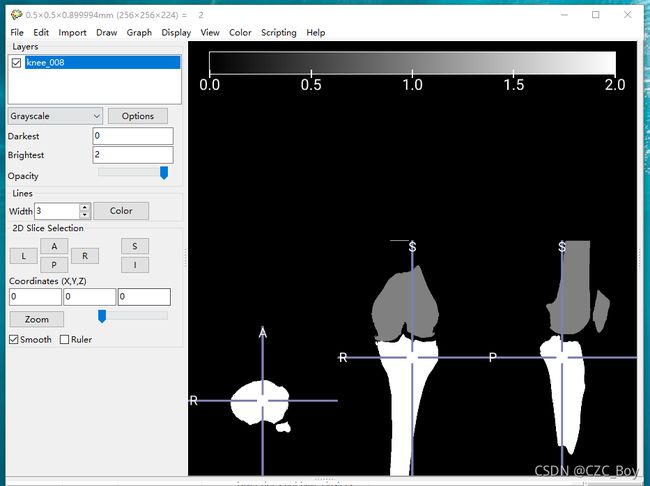
第七步:把dcm文件转换成nii.gz。
同样是找不到怎么转换可以用我资源里的软件,因为训练所需要的必须是nn.gz格式,之后把转换好的dcm文件夹直接拖到MRIcroGL软件内,点击File,在下拉框内点击Save Volume,保存类型选择nii.gz,名字根据自己的需求。
之后使用nnUNet自带的脚本转换一下我们的数据集:
nnUNet_convert_decathlon_task -i /home/czc/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task08_HepaticVessel
之后在Task08_HepaticVessel旁边多了新文件夹Task008_HepaticVessel这就成功了
(如果转换显示未找到命令,运行一下pip install -U setuptools,如果还是不对那就检查一下环境配置是否是版本一致的,不然nnunet安装不上就没有这个默认命令)
数据预处理
nnUNet_plan_and_preprocess -t 8(使用自己的数据集预处理时间会很长,要做各种插值)
如果使用自己的训练集预处理之前最好加上--verify_dataset_integrity检查一下数据内的标签是否正确,因为json里面自己配置的labels中有标签,不对应上是没法开始训练的,会报一个CUDA error: device-side assert triggered的错误
- 开始训练 nnUNet_train 3d_fullres nnUNetTrainerV2 8 4(8为之前数据集的ID,4为五折交叉验证,可以改成0,1,2,3)
- 训练结束
/home/czc/nnUNetFrame/DATASET/nnUNet_trained_models/nnUNet,你会发现这个文件夹下会有一个Task008_HepaticVessel的文件夹,这个就是本次训练模型。
三,开始推理
1.创建推理后的文件夹:
创建一个存放推理结果的inferTs的文件夹。
nnUNet_predict -i /home/czc/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task008_HepaticVessel/imagesTs/ -o /home/czc/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task008_HepaticVessel/inferTs -t 8 -m 3d_fullres -f 4
-t: 后跟着对应的ID;
-f: 后代表使用几折交叉验证;
等待推理结束文件夹内是训练完结果的nn.gz文件,使用app打开即可查看结果。