CT图像获取人体部分掩膜MASK(去除机床)
CT图像获取人体部分掩膜MASK(去除机床)
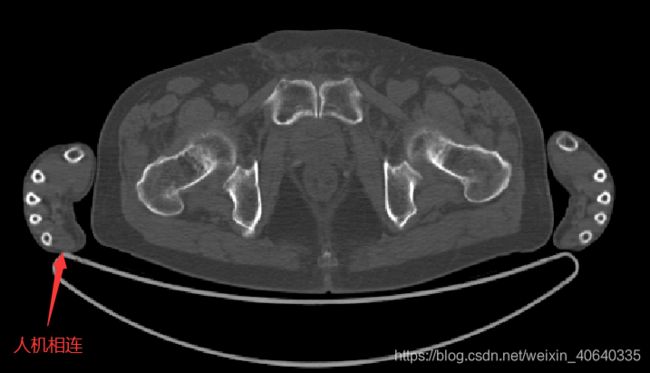
统计CT图像的均值标准差时,希望只针对人体部分进行统计,故需要从CT图像中获取人体部分的掩膜MASK,而人体又经常与机床部分相连,所以也需要去掉机床部分
简单实现方法
理想情况下,通过卡一个较低阈值(-150),能够使人体与机床部分完全分离。这种情况下,直接通过取最大连通域的方式,就可得到人体部分的mask。
代码:
import numpy as np
import os
import SimpleITK as sitk
def getmaxcomponent(mask_array, num_limit=10):
# sitk方法获取连通域
cca = sitk.ConnectedComponentImageFilter()
cca.SetFullyConnected(True)
cca.FullyConnectedOff()
_input = sitk.GetImageFromArray(mask_array.astype(np.uint8))
output_ex = cca.Execute(_input)
labeled_img = sitk.GetArrayFromImage(output_ex)
num = cca.GetObjectCount()
max_label = 0
max_num = 0
# 不必遍历全部连通域,一般在前面就有对应全身mask的label,减少计算时间
for i in range(1, num_limit):
if np.sum(labeled_img == i) < 1e6: # 全身mask的体素数量必然很大,小于设定值的不考虑
continue
if np.sum(labeled_img == i) > max_num:
max_num = np.sum(labeled_img == i)
max_label = i
maxcomponent = np.array((labeled_img == max_label)).astype(np.uint8)
print(str(max_label) + ' num:' + str(max_num)) # 看第几个是最大的
return maxcomponent
def get_body(CT_nii_array):
"""
卡CT阈值获取身体(理想情况下够用了,不过多数情况下会包括到机床部分)
"""
# 阈值二值化,获得最大的3d的连通域
CT_array = np.copy(CT_nii_array)
threshold_all = -200 # 卡的阈值,卡出整个身体以及机床部分
CT_array[CT_array >= threshold_all] = 1
CT_array[CT_array <= threshold_all] = 0
body_mask1 = getmaxcomponent(CT_array, 10)
return body_mask1.astype(np.uint8)
不理想情况的解决方法
不理想的情况指的是,人体与机床有相连,导致取连通域后得到的mask其实包含有机床部分,这种情况在实际中也经常碰到,因此需要去除机床部分,尝试了以下两种方法:
1.形态学方法:
思路:开操作、腐蚀,希望能断开人体与机床部分相连的地方,然后再取一次最大连通域
缺点:该方法在人和机床相连情况不太严重时有一定效果,但是容易导致获得的人体部分经过形态学处理后有点问题
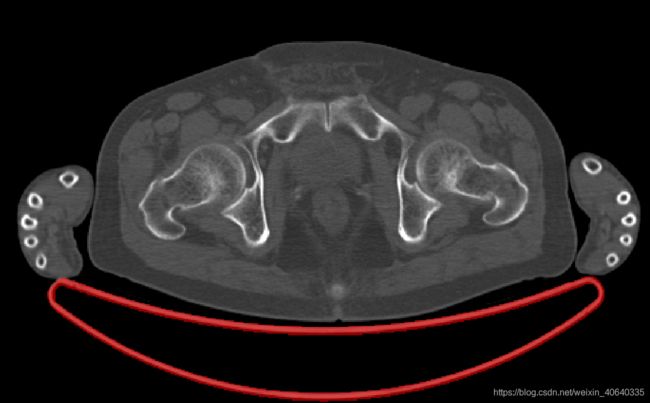
2.先获取机床的mask:
思路:在有多例CT数据的情况下,每例CT图像取到最大连通域(可能包含机床)后,我们叠加所有连通域,由于人体与机床相连的位置各不相同,而机床的位置是固定的,因此相连的地方的重叠次数不高于机床部分,我们保留重叠次数大于设定阈值的区域,再去掉此时的最大连通域(即人体部分),最后经过一下形态学的简单处理,即可获得机床部分的mask
(注意:需要保证这批数据由同样一台机器扫描得到,不然CT不能简单地进行重叠)
获取机床mask的代码(不完整,读取数据部分需要自己写):
body_mask_final = np.zeros([374, 512, 512], dtype=np.uint8)
for i in train_id:
i = str(i)
print('\r'+i, end='')
CT_nii_file = data_folder + sep + i + '_CT.nii.gz'
# SimpleITK读取nii
CT_nii = sitk.ReadImage(CT_nii_file)
CT_a = sitk.GetArrayFromImage(CT_nii)
body_mask = get_body(CT_a)
body_mask_final += body_mask
# 去最大连通域
maxcomponent = getmaxcomponent(machine_mask)
# 去掉最大的身体部分
machine_mask = machine_mask - maxcomponent
# 膨胀3*3,并取取最大连通域
machine_mask_d = dilate_ct(machine_mask, 3, other_axis=False) # 自写的一个函数,用opencv2对每个横截面进行膨胀
machine_mask_s = getmaxcomponent(machine_mask_d)
save_path = r".\machine_mask.nii.gz" # 保存
save_sitk = sitk.GetImageFromArray(machine_mask_s.astype(np.uint8))
sitk.WriteImage(save_sitk, save_path)
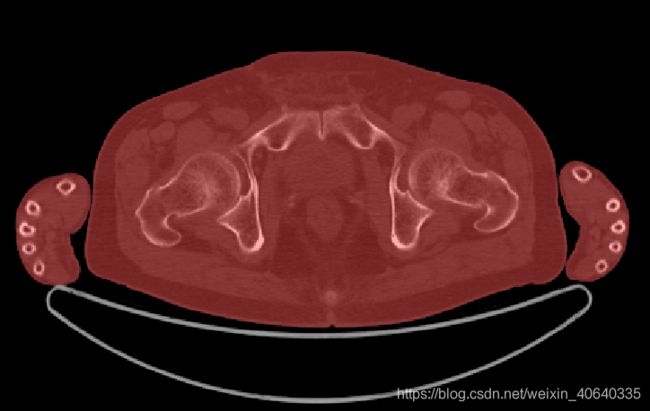
红色部分就是机床mask了,结果还不错,再配合最开始的方法,获得人体的mask
代码:
def cut_machine(CT_nii_array, CT_nii, nii_folder_path, id):
"""
从CT图像中得到去掉机床部分后的人体部分
"""
body_mask_file = nii_folder_path + sep + id + r'_body_mask.nii.gz'
machine_mask_file = r'.\machine_mask.nii.gz' # 机床mask文件
# 阈值二值化,获得最大的3d的连通域
body_mask1 = get_body(CT_nii_array)
# 后处理
# 去掉机床部分
machine_mask = sitk.ReadImage(machine_mask_file)
machine_mask_nii = sitk.GetArrayFromImage(machine_mask)
body_mask2 = body_mask1 - machine_mask_nii
body_mask2[body_mask2 == 255] = 0
body_mask2[body_mask2 < 0] = 0
body_mask3 = getmaxcomponent(body_mask2, 150)
# 填充
body_mask_fill = np.zeros(body_mask3.shape, dtype=np.uint8)
for j in range(body_mask2.shape[0]):
body_mask_fill[j, :, :] = fill_inter_bone(body_mask3[j, :, :])
save_mask = body_mask_fill
save_nii(save_mask, CT_nii, body_mask_file)
# plt.imshow(save_nii[195, :, :])
# plt.show()
return save_mask.astype(np.uint8)
def save_nii(save_nii, CT_nii, save_path, save_mask=True):
"""
保存nii
:param save_nii: 需要保存的nii图像的array
:param CT_nii: 配准的nii图像,用于获取同样的信息
:param save_path: 保存路径
:param save_mask: 保存的是否是mask,默认是True
:return:
"""
if save_mask:
# 保存mask_nii
save_sitk = sitk.GetImageFromArray(save_nii.astype(np.uint8))
else:
# 保存img_nii
save_sitk = sitk.GetImageFromArray(save_nii.astype(np.float))
save_sitk.CopyInformation(CT_nii)
sitk.WriteImage(save_sitk, save_path)
print(save_path + ' processing successfully!')
def fill_inter_bone(mask):
# 对一张图像做孔洞填充,读入的是一层
mask = mask_fill = mask.astype(np.uint8)
if np.sum(mask[:]) != 0: # 即读入图层有值
contours, hierarchy = cv2.findContours(mask, cv2.RETR_TREE, cv2.CHAIN_APPROX_SIMPLE)
len_contour = len(contours)
contour_list = []
for i in range(len_contour):
drawing = np.zeros_like(mask, np.uint8) # create a black image
img_contour = cv2.drawContours(drawing, contours, i, (1, 1, 1), -1)
contour_list.append(img_contour)
mask_fill = sum(contour_list)
mask_fill[mask_fill >= 1] = 1
return mask_fill.astype(np.uint8)
后续想法
针对同一批次数据的情况,其实可以直接取其中一例人体与机床分开的图像单独取得机床mask,后续批量处理中直接取这个mask进行处理即可。大部分情况下是word的。