- 基于python的图像识别_基于PYTHON的图片文字识别
文本资料的图像文件进行分析识别处理,获取文字及版面信息的过程。一般包括以下几个过程:图像输入、图像前处理、预识别:1图像输入:对于不同的图像格式,有着不同的存储格式,不同的压缩方式,目前有OpenCV、CxImage等开源项目。2预处理:主要包括二值化,噪声去除,倾斜较正等。2.1二值化:对摄像头拍摄的图片,大多数是彩色图像,彩色图像所含信息量巨大,对于图片的内容,我们可以简单的分为前景与背景,为
- bug:定时任务因数据库时间滞后导致数据清理失效问题
刘火锅
javaspringcloudspring后端mybatis
问题背景:在数据清理定时任务中,发现理应被删除的数据未被正确清理。经排查发现:定时任务配置在每日00:00:00执行删除数据SQL语句逻辑正常应用服务器时间准确数据库服务器时间比应用服务器慢15秒数据清理SQL使用CURDATE()获取当前日期问题原因分析:当应用服务器在00:00:00触发任务时:应用服务器时间:2025-07-1800:00:00数据库服务器时间:2025-07-1723:59
- 2021-5-11晨间日记
飞翔_8019
今天是什么日子起床:7:05就寝:23:32天气:晴天心情:好纪念日:昨天跟闺蜜聊天一个多小时叫我起床的不是闹钟是梦想年度目标及关键点:财富自由本月重要成果:理财投资学习今日三只青蛙/番茄钟1.读书2.日更文章成功日志-记录三五件有收获的事务1.不能自己欺骗自己,要实事求是能做多少,不能做多少,为什么做不了,要分析原因。不能太迁就自己。2.自己最近做事效率不高。要想办法提高效率。3.规定自己什么时
- 【云原生】Helm来管理Kubernetes集群的详细使用方法与综合应用实战
景天科技苑
云原生K8S零基础到进阶实战云原生kubernetes容器Helmk8sk8s集群
✨✨欢迎大家来到景天科技苑✨✨养成好习惯,先赞后看哦~作者简介:景天科技苑《头衔》:大厂架构师,华为云开发者社区专家博主,阿里云开发者社区专家博主,CSDN全栈领域优质创作者,掘金优秀博主,51CTO博客专家等。《博客》:Python全栈,前后端开发,小程序开发,人工智能,js逆向,App逆向,网络系统安全,数据分析,Django,fastapi,flask等框架,云原生k8s,linux,she
- 李克富 || 培训从“吸吮手指”开始
李克富
昨天上午赶到济南,下午启动仪式后晚上即开启了为武警边防山东总队心理咨询师的培训。这次部队首长明确指示,必须切实解决有关“如何”的问题,比如围绕部队改革中官兵出现的心理波动如何进行心理调试?如何面对改革后可能出现的心理落差?改革后如何制定职业规划?另外,还得通过案例分析和实践演练的方式,教会部队的咨询师们具体的案例处理,诸如婚姻关系问题、亲子关系问题、同事关系问题、突遇家庭变故问题、工作压力大的问题
- 无意识现象
dear心理咨询师黄倩雯
我希望能以少语言尽可能明白的解释无异议整数与在精神分析中意义,并且仅限于这精神分析中的意义。一个出现在意识中的概念和任何其他心理因素,过一会儿就可能消失,再过一段时间,这又可能重新出现。因此我们说他在记忆中并没有发生变化,他的再度出现并不是我们感官重新感觉的结果。为这一点,我们习惯于这样解释假定在一定的时间里纪念只存在于心中,却潜伏在意识里。至于他这样做时采取了什么形式,我们都无法猜测。这是在这点
- 深度学习模型开发部署全流程:以YOLOv11目标检测任务为例
你喜欢喝可乐吗?
deeplearningdeploy深度学习YOLO目标检测
深度学习模型开发部署全流程:以YOLOv11目标检测任务为例深度学习模型从开发到部署的完整流程包含需求分析、数据准备、模型训练、模型优化、模型测试和部署运行六大核心环节。YOLOv11作为新一代目标检测模型,不仅延续了YOLO系列的高效实时性能,还在检测精度和泛化能力上取得显著突破,使其成为工业质检、安防监控、自动驾驶等领域的理想选择。本文将详细阐述这一完整流程,并结合YOLOv11的具体实现,提
- 预测导管原位癌浸润性复发的深度学习:利用组织病理学图像和临床特征
浪漫的诗人
论文深度学习人工智能
文章目录研究内容目的方法数据集模型开发模型训练与评估外部验证统计分析研究结果模型性能风险分层外部验证特征重要性原文链接原文献:Deeplearningforpredictinginvasiverecurrenceofductalcarcinomainsitu:leveraginghistopathologyimagesandclinicalfeatures研究背景【DCIS与IBC的关联】乳腺导管
- 【中项第三版】系统集成项目管理工程师 | 第 7 章 软硬件系统集成
Jackilina_Stone
#系统集成项目管理工程师中项第3版软考中级
前言第7章对应的内容选择题和案例分析都可能会进行考查,学习要以教材为准。目录7.1系统集成基础7.2基础设施集成7.2.1弱电工程7.2.2网络集成1传输子系统2交换子系统3网管子系统4安全子系统5服务子系统7.2.3数据中心集成7.3软件集成7.3.1基础软件集成7.3.2应用软件集成7.3.3其他软件集成7.4业务应用集成7.5本章练习7.1系统集成基础软硬件系统集成是以信息的集成为目标,功能
- Java零基础-数组
全文目录:开篇语前言摘要简介源代码解析应用场景案例优缺点分析类代码方法介绍Java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的一些知识点,并以文字的形式跟大家一起交流,互相学习,一个人虽可以走的更快,但一群人可以走的更远。 我是一名后端开发爱好者,工作
- Java零基础-数组:数组的遍历和操作
喵手
零基础学Javajava算法
全文目录:开篇语前言摘要简介源代码解析应用场景案例优缺点分析类代码方法介绍Java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的一些知识点,并以文字的形式跟大家一起交流,互相学习,一个人虽可以走的更快,但一群人可以走的更远。 我是一名后端开发爱好者,工作
- Java零基础-数组:多维数组
喵手
零基础学Javajava算法
全文目录:开篇语前言摘要简介源代码解析应用场景案例优缺点分析类代码方法介绍具体的Java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的一些知识点,并以文字的形式跟大家一起交流,互相学习,一个人虽可以走的更快,但一群人可以走的更远。 我是一名后端开发爱好者
- Java零基础-数组:数组长度和属性
喵手
零基础学Javajava开发语言
全文目录:开篇语前言摘要简介源代码解析应用场景案例优缺点分析类代码方法介绍Java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的一些知识点,并以文字的形式跟大家一起交流,互相学习,一个人虽可以走的更快,但一群人可以走的更远。 我是一名后端开发爱好者,工作
- Java零基础-数组:声明和初始化数组
喵手
零基础学Javajava开发语言
全文目录:开篇语前言摘要简介静态初始化动态初始化源代码解析应用场景案例优缺点分析类代码方法介绍java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的一些知识点,并以文字的形式跟大家一起交流,互相学习,一个人虽可以走的更快,但一群人可以走的更远。 我是一名
- Java零基础-数组:访问和修改数组元素
全文目录:开篇语前言摘要简介源代码解析数组的定义和初始化访问数组元素修改数组元素应用场景案例优缺点分析优点缺点类代码方法介绍int[]getArray()voidprintArray(int[]arr)Java代码测试用例全文小结总结文末开篇语哈喽,各位小伙伴们,你们好呀,我是喵手。运营社区:C站/掘金/腾讯云/阿里云/华为云/51CTO;欢迎大家常来逛逛 今天我要给大家分享一些自己日常学习到的
- 二手苹果手机和全新安卓手机哪个更值得买?
你若三冬来回
不知道大家有没有注意过这么一个现象,那就是有很多人宁愿去购买一部二手iphone手机,也不愿花同样的价钱去买一款最新的安卓旗舰?今天我们就来分析一下二手苹果手机和全新安卓手机哪个更值得买。1.流畅度虽然现如今国产手机越做越好,经常打广告说什么两年不卡、三年不卡什么的,但事实上真的能做到这一点品牌其实并不多,而相反,苹果则在这方面做得挺不错,iOS系统从流畅度、交互体验、用户体验等方面上都要远超安卓
- 暑期自学嵌入式——Day02(C语言阶段)
一位搞嵌入式的 genius
嵌入式自学专栏linux嵌入式C语言
点关注不迷路哟。你的点赞、收藏,一键三连,是我持续更新的动力哟!!!主页:一位搞嵌入式的genius-CSDN博客https://blog.csdn.net/m0_73589512?spm=1000.2115.3001.5343目录Day02→数据类型(上)数据类型分类基本数据类型整形数据类型字符型数据类型实型数据类型构造数据类型特殊数据类型布尔类型详解基本概念使用注意事项预处理分析知识小结Day
- 安全事件响应分析--基础命令
大大的大大
ctflinuxweb安全
----万能密码'or'='or''1or#1'or'1'='11or1=1=============安全事件响应分析==========------***windoes***------方法开机启动有无异常文件【开始】➜【运行】➜【msconfig】文件排查各个盘下的temp(tmp)相关目录下查看有无异常文件:Windows产生的临时文件可以通过查看日志且通过筛选根据文件夹内文件列表时间进行排
- 关于 Excel 学习和数据分析有什么书籍或网站推荐?
cda2024
excel学习数据分析
在当今信息爆炸的时代,Excel和数据分析已经成为职场人士必备的技能。无论是处理日常工作中的数据、制作报表,还是进行更深入的数据分析,Excel都是一个不可或缺的工具。那么,如何系统地学习Excel和数据分析呢?有哪些书籍或网站能够帮助我们快速上手并掌握这些技能?今天,我们就来探讨一下关于Excel学习和数据分析的书籍与网站推荐。一、Excel学习入门(一)书籍推荐《Excel数据分析与应用实战》
- 运维打铁: Ruby 脚本在运维自动化中的应用探索
懂搬砖
运维打铁原力计划运维ruby自动化
文章目录一、思维导图二、基础介绍1.Ruby语言特点2.运维自动化概念三、应用场景1.服务器配置管理2.定时任务执行3.日志分析处理四、代码示例1.服务器配置脚本2.定时任务脚本3.日志分析脚本五、优势与挑战1.优势2.挑战六、总结与展望一、思维导图Ruby脚本在运维自动化中的应用基础介绍应用场景代码示例优势与挑战总结与展望Ruby语言特点运维自动化概念服务器配置管理定时任务执行日志分析处理服务器
- 何太极:互联网中服务费的商业价值
太极专栏
有一种财富之门,互联网企业绝不轻易开启,一旦打开,就是金山!这是什么呢?那就是服务费!多少互联网企业,成也服务费,败也服务费!一、服务费是互联网掘金的重要秘诀?所谓服务费,就是是对合作者、使用者入驻平台或使用平台服务所收取的使用费。在太极的《大话互联网模式》一书中,对服务费与其他费用进行了对比和分析。1.服务费与会员费服务费有时也是一种会员费,但服务内容如果不形成套餐或会员制,以独立或明细项目方式
- R语言金融工程:量化价值投资中的数据处理技巧
量化价值投资入门到精通
r语言金融开发语言ai
R语言金融工程:量化价值投资中的数据处理技巧关键词:R语言、金融工程、量化价值投资、数据处理、财务指标、时间序列、风险控制摘要:在量化价值投资领域,高质量的数据处理是策略有效性的核心基础。本文系统解析基于R语言的金融数据处理全流程,涵盖数据获取、清洗、特征工程、时间序列分析等关键环节。通过财务指标计算、异常值检测、缺失值处理、因子标准化等实用技巧,结合quantmod、TTR、dplyr等R包的深
- aocache:AOCache 新增功能深度解析:从性能监控到灵活配置的全方位升级
10km
aocachejavajavaaocache性能分析
最近对aocache进行了重要升级,最新版本0.6.0增加了几项新功能:性能分析日志,AOCache性能分析工具,切入点自定义配置,全局配置,本文详细说明这几项目新功能的作用和使用方式。一、性能分析日志需求背景对于哪些方法适合使用aocache注解提高性能,开始我是凭经验和直觉来判断的。但是对于下面这个方法,凭直觉你觉得用方法缓存能提高性能么?publicStringnext(Stringexpr
- Python日志终极指南:深入探索logging日志管理模块
c01dkit
pythonpython开发语言
在任何一个严谨的软件开发项目中,日志(Logging)都是不可或缺的一环。它不仅是调试代码的利器,更是线上问题追踪、性能分析和数据监控的重要依据。相比于随处可见的print()语句,Python内置的logging模块提供了更为强大、灵活且标准化的解决方案。[1][2]这篇博客将带你由浅入深,全面掌握logging模块的使用,从基础配置到高级技巧,再到企业级项目的最佳实践。一、告别print():
- 【Python爬虫(26)】Python爬虫进阶:数据清洗与预处理的魔法秘籍
奔跑吧邓邓子
Python爬虫python爬虫开发语言数据清洗预处理
【Python爬虫】专栏简介:本专栏是Python爬虫领域的集大成之作,共100章节。从Python基础语法、爬虫入门知识讲起,深入探讨反爬虫、多线程、分布式等进阶技术。以大量实例为支撑,覆盖网页、图片、音频等各类数据爬取,还涉及数据处理与分析。无论是新手小白还是进阶开发者,都能从中汲取知识,助力掌握爬虫核心技能,开拓技术视野。目录一、数据清洗的重要性二、数据清洗的常见任务2.1去除噪声数据2.2
- 为什么朋友越来越少?
东心子
越长大朋友越少。试图分析过原因,除了性格越来越趋于被动外,更多的是对血脉亲情之外的其他关系抱有天然的不自信态度。因为深知建立并维系好一段关系是需要耗费大量时间和精力的,而随着年岁的渐长人在诸多不确定面前也变得越发谨慎和小心了。不敢主动靠近,更怕无意义的投入,只想活在自己的舒服区和安全区,想把珍贵的时间和精力用在天然更易亲近的关系上。这好像是件可悲的事,因为人变得越来越现实了。但又好像是件必然的事,
- C#-Linq源码解析之Concat
黑哥聊dotNet
DotNet-Linq详解linqc#
前言在Dotnet开发过程中,Concat作为IEnumerable的扩展方法,十分常用。本文对Concat方法的关键源码进行简要分析,以方便大家日后更好的使用该方法。使用Concat连接两个序列。假如我们有这样的两个集合,我们需要把两个集合进行连接!List lst = new List { "张三", "李四" };List lst2 = new List { "王麻子" };不使用Linq大
- Redis性能测试:工具、参数与实战示例
Seal^_^
数据库专栏#数据库--Redisredis数据库Redis性能测试
Redis性能测试:工具、参数与实战示例1.Redis性能测试概述2.redis-benchmark基础使用2.1基本语法2.2简单示例3.性能测试参数详解4.实战测试示例4.1基础测试4.2指定命令测试4.3带随机key的测试4.4大数据测试4.5管道测试5.性能测试流程图6.测试结果分析与优化建议6.1结果解读6.2优化建议7.高级测试场景7.1持久化影响测试7.2集群测试7.3长时间稳定性测
- C# Linq源码解析之Aggregate
黑哥聊dotNet
DotNet-Linq详解c#linqlist
前言在Dotnet开发过程中,Aggregate作为IEnumerable的扩展方法,十分常用。本文对Aggregate方法的关键源码进行简要分析,以方便大家日后更好的使用该方法。使用Aggregate是对序列应用累加器的函数。看下面一段代码:List lst = new List() { "张三", "李四", "王麻子" };给了我们这样的一个list集合,我们想要得到"张三哈哈哈李四哈哈哈王
- 【实操】信息安全工程师系列-第22关 网站安全需求分析与安全保护工程
披荆斩棘的GG
安全
【实操】信息安全工程师系列-第22关网站安全需求分析与安全保护工程********永远不要信任用户输入。—安全编程格言一、网站安全基础概念与威胁分(一)核心定义**网站安全目标:**保障机密性(数据不泄露)、完整性(数据不被篡改)、可用性(服务不中断)和可控性(管理可控制)。**技术架构:**基于B/S架构,涉及网络通信、操作系统、数据库、Web服务器(如Apache、IIS)、Web应用及相关协
- HQL之投影查询
归来朝歌
HQLHibernate查询语句投影查询
在HQL查询中,常常面临这样一个场景,对于多表查询,是要将一个表的对象查出来还是要只需要每个表中的几个字段,最后放在一起显示?
针对上面的场景,如果需要将一个对象查出来:
HQL语句写“from 对象”即可
Session session = HibernateUtil.openSession();
- Spring整合redis
bylijinnan
redis
pom.xml
<dependencies>
<!-- Spring Data - Redis Library -->
<dependency>
<groupId>org.springframework.data</groupId>
<artifactId>spring-data-redi
- org.hibernate.NonUniqueResultException: query did not return a unique result: 2
0624chenhong
Hibernate
参考:http://blog.csdn.net/qingfeilee/article/details/7052736
org.hibernate.NonUniqueResultException: query did not return a unique result: 2
在项目中出现了org.hiber
- android动画效果
不懂事的小屁孩
android动画
前几天弄alertdialog和popupwindow的时候,用到了android的动画效果,今天专门研究了一下关于android的动画效果,列出来,方便以后使用。
Android 平台提供了两类动画。 一类是Tween动画,就是对场景里的对象不断的进行图像变化来产生动画效果(旋转、平移、放缩和渐变)。
第二类就是 Frame动画,即顺序的播放事先做好的图像,与gif图片原理类似。
- js delete 删除机理以及它的内存泄露问题的解决方案
换个号韩国红果果
JavaScript
delete删除属性时只是解除了属性与对象的绑定,故当属性值为一个对象时,删除时会造成内存泄露 (其实还未删除)
举例:
var person={name:{firstname:'bob'}}
var p=person.name
delete person.name
p.firstname -->'bob'
// 依然可以访问p.firstname,存在内存泄露
- Oracle将零干预分析加入网络即服务计划
蓝儿唯美
oracle
由Oracle通信技术部门主导的演示项目并没有在本月较早前法国南斯举行的行业集团TM论坛大会中获得嘉奖。但是,Oracle通信官员解雇致力于打造一个支持零干预分配和编制功能的网络即服务(NaaS)平台,帮助企业以更灵活和更适合云的方式实现通信服务提供商(CSP)的连接产品。这个Oracle主导的项目属于TM Forum Live!活动上展示的Catalyst计划的19个项目之一。Catalyst计
- spring学习——springmvc(二)
a-john
springMVC
Spring MVC提供了非常方便的文件上传功能。
1,配置Spring支持文件上传:
DispatcherServlet本身并不知道如何处理multipart的表单数据,需要一个multipart解析器把POST请求的multipart数据中抽取出来,这样DispatcherServlet就能将其传递给我们的控制器了。为了在Spring中注册multipart解析器,需要声明一个实现了Mul
- POJ-2828-Buy Tickets
aijuans
ACM_POJ
POJ-2828-Buy Tickets
http://poj.org/problem?id=2828
线段树,逆序插入
#include<iostream>#include<cstdio>#include<cstring>#include<cstdlib>using namespace std;#define N 200010struct
- Java Ant build.xml详解
asia007
build.xml
1,什么是antant是构建工具2,什么是构建概念到处可查到,形象来说,你要把代码从某个地方拿来,编译,再拷贝到某个地方去等等操作,当然不仅与此,但是主要用来干这个3,ant的好处跨平台 --因为ant是使用java实现的,所以它跨平台使用简单--与ant的兄弟make比起来语法清晰--同样是和make相比功能强大--ant能做的事情很多,可能你用了很久,你仍然不知道它能有
- android按钮监听器的四种技术
百合不是茶
androidxml配置监听器实现接口
android开发中经常会用到各种各样的监听器,android监听器的写法与java又有不同的地方;
1,activity中使用内部类实现接口 ,创建内部类实例 使用add方法 与java类似
创建监听器的实例
myLis lis = new myLis();
使用add方法给按钮添加监听器
- 软件架构师不等同于资深程序员
bijian1013
程序员架构师架构设计
本文的作者Armel Nene是ETAPIX Global公司的首席架构师,他居住在伦敦,他参与过的开源项目包括 Apache Lucene,,Apache Nutch, Liferay 和 Pentaho等。
如今很多的公司
- TeamForge Wiki Syntax & CollabNet User Information Center
sunjing
TeamForgeHow doAttachementAnchorWiki Syntax
the CollabNet user information center http://help.collab.net/
How do I create a new Wiki page?
A CollabNet TeamForge project can have any number of Wiki pages. All Wiki pages are linked, and
- 【Redis四】Redis数据类型
bit1129
redis
概述
Redis是一个高性能的数据结构服务器,称之为数据结构服务器的原因是,它提供了丰富的数据类型以满足不同的应用场景,本文对Redis的数据类型以及对这些类型可能的操作进行总结。
Redis常用的数据类型包括string、set、list、hash以及sorted set.Redis本身是K/V系统,这里的数据类型指的是value的类型,而不是key的类型,key的类型只有一种即string
- SSH2整合-附源码
白糖_
eclipsespringtomcatHibernateGoogle
今天用eclipse终于整合出了struts2+hibernate+spring框架。
我创建的是tomcat项目,需要有tomcat插件。导入项目以后,鼠标右键选择属性,然后再找到“tomcat”项,勾选一下“Is a tomcat project”即可。具体方法见源码里的jsp图片,sql也在源码里。
补充1:项目中部分jar包不是最新版的,可能导
- [转]开源项目代码的学习方法
braveCS
学习方法
转自:
http://blog.sina.com.cn/s/blog_693458530100lk5m.html
http://www.cnblogs.com/west-link/archive/2011/06/07/2074466.html
1)阅读features。以此来搞清楚该项目有哪些特性2)思考。想想如果自己来做有这些features的项目该如何构架3)下载并安装d
- 编程之美-子数组的最大和(二维)
bylijinnan
编程之美
package beautyOfCoding;
import java.util.Arrays;
import java.util.Random;
public class MaxSubArraySum2 {
/**
* 编程之美 子数组之和的最大值(二维)
*/
private static final int ROW = 5;
private stat
- 读书笔记-3
chengxuyuancsdn
jquery笔记resultMap配置ibatis一对多配置
1、resultMap配置
2、ibatis一对多配置
3、jquery笔记
1、resultMap配置
当<select resultMap="topic_data">
<resultMap id="topic_data">必须一一对应。
(1)<resultMap class="tblTopic&q
- [物理与天文]物理学新进展
comsci
如果我们必须获得某种地球上没有的矿石,才能够进行某些能量输出装置的设计和建造,而要获得这种矿石,又必须首先进行深空探测,而要进行深空探测,又必须获得这种能量输出装置,这个矛盾的循环,会导致地球联盟在与宇宙文明建立关系的时候,陷入困境
怎么办呢?
- Oracle 11g新特性:Automatic Diagnostic Repository
daizj
oracleADR
Oracle Database 11g的FDI(Fault Diagnosability Infrastructure)是自动化诊断方面的又一增强。
FDI的一个关键组件是自动诊断库(Automatic Diagnostic Repository-ADR)。
在oracle 11g中,alert文件的信息是以xml的文件格式存在的,另外提供了普通文本格式的alert文件。
这两份log文
- 简单排序:选择排序
dieslrae
选择排序
public void selectSort(int[] array){
int select;
for(int i=0;i<array.length;i++){
select = i;
for(int k=i+1;k<array.leng
- C语言学习六指针的经典程序,互换两个数字
dcj3sjt126com
c
示例程序,swap_1和swap_2都是错误的,推理从1开始推到2,2没完成,推到3就完成了
# include <stdio.h>
void swap_1(int, int);
void swap_2(int *, int *);
void swap_3(int *, int *);
int main(void)
{
int a = 3;
int b =
- php 5.4中php-fpm 的重启、终止操作命令
dcj3sjt126com
PHP
php 5.4中php-fpm 的重启、终止操作命令:
查看php运行目录命令:which php/usr/bin/php
查看php-fpm进程数:ps aux | grep -c php-fpm
查看运行内存/usr/bin/php -i|grep mem
重启php-fpm/etc/init.d/php-fpm restart
在phpinfo()输出内容可以看到php
- 线程同步工具类
shuizhaosi888
同步工具类
同步工具类包括信号量(Semaphore)、栅栏(barrier)、闭锁(CountDownLatch)
闭锁(CountDownLatch)
public class RunMain {
public long timeTasks(int nThreads, final Runnable task) throws InterruptedException {
fin
- bleeding edge是什么意思
haojinghua
DI
不止一次,看到很多讲技术的文章里面出现过这个词语。今天终于弄懂了——通过朋友给的浏览软件,上了wiki。
我再一次感到,没有辞典能像WiKi一样,给出这样体贴人心、一清二楚的解释了。为了表达我对WiKi的喜爱,只好在此一一中英对照,给大家上次课。
In computer science, bleeding edge is a term that
- c中实现utf8和gbk的互转
jimmee
ciconvutf8&gbk编码
#include <iconv.h>
#include <stdlib.h>
#include <stdio.h>
#include <unistd.h>
#include <fcntl.h>
#include <string.h>
#include <sys/stat.h>
int code_c
- 大型分布式网站架构设计与实践
lilin530
应用服务器搜索引擎
1.大型网站软件系统的特点?
a.高并发,大流量。
b.高可用。
c.海量数据。
d.用户分布广泛,网络情况复杂。
e.安全环境恶劣。
f.需求快速变更,发布频繁。
g.渐进式发展。
2.大型网站架构演化发展历程?
a.初始阶段的网站架构。
应用程序,数据库,文件等所有的资源都在一台服务器上。
b.应用服务器和数据服务器分离。
c.使用缓存改善网站性能。
d.使用应用
- 在代码中获取Android theme中的attr属性值
OliveExcel
androidtheme
Android的Theme是由各种attr组合而成, 每个attr对应了这个属性的一个引用, 这个引用又可以是各种东西.
在某些情况下, 我们需要获取非自定义的主题下某个属性的内容 (比如拿到系统默认的配色colorAccent), 操作方式举例一则:
int defaultColor = 0xFF000000;
int[] attrsArray = { andorid.r.
- 基于Zookeeper的分布式共享锁
roadrunners
zookeeper分布式共享锁
首先,说说我们的场景,订单服务是做成集群的,当两个以上结点同时收到一个相同订单的创建指令,这时并发就产生了,系统就会重复创建订单。等等......场景。这时,分布式共享锁就闪亮登场了。
共享锁在同一个进程中是很容易实现的,但在跨进程或者在不同Server之间就不好实现了。Zookeeper就很容易实现。具体的实现原理官网和其它网站也有翻译,这里就不在赘述了。
官
- 两个容易被忽略的MySQL知识
tomcat_oracle
mysql
1、varchar(5)可以存储多少个汉字,多少个字母数字? 相信有好多人应该跟我一样,对这个已经很熟悉了,根据经验我们能很快的做出决定,比如说用varchar(200)去存储url等等,但是,即使你用了很多次也很熟悉了,也有可能对上面的问题做出错误的回答。 这个问题我查了好多资料,有的人说是可以存储5个字符,2.5个汉字(每个汉字占用两个字节的话),有的人说这个要区分版本,5.0
- zoj 3827 Information Entropy(水题)
阿尔萨斯
format
题目链接:zoj 3827 Information Entropy
题目大意:三种底,计算和。
解题思路:调用库函数就可以直接算了,不过要注意Pi = 0的时候,不过它题目里居然也讲了。。。limp→0+plogb(p)=0,因为p是logp的高阶。
#include <cstdio>
#include <cstring>
#include <cmath&

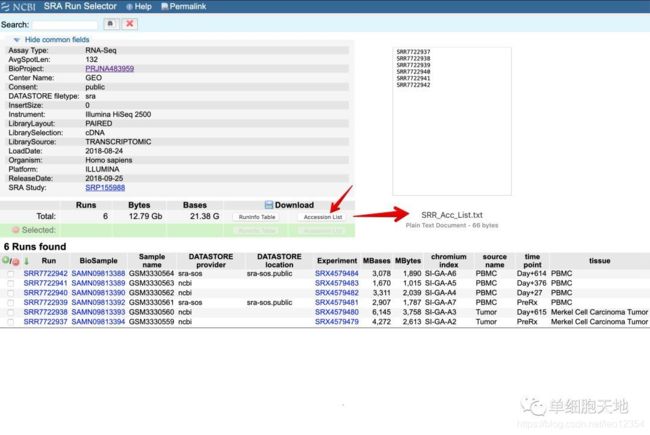
 或者直接去SRA selector 网址,写上咱们所需要的SRR号。
或者直接去SRA selector 网址,写上咱们所需要的SRR号。