Gromacs副本交换分子动力学模拟(REMD)
REMD(副本交换分子动力学)是一种增强采样方法,其在不同温度下对具有相似势能的体系进行采样。通过这种方法,可以增加体系跳出势能面势阱的可能性,从而达到探索新的构象空间的目的。
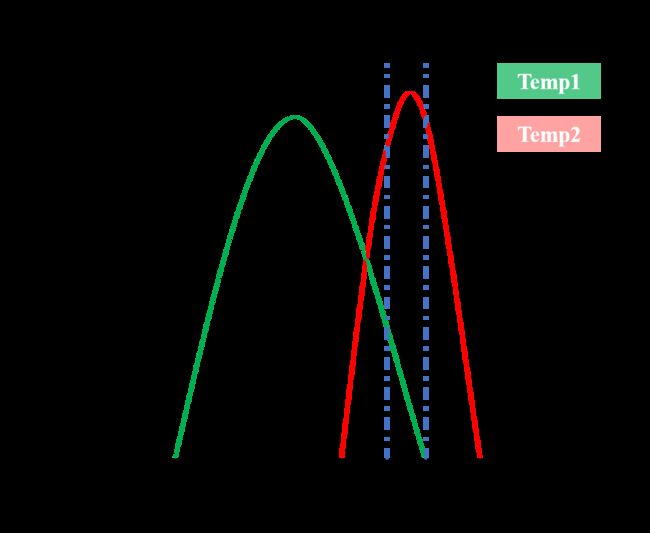
一般来说某一温度下蛋白模拟构象分布满足正态分布规律。如上图所示,展示了同一体系在不同温度下的分布情况,横坐标表示模拟过程中出现的构象,纵坐标表示某一构象存在的丰度。对于体系1(绿色)而言,能达到虚线区域内构象的概率比较小,需要延长模拟时间才能提高这个区域构象出现的次数,若模拟时间不充分甚至有可能采样不到该区域的构象。而对于体系2(红色)而言,却很容易达到蓝色虚线之间的构象,如果在模拟过程中体系1与体系2在这个区间进行了构象交换,那么就可以弥补体系1采样不足的问题。
以上个人愚见,仅供参考!
Gromacs 中实现的方式:
注意:只有单线程 gromacs 不支持 REMD,需要多线程编译版本。安装过程和之前的教程相差无几,只是需要额外安装 openmpi,并在最后 gromacs 编译步骤增加一个选项-DGMX_MPI=ON 即可。编译完成并安装后,执行gmx_mpi可以正常调用。
- 确定所选模拟体系温度变化范围,温度变化梯度;
- 每一个温度下分别进行预平衡(NVT/NPT),每个温度下的预平衡称为一个副本/或系综。在这些副本进行预平衡之前共同都要进行相同的步骤:包括建模、能量最小化。
- 以每个副本预平衡后的终构象作为起始分别开始对应温度下的成品模拟。
实现上述过程并不复杂,我为大家提供了运行脚本:
关注微信公众号grosetta,后台回复“REMD”自取(以溶菌酶模拟为例)
1、模型构建+EM
cd REMD
chmod 777 run.sh
./run.sh
运行完后,在主文件夹中会出现模拟后的文件(后面不需要的都自动删除了),文件1EM.gro及topol.top是必须存在的。
用编辑器打开1EM.gro及topol.top查看蛋白总原子数及水总分子数。
2、副本温度取值确定
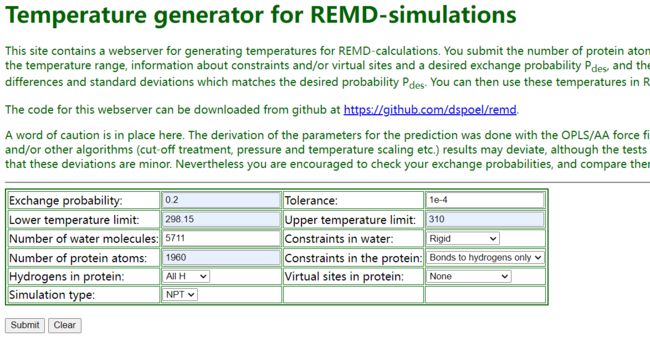
打开网址:
http://virtualchemistry.org/remd-temperature-generator/
其中:
- Exchange probability(副本交换频率)可根据需求自行调整;
- Lower temperature limit 设定温度区间下限(单位K);
- Number of water molecules 体系总的水分子数;
- Number of protein atoms 体系蛋白总原子数;
- Upper temperature limit 设定温度区间上限(单位K);
- Constraints in water 水分子模型是柔性水还是刚性水,一般选刚性水;
- Constraints in the protein 选择只对氢原子的键进行束缚;
其余各项保持默认即可。
点击submit提交,返回程序推荐的各副本模拟温度取值(需要注意的是该程序针对oplsaa力场发展而来,对于应用其他力场的体系,如AMBER、GROMOS,则多少存在些偏差)。
复制红框内的数据以备后用。
3、多副本预平衡模拟
在REMD文件夹一级目录下打开终端
chmod 777 step1.sh
./step1.sh
step1.sh脚本内容如下(第一行的温度数据就是上一步复制结果,替换成你的即可):
#!/bin/bash
T="298.15, 299.47, 300.78, 302.11, 303.44, 304.77, 306.11, 307.47, 308.82, 310.00"
arr_T=(${T//,/ }) #温度数据转为数组
len=${#arr_T[@]} #统计数组元素个数
rm -r ./step1/equ* > /dev/null 2>&1 #删除step1路径下的残存文件夹
for ((i=0; i<=$len-1; i++))
do
mkdir ./step1/equ_${i}
cp ./step1/mdp_file/* ./step1/equ_${i}/.
sed -i 's/TEMP/'${arr_T[$i]}'/g' ./step1/equ_${i}/*.mdp
#预平衡
gmx_mpi grompp -f ./step1/equ_${i}/PR.mdp -c 1EM.gro -r 1EM.gro -p topol.top -o ./step1/equ_${i}/2PR.tpr -maxwarn 99
gmx_mpi mdrun -deffnm ./step1/equ_${i}/2PR -v
#NVT
gmx_mpi grompp -f ./step1/equ_${i}/NVT.mdp -c ./step1/equ_${i}/2PR.gro -p topol.top -o ./step1/equ_${i}/3NVT.tpr -maxwarn 99
gmx_mpi mdrun -deffnm ./step1/equ_${i}/3NVT -v
#NPT
gmx_mpi grompp -f ./step1/equ_${i}/NPT.mdp -c ./step1/equ_${i}/3NVT.gro -p topol.top -o ./step1/equ_${i}/4NPT.tpr -maxwarn 99
gmx_mpi mdrun -deffnm ./step1/equ_${i}/4NPT -v
#删除中间文件
rm -f ./step1/equ_${i}/*.trr ./step1/equ_${i}/*.edr ./step1/equ_${i}/*tpr ./step1/equ_${i}/*.log ./step1/equ_${i}/*.xtc ./step1/equ_${i}/\#* > /dev/null 2>&1
done
此步的mdp模板文件存在路径为step1/mdp_file,可以根据自己需求修改步长、总步数、输出频率等各类参数(注意和温度数值相关参数一概不要修改,脚本可代劳。此外该路径下的模板文件不能删除)。
4、成品模拟(副本交换)
同样是在REMD文件夹一级目录下打开终端
chmod 777 step2.sh
./step2.sh
step2.sh脚本内容如下(同样只需要替换第一行的温度数据,和上一步一样):
#!/bin/bash
T="298.15, 299.47, 300.78, 302.11, 303.44, 304.77, 306.11, 307.47, 308.82, 310.00"
arr_T=(${T//,/ }) #温度数据转为数组
len=${#arr_T[@]} #统计数组元素个数
mid=${len}-1
rm -r ./step2/MD* > /dev/null 2>&1 #删除step2路径下的残存文件夹
for ((i=0; i<=$mid; i++))
do
mkdir ./step2/MD_${i}
cp ./step2/mdp_file/* ./step2/MD_${i}/.
cp ./step1/equ_${i}/4*.gro ./step2/MD_${i}/.
sed -i 's/TEMP/'${arr_T[$i]}'/g' ./step2/MD_${i}/*.mdp
#成品模拟tpr文件生成
gmx_mpi grompp -f ./step2/MD_${i}/md.mdp -c ./step1/equ_${i}/4NPT.gro -p topol.top -o ./step2/MD_${i}/5MD.tpr
done
执行交换模拟
mpirun -np 8 gmx_mpi mdrun -deffnm 5MD -multidir ./step2/MD_[12345678] -replex 1000
- -np 表示调用cpu核心数
- [] 内的数字表示副本所在文件夹名称的后缀数字,我们创建了10个副本,对应文件夹的后缀名是0,1,2,3,4,5,6,7,8,9 但由于cpu个数应该是副本个数整数倍,我是在笔记本上的wsl中进行的测试,只有八个核心,所以最多只能进行8个副本的交换,我选的这八个副本的文件夹后缀序号是1-8,对应在中括号内需要将数字补齐,即12345678
- replex 表示副本模拟多少步进行一次交换,蛋白体系一般需要至少间隔1ps(在步长为2fs情况下,即运行500步)再交换。
由于进行模拟的每个副本在模拟过程中进行了构象交换,所以各自在时间上就不是连续的,可以用gromacs自带脚本进行整合,命令如下:
demux.pl ./step2/MD_1/5MD.log
gmx_mpi trjcat -f ./step2/MD_[12345678]/5MD.xtc -demux replica_index.xvg -o ./traj_merge/{1..8}_traj.xtc
最后整合好的轨迹输出到了traj_merge文件夹内。
整合后的轨迹只代表单一温度下的轨迹