RNA-seq 详细教程:详解DESeq2流程(9)
学习目标
- 了解
DESeq2涉及的不同步骤 - 了解变异的来源并检查
size factors - 检查基因水平的离散估计
- 了解差异表达分析过程中离散的重要性
DESeq2流程
前面,我们使用设计公式创建了 DESeq2 对象,并使用下面两行代码运行DESeq2:
dds <- DESeqDataSetFromTximport(txi, colData = meta, design = ~ sampletype)
dds <- DESeq(dds)
我们用 DESeq2 完成了差异基因表达分析的整个工作流程。分析中的步骤如下:
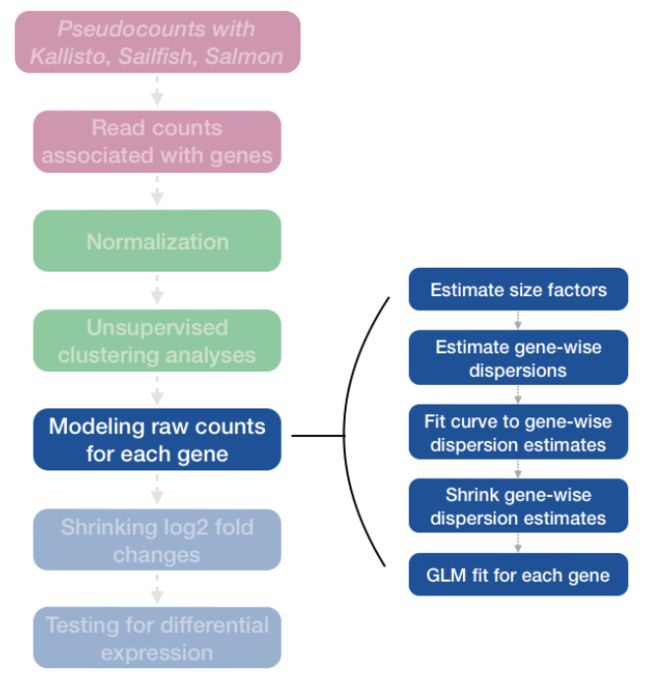 workflow
workflow
我们将详细了解这些步骤中的每一个,以更好地了解 DESeq2 如何执行统计分析,以及我们应该检查哪些指标来验证我们的分析质量。
1. size factors
差异表达分析的第一步是估计大小因子,这正是我们已经对原始计数进行归一化所做的。
 Estimate size factors
Estimate size factors
DESeq2 在执行差异表达分析时会自动估计大小因子。但是,如果您已经像我们之前所做的那样使用 estimateSizeFactors() 生成了大小因子,那么 DESeq2 将使用这些值。
为了归一化计数数据,DESeq2 使用前面教程中讨论的比率中值方法计算每个样本的大小因子。
-
MOV10 DE分析:检查size factors
让我们浏览一下每个样本的大小因子值:
# Check the size factors
sizeFactors(dds)
Irrel_kd_1 Irrel_kd_2 Irrel_kd_3 Mov10_kd_2 Mov10_kd_3 Mov10_oe_1 Mov10_oe_2
1.1149694 0.9606733 0.7492240 1.5633640 0.9359695 1.2262649 1.1405026
Mov10_oe_3
0.6542030
这些数字应该与我们最初运行函数 estimateSizeFactors(dds) 时生成的数字相同。查看每个样本的总读取数:
# Total number of raw counts per sample
colSums(counts(dds))
- 这些数字如何与尺寸因子相关联?
我们看到较大的大小因子对应于具有较高测序深度的样本,这是有道理的,因为要生成我们的归一化计数,我们需要将计数除以大小因子。这解释了样本之间测序深度的差异。
现在看看归一化后的总深度:
# Total number of normalized counts per sample
colSums(counts(dds, normalized=T))
- 样本间的值与每个样本的总计数相比如何?
您可能期望归一化后样本中的计数完全相同。然而,DESeq2 还在归一化过程中考虑了 RNA 组成。通过使用大小因子的中值比值,DESeq2 不应偏向于被少数 DE 基因吸收的大量计数;然而,这可能导致大小因素与仅基于测序深度的预期大不相同。
2. gene-wise dispersion
差异表达分析的下一步是估计基因差异。在我们进入细节之前,我们应该对 DESeq2 中的离散指的是什么有一个很好的了解。
 gene-wise dispersion
gene-wise dispersion
在 RNA-seq 计数数据中,我们知道:
- 为了确定差异表达的基因,我们评估组间表达的变化并将其与组内(重复之间)的变化进行比较。
- 对于每个单独的基因,均值不等于方差。 高表达的基因将具有更一致的变异水平,但会高于平均值。 低表达的基因将表现出徘徊在平均值附近的变异(但具有更高的变异性)。
这种复杂的关系意味着我们不能只使用观察到的方差来解释组内变异。相反,DESeq2 使用离散。
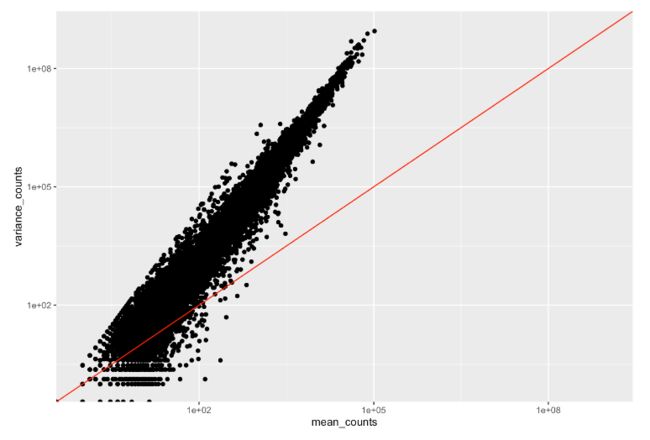 relationship
relationship
- 什么是离散(
dispersion)?
离散参数通过描述方差偏离均值的程度来模拟组内变异性。离散度为 1 表示没有偏离均值(即均值 == 方差)。一个典型的 RNA-seq 数据集,将在重复中表现出一定数量的生物变异性,因此我们将始终具有小于 1 的离散值。
- 离散值是如何计算的?
在 DESeq 中,我们知道给定基因的计数方差由均值和离散度建模:
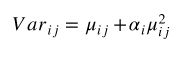 formula1
formula1
现在让我们重新排列公式,以便我们可以看到离散参数等同于什么,以便我们可以更好地理解它与均值和方差的关系:
 formula2
formula2
这也与以下内容相同:
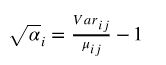 formula3
formula3
| Effect on dispersion | |
|---|---|
| Variance increases | Dispersion increases |
| Mean expression increases | Dispersion decreases |
-
DESeq2中的离散
DESeq2 根据基因的表达水平(组内重复的平均计数)和重复观察到的方差来估计每个基因的离散度,正如我们用上面的公式所证明的那样。这样,具有相同均值的基因的离散估计将仅基于它们的方差而不同。因此,离散估计反映了给定平均值的基因表达的方差。
下面,有一个离散图,其中每个黑点都是一个基因,离散是针对每个基因的平均表达绘制的。如上所述,您可以看到均值和离散之间的反比关系。黑点是根据我们拥有的数据进行的离散估计。每组只有少数 (3-6) 次重复,每个基因的变异估计通常不可靠。
为了解决这个问题,DESeq2 使用一种称为 shrinkage 的方法在基因之间共享信息,以根据基因的平均表达水平生成更准确的变异估计。 DESeq2 假定具有相似表达水平的基因应该具有相似的离散度。蓝点代表缩小的离散值。
 dispersion
dispersion
3. 拟合曲线
流程的下一步是将曲线拟合到基因方面的离散估计。将曲线拟合到数据背后的想法是,不同的基因将具有不同规模的生物变异性,但是,在所有基因中,将存在合理的离散估计分布。
 Fit curve
Fit curve
这条曲线在下图中显示为一条红线,它绘制了给定表达强度的基因的预期离散值的估计值。每个黑点都是一个基因,具有相关的平均表达水平和离散的最大似然估计 (MLE)(步骤 1)。
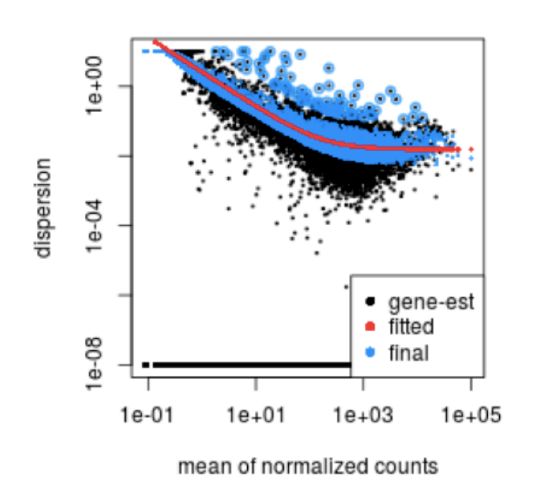 Fit curve
Fit curve
4. Shrink
流程的下一步是将基因方面的离散估计缩小到预期的离散值。
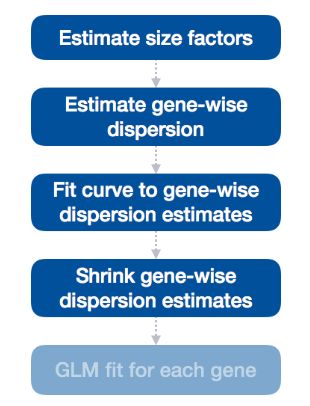 Shrink
Shrink
当样本量较小时,该曲线可以更准确地识别差异表达的基因,并且每个基因的收缩强度取决于:
- 基因离散离曲线有多近
- 样本量(更多样本 = 更少收缩)
这种收缩方法对于减少差异表达分析中的误报尤为重要。具有低离散估计的基因向曲线收缩,并且输出更准确、更高收缩值用于模型拟合和差异表达测试。这些缩小的估计值代表了确定跨组基因表达是否显著不同所需的组内变异。
略高于曲线的离散估计也会向曲线收缩,以获得更好的离散估计;然而,具有极高离散值的基因则不然。这是由于该基因可能不遵循建模假设,并且由于生物学或技术原因比其他基因具有更高的变异性。向曲线收缩值可能会导致误报,因此这些值不会收缩。这些基因显示在下面的蓝色圆圈中。
 Dispersion estimates
Dispersion estimates
这是一个很好的检查方式,以确保您的数据非常适合 DESeq2 模型。您希望您的数据通常散布在曲线周围,散布随着平均表达水平的增加而降低。如果您看到云或不同的形状,那么您可能想要更多地探索您的数据以查看您是否有污染(线粒体等)或异常样本。请注意,对于任何低自由度的实验,在 plotDispEsts() 图中的整个均值范围内收缩了多少。
令人担忧的离散图示例如下所示:
下图显示了离散值云,这些值通常不遵循曲线。这会令人担忧,并表明数据与模型的拟合不佳。
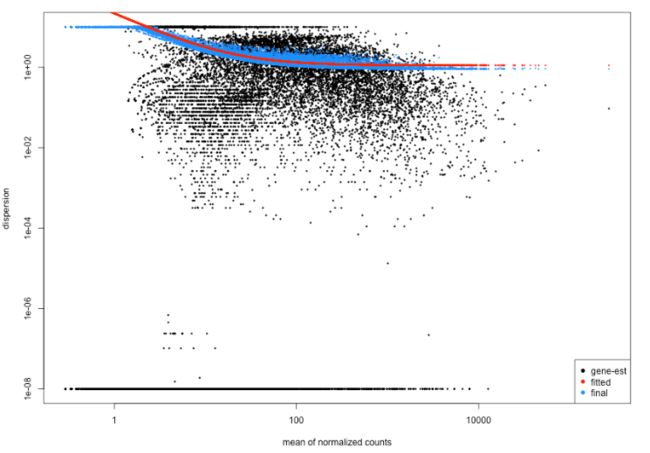 worrisome
worrisome
下图显示离散值最初下降,然后随着较大的表达值而增加。根据我们的预期,较大的平均表达值不应该有较大的离散——我们期望离散随着均值的增加而减小。这表明比预期的更高度表达的基因的变异更少。这也表明我们的分析中可能存在异常样本或污染。
 worrisome
worrisome
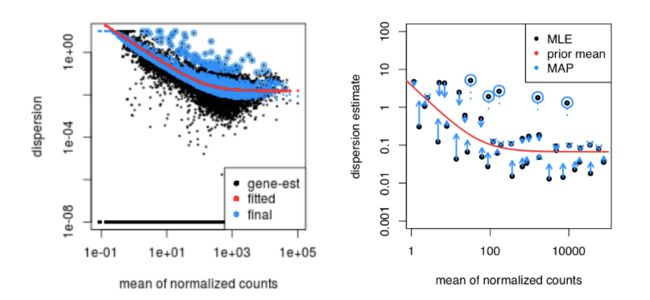
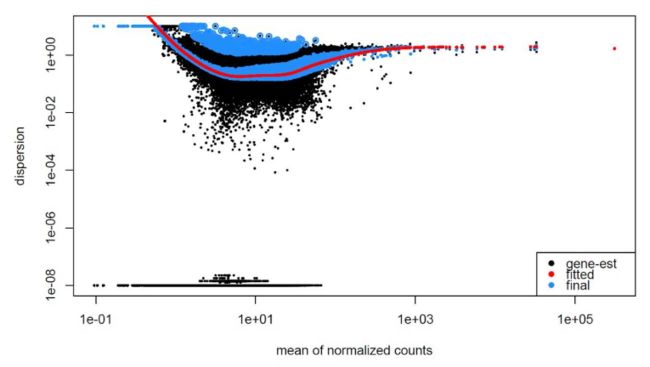
5. MOV10
让我们看一下 MOV10 数据的离散估计:
# Plot dispersion estimates
plotDispEsts(dds)
 MOV10
MOV10
由于我们的样本量很小,因此对于许多基因,我们看到了相当多的收缩。那么我们的数据适合该模型吗?
随着平均表达的增加,我们看到离散度有很好的降低,这是好的。我们还看到离散估计通常围绕曲线,这也是预期的。总的来说,看起来不错。我们确实看到了强烈的收缩,这可能是因为我们的一个样本组只有两个重复。我们拥有的重复越多,对离散估计应用的收缩越少,能够识别的 DE 基因就越多。我们通常建议每个条件至少进行 4 次生物重复,以便更好地估计变异。
欢迎Star -> 学习目录
更多教程 -> 学习目录
本文由 mdnice 多平台发布