YOLOv5的Tricks | 【Trick5】遗传算法实现超参数进化(Hyperparameter Evolution)
如有错误,恳请指出。
文章目录
- 1. 遗传算法介绍
- 2. 遗传算法进化超参数
-
- 2.1 实现思路
- 2.2 实现代码
- 3. Hyperparameter Evolution使用
Hyperparameter evolution超参数演化是一种使用遗传算法(GA)进行优化的超参数优化方法。ML 中的超参数控制训练的各个方面,为它们找到最佳值可能是一个挑战。由于 1) 高维搜索空间 2) 维度之间的未知相关性,以及 3) 评估每个点的适应度的昂贵性质,网格搜索等传统方法很快就会变得难以处理,这使得 GA 成为超参数搜索的合适候选者。
以上是官方文档的介绍:Hyperparameter Evolution,其实简单的来说,就是使用遗传算法来对超参数已经交叉变异以获得更好的结果,而不需要传统的网格搜索。这个想法其实在之前yolov3-spp中也介绍过,对于anchor聚类结果使用遗传算法来获取更好的anchor设置,笔记见:使用kmeans与遗传算法聚类anchor
至此,在整个yolov5项目中,其实对遗传算法使用了在两个地方中,一个是对anchor进行变异优化;另外一个就是这篇笔记所介绍的,对超参数进行变异优化。
1. 遗传算法介绍
具体介绍可以参考:遗传算法详解 附python代码实现,这里稍微提取一些概念。
-
简要
遗传算法是用于解决最优化问题的一种搜索算法。从名字来看,遗传算法借用了生物学里达尔文的进化理论:”适者生存,不适者淘汰“,将该理论以算法的形式表现出来就是遗传算法的过程。 -
种群和个体的概念
遗传算法启发自进化理论,而我们知道进化是由种群为单位的,种群是什么呢?维基百科上解释为:在生物学上,是在一定空间范围内同时生活着的同种生物的全部个体。显然要想理解种群的概念,又先得理解个体的概念,在遗传算法里,个体通常为某个问题的一个解,并且该解在计算机中被编码为一个向量表示。求最大值问题的一个可能解,也就是遗传算法里的个体,把这样的一组一组的可能解的集合就叫做种群。 -
编码、解码与染色体的概念
在上面个体概念里提到个体(也就是一组可能解)在计算机程序中被编码为一个向量表示,而在我们这个问题中,个体是x , y的取值,是两个实数,所以问题就可以转化为如何将实数编码为一个向量表示。
只要我们能够将不同的实数表示成不同的0,1二进制串表示就完成了编码,也就是说其实我们并不需要去了解一个实数对应的二进制具体是多少,我们只需要保证有一个映射能够将十进制的数编码为二进制即可,至于这个映射是什么,其实可以不必关心。将个体(可能解)编码后的二进制串叫做染色体,染色体(或者有人叫DNA)就是个体(可能解)的二进制编码表示。
而在最后我们肯定要将编码后的二进制串转换为我们理解的十进制串,也就是将二进制转化为十进制,这个过程叫做解码。 -
适应度和选择
已经得到了一个种群,现在要根据适者生存规则把优秀的个体保存下来,同时淘汰掉那些不适应环境的个体。现在摆在我们面前的问题是如何评价一个个体对环境的适应度?在我们的求最大值的问题中可以直接用可能解(个体)对应的函数的函数值的大小来评估,这样可能解对应的函数值越大越有可能被保留下来。
不同问题的适应度设置不同。遗传算法依据原则:适应度越高,被选择的机会越高,而适应度低的,被选择的机会就低。 -
交叉、变异
通过选择我们得到了当前看来“还不错的基因”,但是这并不是最好的基因,我们需要通过繁殖后代(包含有交叉+变异过程)来产生比当前更好的基因,但是繁殖后代并不能保证每个后代个体的基因都比上一代优秀,这时需要继续通过选择过程来让试应环境的个体保留下来,从而完成进化,不断迭代上面这个过程种群中的个体就会一步一步地进化。
具体地繁殖后代过程包括交叉和变异两步。交叉是指每一个个体是由父亲和母亲两个个体繁殖产生,子代个体的DNA(二进制串)获得了一半父亲的DNA,一半母亲的DNA,但是这里的一半并不是真正的一半,这个位置叫做交配点,是随机产生的,可以是染色体的任意位置。通过交叉子代获得了一半来自父亲一半来自母亲的DNA,但是子代自身可能发生变异,使得其DNA即不来自父亲,也不来自母亲,在某个位置上发生随机改变,通常就是改变DNA的一个二进制位(0变到1,或者1变到0)。
需要说明的是交叉和变异不是必然发生,而是有一定概率发生。
参考代码:
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import cm
from mpl_toolkits.mplot3d import Axes3D
DNA_SIZE = 24
POP_SIZE = 200
CROSSOVER_RATE = 0.8
MUTATION_RATE = 0.005
N_GENERATIONS = 50
X_BOUND = [-3, 3]
Y_BOUND = [-3, 3]
# 定义的一个优化问题
def F(x, y):
return 3*(1-x)**2*np.exp(-(x**2)-(y+1)**2)- 10*(x/5 - x**3 - y**5)*np.exp(-x**2-y**2)- 1/3**np.exp(-(x+1)**2 - y**2)
# 绘制3D图像
def plot_3d(ax):
X = np.linspace(*X_BOUND, 100)
Y = np.linspace(*Y_BOUND, 100)
X,Y = np.meshgrid(X, Y)
Z = F(X, Y)
ax.plot_surface(X,Y,Z,rstride=1,cstride=1,cmap=cm.coolwarm)
ax.set_zlim(-10,10)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_zlabel('z')
plt.pause(3)
plt.show()
# 解码过程
# 说明: 设置DNA_SIZE=24(一个实数DNA长度),两个实数x,y一共用48位二进制编码,我同时将x和y编码到同一个48位的二进制串里,每一个变量用24位表示
# 奇数24列为x的编码表示,偶数24列为y的编码表示
def translateDNA(pop): #pop表示种群矩阵,一行表示一个二进制编码表示的DNA,矩阵的行数为种群数目
x_pop = pop[:,1::2]#奇数列表示X
y_pop = pop[:,::2] #偶数列表示y
#pop:(POP_SIZE,DNA_SIZE)*(DNA_SIZE,1) --> (POP_SIZE,1)
x = x_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(X_BOUND[1]-X_BOUND[0])+X_BOUND[0]
y = y_pop.dot(2**np.arange(DNA_SIZE)[::-1])/float(2**DNA_SIZE-1)*(Y_BOUND[1]-Y_BOUND[0])+Y_BOUND[0]
return x,y
# 交叉变异操作
# 说明: 交叉是指每一个个体是由父亲和母亲两个个体繁殖产生,子代个体的DNA(二进制串)获得了一半父亲的DNA,一半母亲的DNA.
# 但是这里的一半并不是真正的一半,这个位置叫做交配点,是随机产生的,可以是染色体的任意位置。通过交叉子代获得了一半来自父亲一半来自母亲的DNA
def crossover_and_mutation(pop, CROSSOVER_RATE = 0.8):
new_pop = []
for father in pop: #遍历种群中的每一个个体,将该个体作为父亲
child = father #孩子先得到父亲的全部基因(这里我把一串二进制串的那些0,1称为基因)
if np.random.rand() < CROSSOVER_RATE: #产生子代时不是必然发生交叉,而是以一定的概率发生交叉
mother = pop[np.random.randint(POP_SIZE)] #再种群中选择另一个个体,并将该个体作为母亲
cross_points = np.random.randint(low=0, high=DNA_SIZE*2) #随机产生交叉的点
child[cross_points:] = mother[cross_points:] #孩子得到位于交叉点后的母亲的基因
mutation(child) #每个后代有一定的机率发生变异
new_pop.append(child)
return new_pop
# 变异操作
# 说明: 在交叉过程外,子代自身可能发生变异,使得其DNA即不来自父亲,也不来自母亲,在某个位置上发生随机改变
# 通常就是改变DNA的一个二进制位(0变到1,或者1变到0)
def mutation(child, MUTATION_RATE=0.003):
if np.random.rand() < MUTATION_RATE: #以MUTATION_RATE的概率进行变异
mutate_point = np.random.randint(0, DNA_SIZE*2) #随机产生一个实数,代表要变异基因的位置
child[mutate_point] = child[mutate_point]^1 #将变异点的二进制为反转
# 适应度
# 说明: 求最大值的问题中可以直接用可能解(个体)对应的函数的函数值的大小来评估,这样可能解对应的函数值越大越有可能被保留下来
def get_fitness(pop):
x,y = translateDNA(pop) # 解码, 将二进制串映射到指定范围内(也就是区间[-3, 3])
# pred是将可能解带入函数F中得到的预测值,因为后面的选择过程需要根据个体适应度确定每个个体被保留下来的概率,而概率不能是负值,所以减去预测中的最小值把适应度值的最小区间提升到从0开始
# 但是如果适应度为0,其对应的概率也为0,表示该个体不可能在选择中保留下来,这不符合算法思想,遗传算法不绝对否定谁也不绝对肯定谁,所以最后加上了一个很小的正数。
pred = F(x, y)
return (pred - np.min(pred)) + 1e-3 #减去最小的适应度是为了防止适应度出现负数,通过这一步fitness的范围为[0, np.max(pred)-np.min(pred)],最后在加上一个很小的数防止出现为0的适应度
# 种群选择
# 说明: 选择则是根据新个体的适应度进行,但同时不意味着完全以适应度高低为导向(选择top k个适应度最高的个体,容易陷入局部最优解)
# 因为单纯选择适应度高的个体将可能导致算法快速收敛到局部最优解而非全局最优解,我们称之为早熟
# 作为折中,遗传算法依据原则:适应度越高,被选择的机会越高,而适应度低的,被选择的机会就低
def select(pop, fitness): # nature selection wrt pop's fitness
# 这里主要是使用了choice里的最后一个参数p,参数p描述了从np.arange(POP_SIZE)里选择每一个元素的概率,概率越高约有可能被选中
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=(fitness)/(fitness.sum()) ) # 返回的是被选择种群的所以,这里可能是重复的索引
# 最后返回被选中的个体即可
return pop[idx]
# 打印相关信息
def print_info(pop):
fitness = get_fitness(pop)
max_fitness_index = np.argmax(fitness)
print("max_fitness:", fitness[max_fitness_index])
x,y = translateDNA(pop)
print("最优的基因型:", pop[max_fitness_index])
print("(x, y):", (x[max_fitness_index], y[max_fitness_index]))
if __name__ == "__main__":
fig = plt.figure()
ax = Axes3D(fig)
plt.ion()#将画图模式改为交互模式,程序遇到plt.show不会暂停,而是继续执行
plot_3d(ax)
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE*2)) #matrix (POP_SIZE, DNA_SIZE)
for _ in range(N_GENERATIONS):#迭代N代
x,y = translateDNA(pop)
if 'sca' in locals():
sca.remove()
sca = ax.scatter(x, y, F(x,y), c='black', marker='o');plt.show();plt.pause(0.1)
pop = np.array(crossover_and_mutation(pop, CROSSOVER_RATE))
#F_values = F(translateDNA(pop)[0], translateDNA(pop)[1])#x, y --> Z matrix
fitness = get_fitness(pop)
pop = select(pop, fitness) #选择生成新的种群
print_info(pop)
plt.ioff()
plot_3d(ax)
以上为完整的代码,解码、适应度与选择、交叉变异这些步骤是遗传算法的核心模块,将这些模块在主函数中迭代起来,让种群去进化,核心的迭代代码如下所示:
# 随机生成种群 matrix (POP_SIZE, DNA_SIZE)
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE*2))
for _ in range(N_GENERATIONS): # 种群迭代进化N_GENERATIONS代
crossover_and_mutation(pop, CROSSOVER_RATE) # 种群通过交叉变异产生后代
fitness = get_fitness(pop) # 对种群中的每个个体进行评估
pop = select(pop, fitness) # 选择生成新的种群
2. 遗传算法进化超参数
yolov5中包含差不多30个超参数来对训练过程进行设置,如此多的超参数如果使用网格搜索来获得最佳结果是比较困难的,所以这里作者使用了遗传算法来求出一个局部最优解——获得较好的超参数结果。
2.1 实现思路
这里一般用对COCO训练的默认超参数作为初始(演化前)的超参数:hyp.scratch.yaml,其内容如下所示:
# YOLOv5 by Ultralytics, GPL-3.0 license
# Hyperparameters for COCO training from scratch
# python train.py --batch 40 --cfg yolov5m.yaml --weights '' --data coco.yaml --img 640 --epochs 300
# See tutorials for hyperparameter evolution https://github.com/ultralytics/yolov5#tutorials
lr0: 0.01 # initial learning rate (SGD=1E-2, Adam=1E-3)
lrf: 0.1 # final OneCycleLR learning rate (lr0 * lrf)
momentum: 0.937 # SGD momentum/Adam beta1
weight_decay: 0.0005 # optimizer weight decay 5e-4
warmup_epochs: 3.0 # warmup epochs (fractions ok)
warmup_momentum: 0.8 # warmup initial momentum
warmup_bias_lr: 0.1 # warmup initial bias lr
box: 0.05 # box loss gain
cls: 0.5 # cls loss gain
cls_pw: 1.0 # cls BCELoss positive_weight
obj: 1.0 # obj loss gain (scale with pixels)
obj_pw: 1.0 # obj BCELoss positive_weight
iou_t: 0.20 # IoU training threshold
anchor_t: 4.0 # anchor-multiple threshold
# anchors: 3 # anchors per output layer (0 to ignore)
fl_gamma: 0.0 # focal loss gamma (efficientDet default gamma=1.5)
hsv_h: 0.015 # image HSV-Hue augmentation (fraction)
hsv_s: 0.7 # image HSV-Saturation augmentation (fraction)
hsv_v: 0.4 # image HSV-Value augmentation (fraction)
degrees: 0.0 # image rotation (+/- deg)
translate: 0.1 # image translation (+/- fraction)
scale: 0.5 # image scale (+/- gain)
shear: 0.0 # image shear (+/- deg)
perspective: 0.0 # image perspective (+/- fraction), range 0-0.001
flipud: 0.0 # image flip up-down (probability)
fliplr: 0.5 # image flip left-right (probability)
mosaic: 1.0 # image mosaic (probability)
mixup: 0.0 # image mixup (probability)
copy_paste: 0.0 # segment copy-paste (probability)
可以看见,这一系列的超参数是控制训练过程中的一些参数设置,所以可以将其看成是一个自变量x。对这些超参数x进行训练会得到一个结果y,这个y返回了7个结果,分别是:'metrics/precision', 'metrics/recall', 'metrics/mAP_0.5', 'metrics/mAP_0.5:0.95', 'val/box_loss', 'val/obj_loss', 'val/cls_loss'
对于这个输入x需要判断其效果,也就是需要定义一个适应度fitness,这里定义的fitness是精确度R,召回率R,以及mAP_0.5与mAP_0.5:0.95的分数加权和,与Loss部分无关(当然可以自行改动这个fitness的定义),如下所示:
def fitness(x):
# Model fitness as a weighted combination of metrics
w = [0.0, 0.0, 0.1, 0.9] # weights for [P, R, [email protected], [email protected]:0.95]
return (x[:, :4] * w).sum(1)
那么如果进行变异后的x,所返回的结果y,计算得到其适应度是比当前要高的,那么就说明当前的变异是对效果是提升的,变异后的参数是可取的。这里会默认会变异300次,然后每一次变异选择当前之前变异效果最好的前n个超参数x’来作为下一次的变异结果。
这里yolov5提供了两个不同进化方式获得base hyp
- single方式: 根据每个hyp的权重随机选择一个之前的hyp作为base hyp
- weighted方式: 根据每个hyp的权重对之前所有的hyp进行融合获得一个base hyp
根据前n次效果最好的超参数x来进行变异处理,获得下一个待输入的x’,依次反复迭代300次,变异300回来获得效果最好的x。
2.2 实现代码
yolov5实现代码参考如下:
def main(opt, callbacks=Callbacks()):
...
# Resume
if opt.resume and not check_wandb_resume(opt) and not opt.evolve: # resume an interrupted run
ckpt = opt.resume if isinstance(opt.resume, str) else get_latest_run() # specified or most recent path
assert os.path.isfile(ckpt), 'ERROR: --resume checkpoint does not exist'
with open(Path(ckpt).parent.parent / 'opt.yaml', errors='ignore') as f:
opt = argparse.Namespace(**yaml.safe_load(f)) # replace
opt.cfg, opt.weights, opt.resume = '', ckpt, True # reinstate
LOGGER.info(f'Resuming training from {ckpt}')
else:
opt.data, opt.cfg, opt.hyp, opt.weights, opt.project = \
check_file(opt.data), check_yaml(opt.cfg), check_yaml(opt.hyp), str(opt.weights), str(opt.project) # checks
assert len(opt.cfg) or len(opt.weights), 'either --cfg or --weights must be specified'
if opt.evolve:
opt.project = str(ROOT / 'runs/evolve')
opt.exist_ok, opt.resume = opt.resume, False # pass resume to exist_ok and disable resume
opt.save_dir = str(increment_path(Path(opt.project) / opt.name, exist_ok=opt.exist_ok))
...
# Train
if not opt.evolve:
train(opt.hyp, opt, device, callbacks)
if WORLD_SIZE > 1 and RANK == 0:
LOGGER.info('Destroying process group... ')
dist.destroy_process_group()
# Evolve hyperparameters (optional)
else:
# Hyperparameter evolution metadata (mutation scale 0-1, lower_limit, upper_limit)
# 三个数值分别对应着: 变异初始概率, 最低限值, 最大限值
meta = {'lr0': (1, 1e-5, 1e-1), # initial learning rate (SGD=1E-2, Adam=1E-3)
'lrf': (1, 0.01, 1.0), # final OneCycleLR learning rate (lr0 * lrf)
'momentum': (0.3, 0.6, 0.98), # SGD momentum/Adam beta1
'weight_decay': (1, 0.0, 0.001), # optimizer weight decay
'warmup_epochs': (1, 0.0, 5.0), # warmup epochs (fractions ok)
'warmup_momentum': (1, 0.0, 0.95), # warmup initial momentum
'warmup_bias_lr': (1, 0.0, 0.2), # warmup initial bias lr
'box': (1, 0.02, 0.2), # box loss gain
'cls': (1, 0.2, 4.0), # cls loss gain
'cls_pw': (1, 0.5, 2.0), # cls BCELoss positive_weight
'obj': (1, 0.2, 4.0), # obj loss gain (scale with pixels)
'obj_pw': (1, 0.5, 2.0), # obj BCELoss positive_weight
'iou_t': (0, 0.1, 0.7), # IoU training threshold
'anchor_t': (1, 2.0, 8.0), # anchor-multiple threshold
'anchors': (2, 2.0, 10.0), # anchors per output grid (0 to ignore)
'fl_gamma': (0, 0.0, 2.0), # focal loss gamma (efficientDet default gamma=1.5)
'hsv_h': (1, 0.0, 0.1), # image HSV-Hue augmentation (fraction)
'hsv_s': (1, 0.0, 0.9), # image HSV-Saturation augmentation (fraction)
'hsv_v': (1, 0.0, 0.9), # image HSV-Value augmentation (fraction)
'degrees': (1, 0.0, 45.0), # image rotation (+/- deg)
'translate': (1, 0.0, 0.9), # image translation (+/- fraction)
'scale': (1, 0.0, 0.9), # image scale (+/- gain)
'shear': (1, 0.0, 10.0), # image shear (+/- deg)
'perspective': (0, 0.0, 0.001), # image perspective (+/- fraction), range 0-0.001
'flipud': (1, 0.0, 1.0), # image flip up-down (probability)
'fliplr': (0, 0.0, 1.0), # image flip left-right (probability)
'mosaic': (1, 0.0, 1.0), # image mixup (probability)
'mixup': (1, 0.0, 1.0), # image mixup (probability)
'copy_paste': (1, 0.0, 1.0)} # segment copy-paste (probability)
with open(opt.hyp, errors='ignore') as f:
hyp = yaml.safe_load(f) # load hyps dict
if 'anchors' not in hyp: # anchors commented in hyp.yaml
hyp['anchors'] = 3
opt.noval, opt.nosave, save_dir = True, True, Path(opt.save_dir) # only val/save final epoch
# ei = [isinstance(x, (int, float)) for x in hyp.values()] # evolvable indices
evolve_yaml, evolve_csv = save_dir / 'hyp_evolve.yaml', save_dir / 'evolve.csv'
if opt.bucket:
os.system(f'gsutil cp gs://{opt.bucket}/evolve.csv {save_dir}') # download evolve.csv if exists
# 默认迭代演化300次, 每次变异都需要训练一次要查看效果result, 所以计算量呈倍数递增
# 官方建议至少进行 300 代进化以获得最佳结果, 而基础场景被训练了数百次, 可能需要数百或数千个GPU小时
for _ in range(opt.evolve): # generations to evolve
if evolve_csv.exists(): # if evolve.csv exists: select best hyps and mutate
# Select parent(s):选择超参进化方式 只用single和weighted两种
parent = 'single' # parent selection method: 'single' or 'weighted'
# 加载evolve.csv文件, skiprows=1跳过第一行, delimiter加载文件分隔符
x = np.loadtxt(evolve_csv, ndmin=2, delimiter=',', skiprows=1)
# 最多选择5个最好的变异结果来挑选
n = min(5, len(x)) # number of previous results to consider
# np.argsort只能从小到大排序, 添加负号实现从大到小排序, 算是排序的一个代码技巧
# 挑选出适应度最好的前n个样本数据, 每个样本包含29个超参数变异结构{array:29}
x = x[np.argsort(-fitness(x))][:n] # top n mutations
# 根据(mp, mr, map50, map)的加权和来作为权重
w = fitness(x) - fitness(x).min() + 1E-6 # weights (sum > 0)
# 根据不同进化方式获得base hyp
# single方式: 根据每个hyp的权重随机选择一个之前的hyp作为base hyp
# weighted方式: 根据每个hyp的权重对之前所有的hyp进行融合获得一个base hyp
if parent == 'single' or len(x) == 1:
# 根据权重的几率随机挑选适应度历史前5的其中一个
# x = x[random.randint(0, n - 1)] # random selection
x = x[random.choices(range(n), weights=w)[0]] # weighted selection
elif parent == 'weighted':
# 对hyp乘上对应的权重融合层一个hpy, 再取平均(除以权重和)
x = (x * w.reshape(n, 1)).sum(0) / w.sum() # weighted combination
# Mutate 超参进化
mp, s = 0.8, 0.2 # mutation probability, sigma 设置突变概率与方差
npr = np.random
npr.seed(int(time.time()))
# 获取突变初始值, 也就是meta三个值的第一个数据
# 三个数值分别对应着: 变异初始概率, 最低限值, 最大限值(mutation scale 0-1, lower_limit, upper_limit)
g = np.array([meta[k][0] for k in hyp.keys()]) # gains 0-1
ng = len(meta)
v = np.ones(ng) # 确保至少其中有一个超参变异了
# 其实这里的v都是1附近的一个数值, 也就是对超参进行一个1附近微亮的变异
while all(v == 1): # mutate until a change occurs (prevent duplicates)
v = (g * (npr.random(ng) < mp) * npr.randn(ng) * npr.random() * s + 1).clip(0.3, 3.0)
for i, k in enumerate(hyp.keys()): # plt.hist(v.ravel(), 300)
# [i+7]是因为x中前7个数字为results的指标(P,R,mAP,F1,test_loss=(box,obj,cls)),之后才是超参数hyp
hyp[k] = float(x[i + 7] * v[i]) # mutate 超参变异
# Constrain to limits 限制超参范围
for k, v in meta.items():
hyp[k] = max(hyp[k], v[1]) # lower limit
hyp[k] = min(hyp[k], v[2]) # upper limit
hyp[k] = round(hyp[k], 5) # significant digits: 有效数字为5, 也就是留5个小数
# Train mutation: result{tuple 7}: mp, mr, map50, map, box, obj, cls
# 具体返回的是: 'metrics/precision', 'metrics/recall', 'metrics/mAP_0.5', 'metrics/mAP_0.5:0.95',
# 'val/box_loss', 'val/obj_loss', 'val/cls_loss'
results = train(hyp.copy(), opt, device, callbacks)
# Write mutation results
# 将每一代的演化结果与训练结果记录在evolve.csv文件中
print_mutation(results, hyp.copy(), save_dir, opt.bucket)
# Plot results
# 绘图: 每个超参数有一个子图, 显示适应度(y 轴)与超参数值(x 轴).黄色表示更高的浓度
plot_evolve(evolve_csv)
print(f'Hyperparameter evolution finished\n'
f"Results saved to {colorstr('bold', save_dir)}\n"
f'Use best hyperparameters example: $ python train.py --hyp {evolve_yaml}')
# 适应度的计算(用来挑选变异结果)
def fitness(x):
# Model fitness as a weighted combination of metrics
w = [0.0, 0.0, 0.1, 0.9] # weights for [P, R, [email protected], [email protected]:0.95]
return (x[:, :4] * w).sum(1)
# 将每一代的演化结果与训练结果记录在evolve.csv文件中
def print_mutation(results, hyp, save_dir, bucket):
# keys/vals: (mp, mr, map50, map, box, obj, cls) + (lr0, lr1, ... , copy_paste, anchors)
evolve_csv, results_csv, evolve_yaml = save_dir / 'evolve.csv', save_dir / 'results.csv', save_dir / 'hyp_evolve.yaml'
keys = ('metrics/precision', 'metrics/recall', 'metrics/mAP_0.5', 'metrics/mAP_0.5:0.95',
'val/box_loss', 'val/obj_loss', 'val/cls_loss') + tuple(hyp.keys()) # [results + hyps]
keys = tuple(x.strip() for x in keys)
vals = results + tuple(hyp.values())
n = len(keys)
# Download (optional)
if bucket:
url = f'gs://{bucket}/evolve.csv'
if gsutil_getsize(url) > (os.path.getsize(evolve_csv) if os.path.exists(evolve_csv) else 0):
os.system(f'gsutil cp {url} {save_dir}') # download evolve.csv if larger than local
# Log to evolve.csv:记录当前代目的演化超参数数据与训练数据
s = '' if evolve_csv.exists() else (('%20s,' * n % keys).rstrip(',') + '\n') # add header
with open(evolve_csv, 'a') as f:
f.write(s + ('%20.5g,' * n % vals).rstrip(',') + '\n')
# Print to screen
print(colorstr('evolve: ') + ', '.join(f'{x.strip():>20s}' for x in keys))
print(colorstr('evolve: ') + ', '.join(f'{x:20.5g}' for x in vals), end='\n\n\n')
# Save yaml
with open(evolve_yaml, 'w') as f:
data = pd.read_csv(evolve_csv)
data = data.rename(columns=lambda x: x.strip()) # strip keys
# 根据曾经所有训练测试的结果: mp, mr, map50, map来计算适应度(fitness)
# 挑选出其中适应度最强的代目(其实就是根据mp, mr, map50, map的权重和分数, 越高适应度最强)
i = np.argmax(fitness(data.values[:, :7]))
# 将适应度最好的那一代的相关结果写在hyp_evolve.yaml文件的注释中
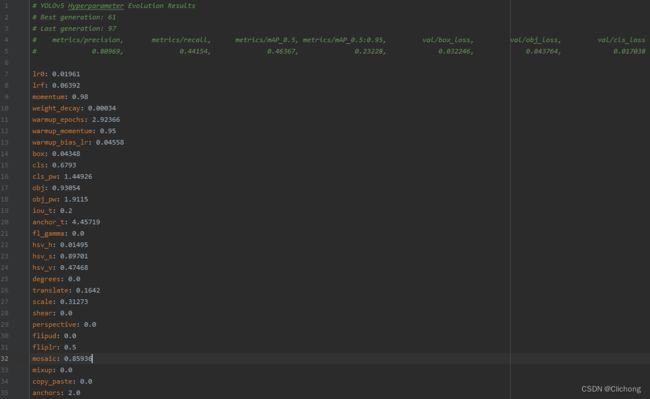
f.write('# YOLOv5 Hyperparameter Evolution Results\n' +
f'# Best generation: {i}\n' +
f'# Last generation: {len(data)}\n' +
'# ' + ', '.join(f'{x.strip():>20s}' for x in keys[:7]) + '\n' +
'# ' + ', '.join(f'{x:>20.5g}' for x in data.values[i, :7]) + '\n\n')
# 保存当前的演化超参数hpy在hyp_evolve.yaml文件
yaml.safe_dump(hyp, f, sort_keys=False)
if bucket:
os.system(f'gsutil cp {evolve_csv} {evolve_yaml} gs://{bucket}') # upload
具体过程见注释。
3. Hyperparameter Evolution使用
具体介绍见:Hyperparameter Evolution
使用方式其实很简单,就是设置一些evolve参数即可
python train.py --epochs 10 --data coco128.yaml --weights yolov5s.pt --cache --evolve
或者我对args进行了改动,使得可以设定迭代演化次数:
parser.add_argument('--evolve', type=int, default=300, help='evolve hyperparameters for x generations')
...
python train.py --epochs 10 --data coco128.yaml --weights yolov5s.pt --cache --evolve 10
Hyperparameter Evolution主要的遗传算子是交叉和变异。在这项工作中,使用突变,以 80% 的概率和 0.04 的方差基于所有前几代最好的父母的组合来创造新的后代。这里结果被记录到runs/evolve/exp/evolve.csv中,最高适应度的后代被保存为每一代runs/evolve/hyp_evolved.yaml
- evolve.csv展示:
- hyp_evolved.yaml展示:
参考资料:
1. 遗传算法详解 附python代码实现
2. 目标检测的Tricks | 【Trick13】使用kmeans与遗传算法聚类anchor
3. Hyperparameter Evolution
4. 【YOLOV5-5.x 源码解读】train.py