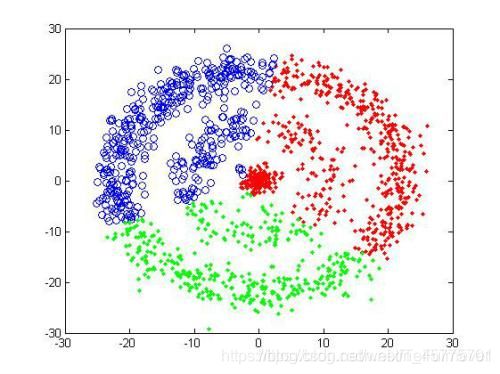
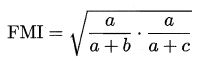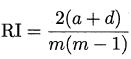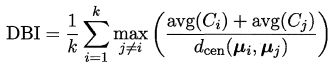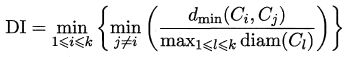《机器学习》周志华--第9章聚类 笔记+习题
https://www.cnblogs.com/hzcya1995/p/13302691.html
9.1聚类任务
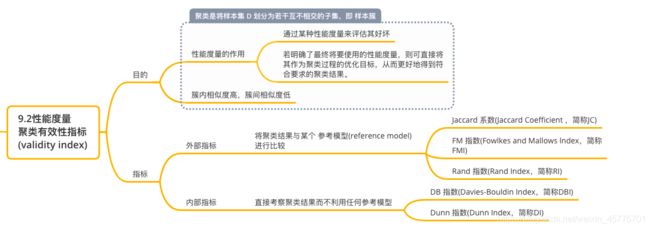
9.2性能度量

基于式 (9.1 ) ~ (9.4),可导出下面这些常用的聚类性能度量外部指标:
基于这四个式子,可导出下面这些常用的聚类性能度量内部指标:
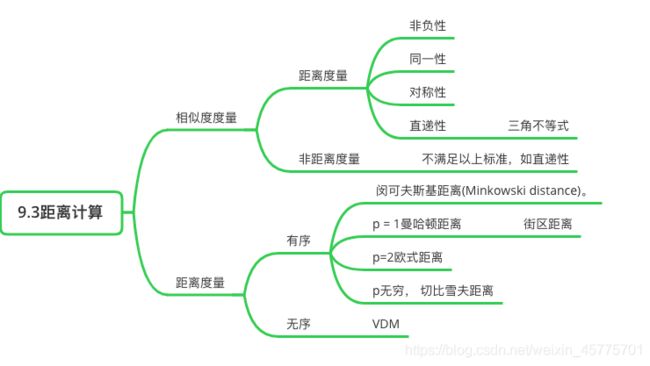
9.3距离计算
距离度量
属性经常划分为 连续属性(continuous attribute),亦称 数值属性(numerical att巾ute);和 离散属性(categorical attribute),亦称 列名属性(nominal attribute)。前者在定义域上有无穷多个可能的取值,后者在定义域上是有限个取值。
然而,在讨论距离计算时,属性上是否定了"序"关系更为重要。
9.4原型聚类

其中第 1 行对均值向量进行初始化,在第 4-8 行与第 9一16 行依次对当前簇划分及均值向量选代更新,若迭代更新后聚类结果保持不变,则在第 18 行将当前簇划分结果返回。
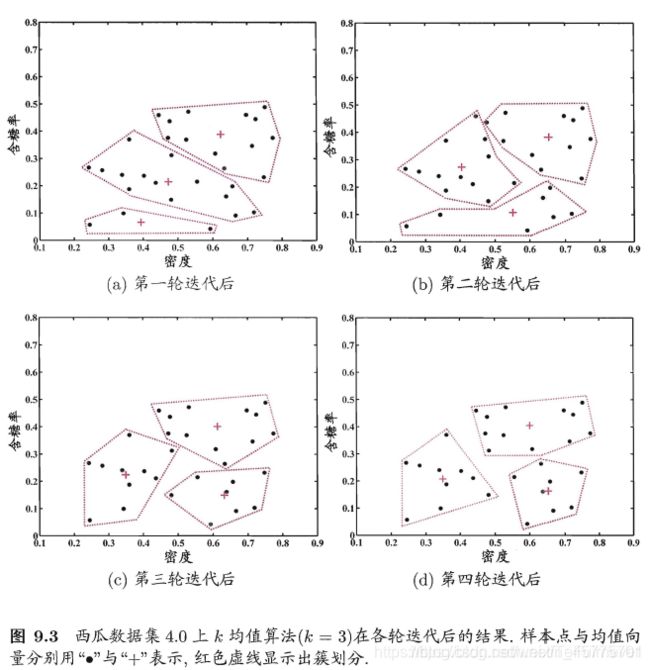
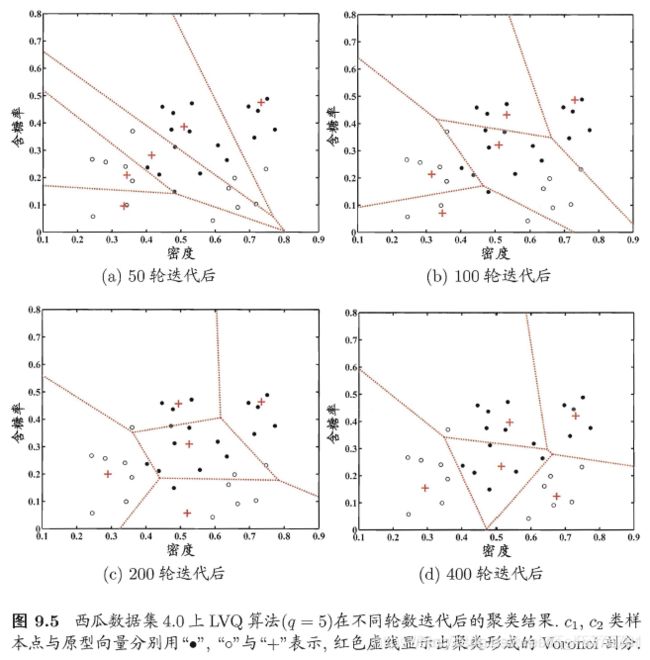
- 学习向量量化
与 k 均值算法类似,学习向量量化(Learning Vector Quantization,简称LVQ) 也是试图找到一组原型向量来刻画聚类结构, 但与一般聚类算法不同的是, LVQ 假设数据样本带有类别标记,学习过程利用样本的这些监督信息来辅助聚类。

LVQ 算法描述如下图所示:


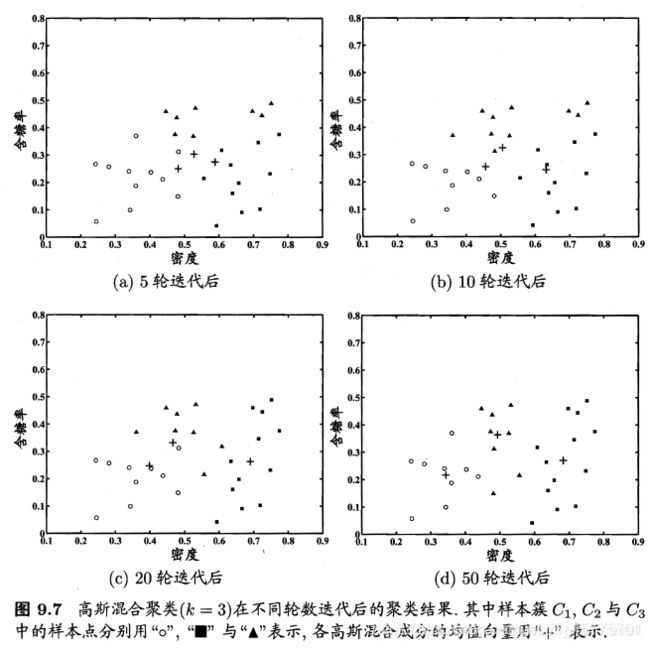
- 高斯混合聚类
高斯混合聚类与 k 均值、LVQ 用原型向量来刻画聚类结构不同,高斯混合(Mixture-of-Gaussian)聚类 采用概率模型来表达聚类原型。
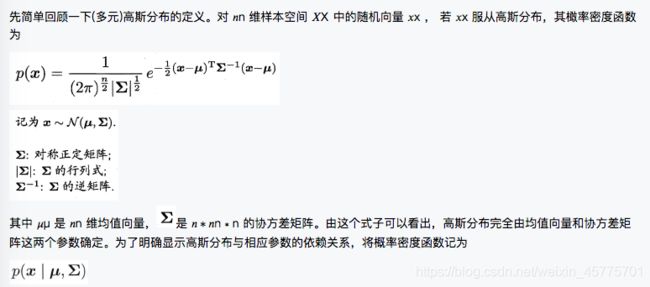

因此,从原型聚类的角度来看,高斯混合聚类是采用概率模型(高斯分布)对原型进行刻画,簇划分则由原型对应后验概率确定。那么模型参数如何求解呢?
可采用极大似然估计,即最大化(对数)似然: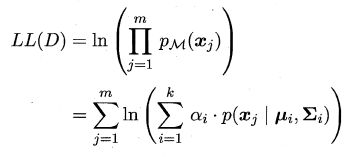
常采用EM 算法进行迭代优化求解。
若参数能最大化似然函数,则由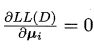
有如下


即每个高斯成分的混合系数由样本属于该成分的平均后验概率确定。
由上述推导即可获得高斯混合模型的 EM 算法。
9.5密度聚类

简单来说,就是字面意思,核心对象的核心,密度直达的直达,密度可达的可达,密度相连的相连,就好比集合之间的碰撞一下。
基于这些概念, DBSCAN 将"簇"定义为:由密度可达关系导出的最大的密度相连样本集合。形式化地说,给定邻域参数 , 簇 C 是满足以下性质的非空样本子集:
于是, DBSCAN 算法先任选数据集中的一个核心对象为种子(seed),再由此出发确定相应的聚类簇,算法描述如下所示:
在第 1-7 行中,算法先根据给定的邻域参数找出所有核心对象;然后在第 10-24 行中,以任一核心对象为出发点,找出由其密度可达的样本生成聚类簇,直到所有核心对象均被访问过为止。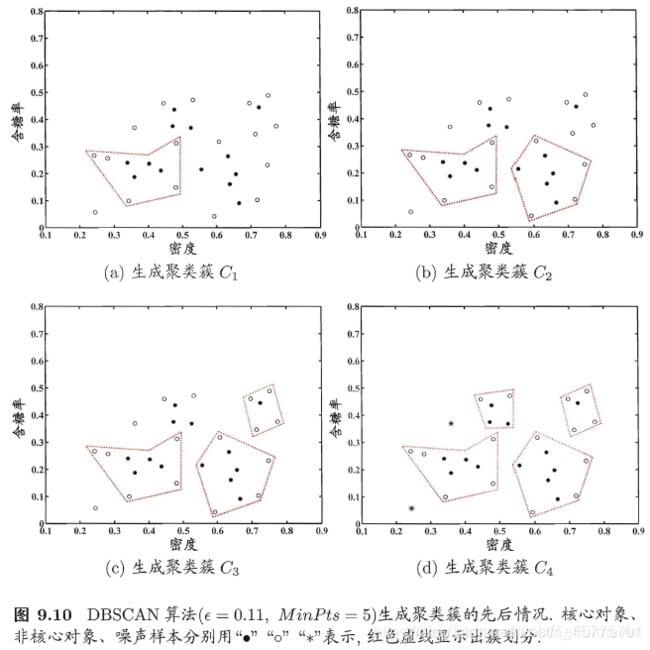

9.6层次聚类
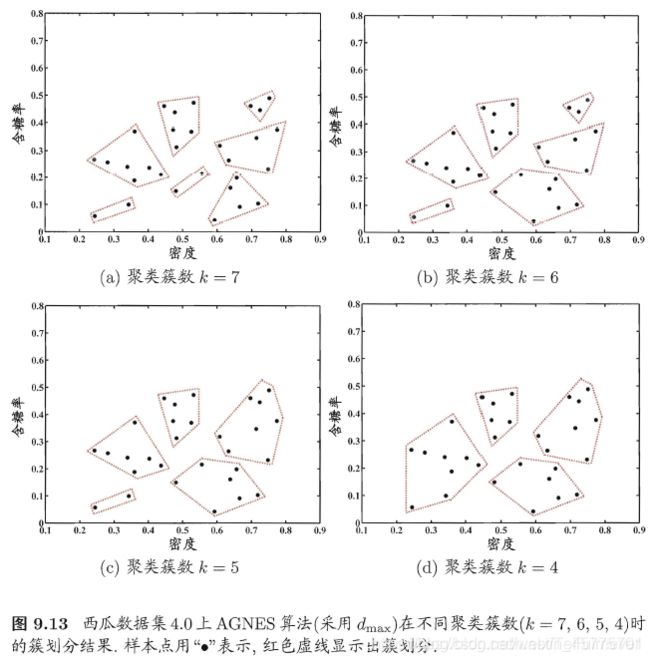
在第 1-9 行,算法先对仅含一个样本的初始聚类簇和相应的距离矩阵进行初始化;然后在第 11-23 行, AGNES 不断合
并距离最近的聚类簇,井对合并得到的聚类簇的距离矩阵进行更新;上述过程不断重复,直至达到预设的聚类簇数。
补充
聚类也许是机器学习中"新算法"出现最多、最快的领域一个重要原因是聚类不存在客观标准;给定数据集7 总能从某个角度找到以往算法未覆盖的某种标准从而设计出新算法。相对于机器学习其他分支来说, 聚类的知识还不够系统化。因此著名教科书 [Mitchell,1997] 中甚至没有关于聚类的章节,但聚类技术本身在现实任务中非常重要!!!
聚类性能度量除上面的内容外,常见的还有:
- F 值 互信息(mutual information) 平均廓宽(average silhouette width)等等
距离计算是很多学习任务的核心技术。间可夫斯基距离提供了距离计算的一般形式,除闵可夫斯基距离之外,内积距离、余弦距离等也很常用。
习题
https://www.cnblogs.com/hzcya1995/p/13302681.html

回顾一下性质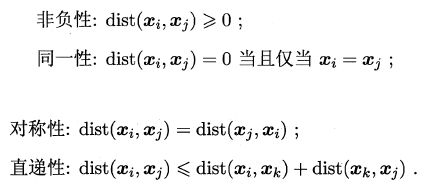
非负性、同一性、对称性很显然都是符合的,关键是直递性了,关于直递性就是闵可夫斯基不等式的证明,具体参考:闵可夫斯基不等式link.

不能,因为 k 均值本身是 NP 问题,而且 9.24 是非凸的(具体证明不太懂),容易陷入局部最优,所以在使用 k 均值时常常多次随机初始化中心点,然后在中心点附近挑选结果最好的一个,即局部最优解,无法找到全局最优解。
import numpy as np
import matplotlib.pyplot as plt
from scipy.spatial import ConvexHull
class KMeans(object):
def __init__(self, k):
self.k = k
def fit(self, X, initial_centroid_index=None, max_iters=10, seed=16, plt_process=False):
m, n = X.shape
# 没有指定中心点时,随机初始化中心点
if initial_centroid_index is None:
np.random.seed(seed)
initial_centroid_index = np.random.randint(0, m, self.k)
centroid = X[initial_centroid_index, :]
idx = None
# 打开交互模式
plt.ion()
for i in range(max_iters):
# 按照中心点给样本分类
idx = self.find_closest_centroids(X, centroid)
if plt_process:
self.plot_converge(X, idx, initial_centroid_index)
# 重新计算中心点
centroid = self.compute_centroids(X, idx)
# 关闭交互模式
plt.ioff()
plt.show()
return centroid, idx
def find_closest_centroids(self, X, centroid):
# 这种方式利用 numpy 的广播机制,直接计算样本到各中心的距离,不用循环,速度比较快,但是在样本比较大时,更消耗内存
distance = np.sum((X[:, np.newaxis, :] - centroid) ** 2, axis=2)
idx = distance.argmin(axis=1)
return idx
def compute_centroids(self, X, idx):
centroids = np.zeros((self.k, X.shape[1]))
for i in range(self.k):
centroids[i, :] = np.mean(X[idx == i], axis=0)
return centroids
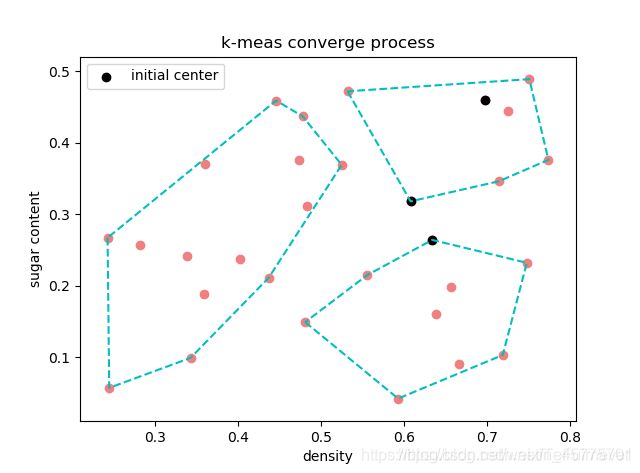
def plot_converge(self, X, idx, initial_idx):
plt.cla() # 清除原有图像
plt.title("k-meas converge process")
plt.xlabel('density')
plt.ylabel('sugar content')
plt.scatter(X[:, 0], X[:, 1], c='lightcoral')
# 标记初始化中心点
plt.scatter(X[initial_idx, 0], X[initial_idx, 1], label='initial center', c='k')
# 画出每个簇的凸包
for i in range(self.k):
X_i = X[idx == i]
# 获取当前簇的凸包索引
hull = ConvexHull(X_i).vertices.tolist()
hull.append(hull[0])
plt.plot(X_i[hull, 0], X_i[hull, 1], 'c--')
plt.legend()
plt.pause(0.5)
if __name__ == '__main__':
data = np.loadtxt('..\data\watermelon4_0_Ch.txt', delimiter=', ')
centroid, idx = KMeans(3).fit(data, plt_process=True, seed=24)
![]()
最小距离由两个簇的最近样本决定,最大距离由两个簇的最远样本决定。具体区别如下:
-
最大距离,可以认为是所有类别首先生成一个能包围所有类内样本的最小圆,然后所有圆同时慢慢扩大相同的半径,直到某个类圆能完全包围另一个类的所有点就停止,并合并这两个类。由于此时的圆已经包含另一个类的全部样本,所以称为全连接。
-
最小距离,可以认为是扩大时遇到第一个非自己类的点就停止,并合并这两个类。由于此时的圆只包含另一个类的一个点,所以称为单连接。
-
原型聚类:输出线性分类边界的聚类算法显然都是凸聚类,这样的算法有:K均值,LVQ;而曲线分类边界的也显然是非凸聚类,高斯混合聚类,在簇间方差不同时,其决策边界为弧线,所以高混合聚类为非凸聚类;
-
密度聚类:DBSCAN是非凸聚类;