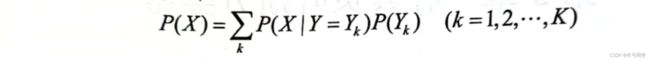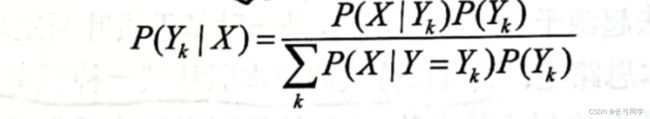数据挖掘学习——朴素贝叶斯分类算法beast cancer实战
目录
1.朴素贝叶斯分类算法相关的统计学知识
2.朴素贝叶斯分类器
3.朴素贝叶斯分类器python实现
(1)调用sklearn库,需要安装
(2)实例1(查看数据的分布情况和数据格式)
(3)实例2(用朴素贝叶斯分类算法对整个breast cancer数据集进行分析,训练得到判断肿瘤是良性还是恶性的模型)
1.查看数据集的特征和标签
2.通过train_test_split()将整个数据集划分为训练集和测试集并查看数据形态
3.用高斯朴素贝叶斯分类算法对训练数据集进行拟合
4.完整高斯朴素贝叶斯分类算法训练breast cancer数据集代码
1.朴素贝叶斯分类算法相关的统计学知识
(1)条件独立公式:P(X,Y)=P(X)*P(Y)
(2)条件概率公式:P(X|Y)=P(X,Y)/P(Y),P(Y|X)=P(X,Y)/P(X)
(3)全概率公式:
(4)贝叶斯公式:
2.朴素贝叶斯分类器
基本思路:假设待分类的样本服从某一种概率分布,首先通过已分类好的样本数据估计某未分类样本的先验概率,然后利用贝叶斯公式计算出未分类样本的后验概率(即预测该样本属于某一类的概率),最后选择具有最大后验概率的类别作为该未分类样本所属的类别。
3.朴素贝叶斯分类器python实现
(1)调用sklearn库,需要安装
数据集说明:使用sklearn库中自带的数据集breast cancer(乳腺癌患者数据:共有569个实例,包括212个良性实例,357个恶性实例。每个实例包括30个属性值,每个属性值取自乳房硬块的细针穿刺数字影像,包括10种特征的平均值和方差。而这10种特征又包括半径、周长和面积等)
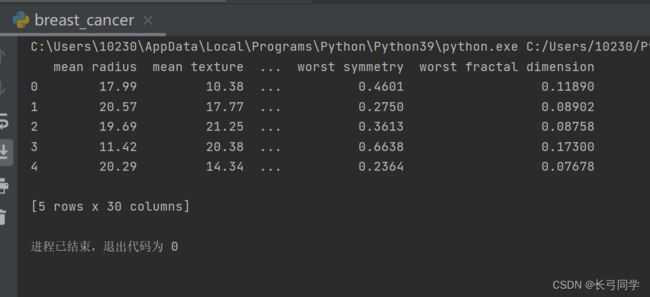
(2)实例1(查看数据的分布情况和数据格式)
实例代码:
import pandas as pd
from sklearn.datasets import load_breast_cancer
cancer=load_breast_cancer()
cancerdf=pd.DataFrame(cancer.data,columns=cancer.feature_names)
print(cancerdf.head()) # head()默认显示前5行数据运行结果(显示前5行数据):
(3)实例2(用朴素贝叶斯分类算法对整个breast cancer数据集进行分析,训练得到判断肿瘤是良性还是恶性的模型)
1.查看数据集的特征和标签
代码:
import pandas as pd
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import confusion_matrix
from matplotlib import pyplot as plt
import seaborn as sns
cancer=load_breast_cancer()
print("肿瘤的分类:",cancer['target_names'])
print("肿瘤的分类:",cancer['feature_names'])运行结果:
2.通过train_test_split()将整个数据集划分为训练集和测试集并查看数据形态
代码:
import pandas as pd
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import confusion_matrix
from matplotlib import pyplot as plt
import seaborn as sns
cancer=load_breast_cancer()
x,y=cancer.data,cancer.target
x_train,x_test,y_train,y_test=train_test_split(x,y,test_size=0.3,random_state=3)
print(x_train.shape)# 查看训练集数据形态
print(x_test.shape)# 查看测试集数据形态
运行结果:
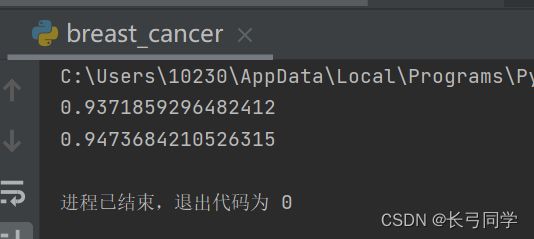
3.用高斯朴素贝叶斯分类算法对训练数据集进行拟合
代码:
clf=GaussianNB()
clf.fit(x_train,y_train)#对训练集进行拟合
print(clf.score(x_train,y_train))
print(clf.score(x_test,y_test))运行结果:
(可以看到,测试集准确率高达0.947)
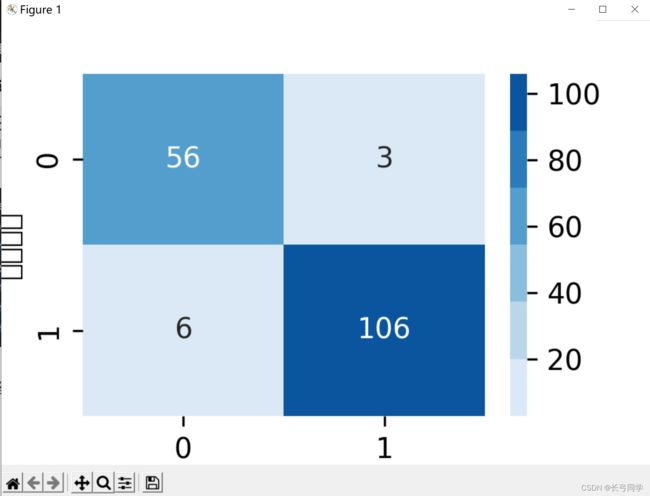
4.绘制混淆矩阵
代码:
pred=clf.predict(x_test)
cm=confusion_matrix(pred,y_test)
plt.figure(dpi=300)
sns.heatmap(cm,cmap=sns.color_palette("Blues"),annot=True,fmt='d')
plt.xlabel('实际类别')
plt.ylabel('预测类别')
plt.show() 运行结果:
4.完整高斯朴素贝叶斯分类算法训练breast cancer数据集代码
对以上的代码进行函数封装并且添加一个将混淆矩阵可视化的函数代码,就可以得到完整训练过程的代码,如下:
(以下代码,是博主按照个人需求写的,也可以根据个人需求进行调整)
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import confusion_matrix
from matplotlib import pyplot as plt
import seaborn as sns
#训练模型函数
def model_fit(x_train,y_train,x_test,y_test):
clf=GaussianNB()
clf.fit(x_train,y_train)#对训练集进行拟合
print(clf.score(x_train,y_train))
print(clf.score(x_test,y_test))
pred=clf.predict(x_test)
cm=confusion_matrix(pred,y_test)
return cm
#混淆矩阵可视化
def matplotlib_show(cm):
plt.figure(dpi=100)#设置窗口大小(分辨率)
plt.title('Confusion Matrix')
labels = ['a', 'b', 'c', 'd']
tick_marks = np.arange(len(labels))
plt.xticks(tick_marks, labels)
plt.yticks(tick_marks, labels)
sns.heatmap(cm, cmap=sns.color_palette("Blues"), annot=True, fmt='d')
plt.ylabel('real_type')#x坐标为实际类别
plt.xlabel('pred_type')#y坐标为预测类别
plt.show()
if __name__ == '__main__':
cancer = load_breast_cancer()
x, y = cancer.data, cancer.target
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.3, random_state=3)
cm=model_fit(x_train,y_train,x_test,y_test)
matplotlib_show(cm)
运行结果: