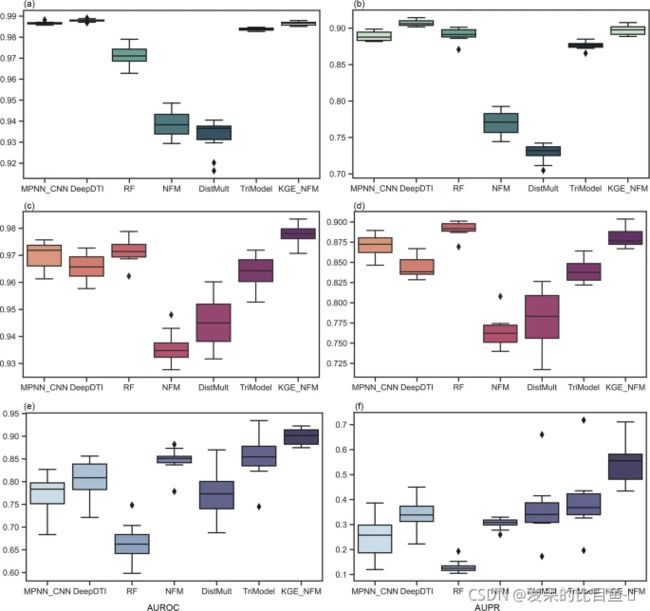
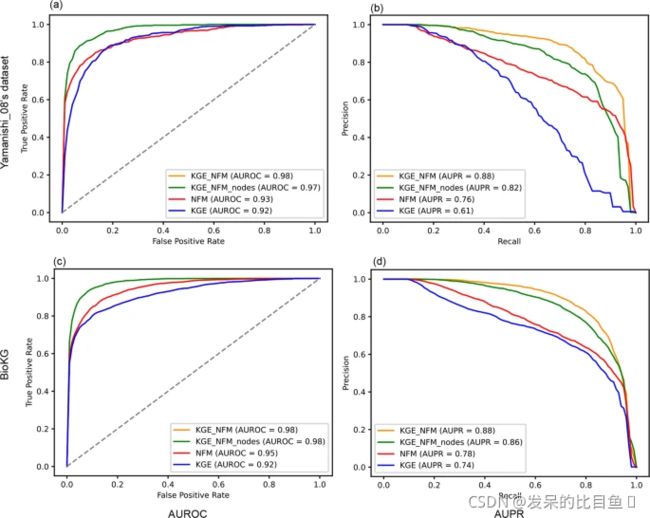
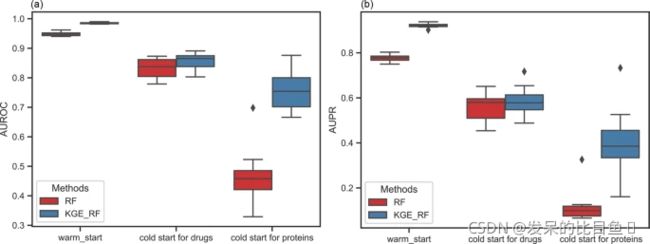
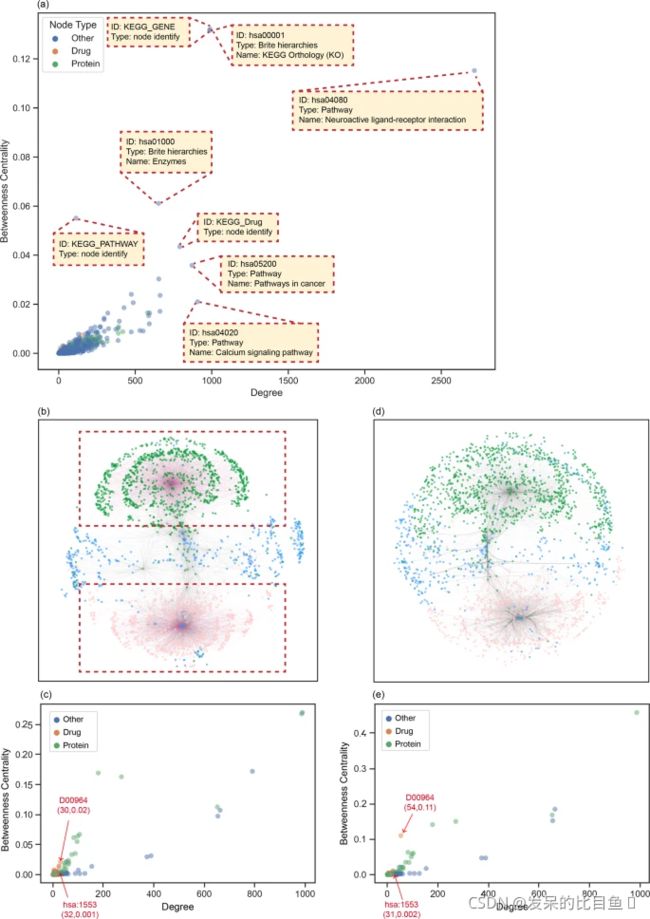
- 高效批量单词翻译工具的设计与应用
本文还有配套的精品资源,点击获取简介:在信息技术飞速发展的今天,批量单词翻译工具通过计算机的数据处理能力,大大提高了语言学习和文字处理的效率。用户通过简单输入单词列表到一个文本文件,并运行翻译程序,即可获得翻译结果并保存至指定文件。该工具集成了内置或外部翻译引擎,利用自然语言处理技术实现快速准确的翻译,并可能提供词性识别等附加功能。尽管机器翻译无法完全取代人工校对,但它为用户提供了一种高效的翻译解
- PyTorch & TensorFlow速成复习:从基础语法到模型部署实战(附FPGA移植衔接)
阿牛的药铺
算法移植部署pytorchtensorflowfpga开发
PyTorch&TensorFlow速成复习:从基础语法到模型部署实战(附FPGA移植衔接)引言:为什么算法移植工程师必须掌握框架基础?针对光学类产品算法FPGA移植岗位需求(如可见光/红外图像处理),深度学习框架是算法落地的"桥梁"——既要用PyTorch/TensorFlow验证算法可行性,又要将训练好的模型(如CNN、目标检测)转换为FPGA可部署的格式(ONNX、TFLite)。本文采用"
- LangChain中的向量数据库接口-Weaviate
洪城叮当
langchain数据库经验分享笔记交互人工智能知识图谱
文章目录前言一、原型定义二、代码解析1、add_texts方法1.1、应用样例2、from_texts方法2.1、应用样例3、similarity_search方法3.1、应用样例三、项目应用1、安装依赖2、引入依赖3、创建对象4、添加数据5、查询数据总结前言 Weaviate是一个开源的向量数据库,支持存储来自各类机器学习模型的数据对象和向量嵌入,并能无缝扩展至数十亿数据对象。它提供存储文档嵌
- 深度学习模型表征提取全解析
ZhangJiQun&MXP
教学2024大模型以及算力2021AIpython深度学习人工智能pythonembedding语言模型
模型内部进行表征提取的方法在自然语言处理(NLP)中,“表征(Representation)”指将文本(词、短语、句子、文档等)转化为计算机可理解的数值形式(如向量、矩阵),核心目标是捕捉语言的语义、语法、上下文依赖等信息。自然语言表征技术可按“静态/动态”“有无上下文”“是否融入知识”等维度划分一、传统静态表征(无上下文,词级为主)这类方法为每个词分配固定向量,不考虑其在具体语境中的含义(无法解
- 【Qualcomm】高通SNPE框架简介、下载与使用
Jackilina_Stone
人工智能QualcommSNPE
目录一高通SNPE框架1SNPE简介2QNN与SNPE3Capabilities4工作流程二SNPE的安装与使用1下载2Setup3SNPE的使用概述一高通SNPE框架1SNPE简介SNPE(SnapdragonNeuralProcessingEngine),是高通公司推出的面向移动端和物联网设备的深度学习推理框架。SNPE提供了一套完整的深度学习推理框架,能够支持多种深度学习模型,包括Pytor
- Python的科学计算库NumPy(一)
linlin_1998
pythonnumpy开发语言
NumPy(NumericalPython)是Python中最基础、最重要的科学计算库之一,提供了高性能的多维数组(ndarray)对象和大量数学函数,是许多数据科学、机器学习库(如Pandas、SciPy、TensorFlow等)的基础依赖。1.创建一个numpy里面的一维数组importnumpyasnp###通过array方法创建一个ndarrayarray1=np.array([1,2,3
- 深度学习篇---昇腾NPU&CANN 工具包
Atticus-Orion
上位机知识篇图像处理篇深度学习篇深度学习人工智能NPU昇腾CANN
介绍昇腾NPU是华为推出的神经网络处理器,具有强大的AI计算能力,而CANN工具包则是面向AI场景的异构计算架构,用于发挥昇腾NPU的性能优势。以下是详细介绍:昇腾NPU架构设计:采用达芬奇架构,是一个片上系统,主要由特制的计算单元、大容量的存储单元和相应的控制单元组成。集成了多个CPU核心,包括控制CPU和AICPU,前者用于控制处理器整体运行,后者承担非矩阵类复杂计算。此外,还拥有AICore
- 深度学习图像分类数据集—桃子识别分类
AI街潜水的八角
深度学习图像数据集深度学习分类人工智能
该数据集为图像分类数据集,适用于ResNet、VGG等卷积神经网络,SENet、CBAM等注意力机制相关算法,VisionTransformer等Transformer相关算法。数据集信息介绍:桃子识别分类:['B1','M2','R0','S3']训练数据集总共有6637张图片,每个文件夹单独放一种数据各子文件夹图片统计:·B1:1601张图片·M2:1800张图片·R0:1601张图片·S3:
- 微算法科技的前沿探索:量子机器学习算法在视觉任务中的革新应用
MicroTech2025
量子计算算法
在信息技术飞速发展的今天,计算机视觉作为人工智能领域的重要分支,正逐步渗透到我们生活的方方面面。从自动驾驶到人脸识别,从医疗影像分析到安防监控,计算机视觉技术展现了巨大的应用潜力。然而,随着视觉任务复杂度的不断提升,传统机器学习算法在处理大规模、高维度数据时遇到了计算瓶颈。在此背景下,量子计算作为一种颠覆性的计算模式,以其独特的并行处理能力和指数级增长的计算空间,为解决这一难题提供了新的思路。微算
- 【AI大模型】LLM模型架构深度解析:BERT vs. GPT vs. T5
我爱一条柴ya
学习AI记录ai人工智能AI编程python
引言Transformer架构的诞生(Vaswanietal.,2017)彻底改变了自然语言处理(NLP)。在其基础上,BERT、GPT和T5分别代表了三种不同的模型范式,主导了预训练语言模型的演进。理解它们的差异是LLM开发和学习的基石。一、核心架构对比特性BERT(BidirectionalEncoder)GPT(GenerativePre-trainedTransformer)T5(Text
- GPT实操——利用GPT创建一个应用
狗木马
深度学习gpt-3gpt
功能描述信息查询:用户可以询问各种问题,如天气、新闻、股票等,机器人会返回相关信息。任务执行:用户可以要求机器人执行一些简单的任务,如设置提醒、发送邮件等。情感支持:机器人可以与用户进行情感交流,提供安慰和支持。个性化设置:用户可以自定义机器人的回复风格和偏好。技术栈前端:React.js后端:Node.js+Express数据库:MongoDB自然语言处理:OpenAIGPT-3API其他工具:
- NumPy-@运算符详解
GG不是gg
numpynumpy
NumPy-@运算符详解一、@运算符的起源与设计目标1.从数学到代码:符号的统一2.设计目标二、@运算符的核心语法与运算规则1.基础用法:二维矩阵乘法2.一维向量的矩阵语义3.高维数组:批次矩阵运算4.广播机制:灵活的形状匹配三、@运算符与其他乘法方式的核心区别1.对比`np.dot()`2.对比元素级乘法`*`3.对比`np.matrix`的`*`运算符四、典型应用场景:从基础到高阶1.深度学习
- Python爬虫实战:使用最新技术爬取新华网新闻数据
Python爬虫项目
2025年爬虫实战项目python爬虫开发语言scrapy音视频
一、前言在当今信息爆炸的时代,网络爬虫技术已经成为获取互联网数据的重要手段。作为国内权威新闻媒体,新华网每天发布大量高质量的新闻内容,这些数据对于舆情分析、市场研究、自然语言处理等领域具有重要价值。本文将详细介绍如何使用Python最新技术构建一个高效、稳定的新华网新闻爬虫系统。二、爬虫技术选型2.1技术栈选择在构建新华网爬虫时,我们选择了以下技术栈:请求库:httpx(支持HTTP/2,异步请求
- NLP_知识图谱_大模型——个人学习记录
macken9999
自然语言处理知识图谱大模型自然语言处理知识图谱学习
1.自然语言处理、知识图谱、对话系统三大技术研究与应用https://github.com/lihanghang/NLP-Knowledge-Graph深度学习-自然语言处理(NLP)-知识图谱:知识图谱构建流程【本体构建、知识抽取(实体抽取、关系抽取、属性抽取)、知识表示、知识融合、知识存储】-元気森林-博客园https://www.cnblogs.com/-402/p/16529422.htm
- 解决 Python 包安装失败问题:以 accelerate 为例
在使用Python开发项目时,我们经常会遇到依赖包安装失败的问题。今天,我们就以accelerate包为例,详细探讨一下可能的原因以及解决方法。通过这篇文章,你将了解到Python包安装失败的常见原因、如何切换镜像源、如何手动安装包,以及一些实用的注意事项。一、问题背景在开发一个深度学习项目时,我需要安装accelerate包来优化模型的训练过程。然而,当我运行以下命令时:bash复制pipins
- 在mac m1基于llama.cpp运行deepseek
lama.cpp是一个高效的机器学习推理库,目标是在各种硬件上实现LLM推断,保持最小设置和最先进性能。llama.cpp支持1.5位、2位、3位、4位、5位、6位和8位整数量化,通过ARMNEON、Accelerate和Metal支持Apple芯片,使得在MACM1处理器上运行Deepseek大模型成为可能。1下载llama.cppgitclonehttps://github.com/ggerg
- 从RNN循环神经网络到Transformer注意力机制:解析神经网络架构的华丽蜕变
熊猫钓鱼>_>
神经网络rnntransformer
1.引言在自然语言处理和序列建模领域,神经网络架构经历了显著的演变。从早期的循环神经网络(RNN)到现代的Transformer架构,这一演变代表了深度学习方法在处理序列数据方面的重大进步。本文将深入比较这两种架构,分析它们的工作原理、优缺点,并通过实验结果展示它们在实际应用中的性能差异。2.循环神经网络(RNN)2.1基本原理循环神经网络是专门为处理序列数据而设计的神经网络架构。RNN的核心思想
- 【机器学习笔记Ⅰ】9 特征缩放
巴伦是只猫
机器学习机器学习笔记人工智能
特征缩放(FeatureScaling)详解特征缩放是机器学习数据预处理的关键步骤,旨在将不同特征的数值范围统一到相近的尺度,从而加速模型训练、提升性能并避免某些特征主导模型。1.为什么需要特征缩放?(1)问题背景量纲不一致:例如:特征1:年龄(范围0-100)特征2:收入(范围0-1,000,000)梯度下降的困境:量纲大的特征(如收入)会导致梯度更新方向偏离最优路径,收敛缓慢。量纲小的特征(如
- 如何使用Python实现交通工具识别
如何使用Python实现交通工具识别文章目录技术架构功能流程识别逻辑用户界面增强特性依赖项主要类别内容展示该系统是一个基于深度学习的交通工具识别工具,具备以下核心功能与特点:技术架构使用预训练的ResNet50卷积神经网络模型(来自ImageNet数据集)集成图像增强预处理技术(随机裁剪、旋转、翻转等)采用多数投票机制提升预测稳定性基于置信度评分的结果筛选策略功能流程用户通过GUI界面选择待识别图
- Python OpenCV教程从入门到精通的全面指南【文末送书】
一键难忘
pythonopencv开发语言
文章目录PythonOpenCV从入门到精通1.安装OpenCV2.基本操作2.1读取和显示图像2.2图像基本操作3.图像处理3.1图像转换3.2图像阈值处理3.3图像平滑4.边缘检测和轮廓4.1Canny边缘检测4.2轮廓检测5.高级操作5.1特征检测5.2目标跟踪5.3深度学习与OpenCVPythonOpenCV从入门到精通【文末送书】PythonOpenCV从入门到精通OpenCV(Ope
- 第八周 tensorflow实现猫狗识别
降花绘
365天深度学习tensorflow系列tensorflow深度学习人工智能
本文为365天深度学习训练营内部限免文章(版权归K同学啊所有)**参考文章地址:[TensorFlow入门实战|365天深度学习训练营-第8周:猫狗识别(训练营内部成员可读)]**作者:K同学啊文章目录一、本周学习内容:1、自己搭建VGG16网络2、了解model.train_on_batch()3、了解tqdm,并使用tqdm实现可视化进度条二、前言三、电脑环境四、前期准备1、导入相关依赖项2、
- 深度学习实战-使用TensorFlow与Keras构建智能模型
程序员Gloria
Python超入门TensorFlowpython
深度学习实战-使用TensorFlow与Keras构建智能模型深度学习已经成为现代人工智能的重要组成部分,而Python则是实现深度学习的主要编程语言之一。本文将探讨如何使用TensorFlow和Keras构建深度学习模型,包括必要的代码实例和详细的解析。1.深度学习简介深度学习是机器学习的一个分支,使用多层神经网络来学习和表示数据中的复杂模式。其广泛应用于图像识别、自然语言处理、推荐系统等领域。
- AI在垂直领域的深度应用:医疗、金融与自动驾驶的革新之路
AI在垂直领域的深度应用:医疗、金融与自动驾驶的革新之路一、医疗领域:AI驱动的精准诊疗与效率提升1.医学影像诊断AI算法通过深度学习技术,已实现对X光、CT、MRI等影像的快速分析,辅助医生检测癌症、骨折等疾病。例如,GoogleDeepMind的AI系统在乳腺癌筛查中,误检率比人类专家低9.4%;中国的推想医疗AI系统可在20秒内完成肺部CT扫描分析,为急诊救治争取黄金时间。2.药物研发传统药
- 专题:2025云计算与AI技术研究趋势报告|附200+份报告PDF、原数据表汇总下载
原文链接:https://tecdat.cn/?p=42935关键词:2025,云计算,AI技术,市场趋势,深度学习,公有云,研究报告云计算和AI技术正以肉眼可见的速度重塑商业世界。过去十年,全球云服务收入激增8倍,中国云计算市场规模突破6000亿元,而深度学习算法的应用量更是暴涨400倍。这些数字背后,是企业从“自建机房”到“云原生开发”的转型,是AI从“实验室”走向“产业级应用”的跨越。本报告
- 【大模型与机器学习解惑】什么是A/B测试,为何进行A/B测试?
以下内容将围绕机器学习中的A/B测试展开,从概念与背景到实施细节、示例代码、优化思路和未来建议,并在最后给出一个整体的“输出目录”供参考。目录什么是机器学习的A/B测试为何要进行A/B测试A/B测试的实施流程示例代码与详细解释优化方向与未来建议结语1.什么是机器学习的A/B测试A/B测试(也常被称作对照试验、SplitTest)最早多用于互联网产品的功能或界面迭代中,指的是将用户或样本随机分为两组
- 【深度学习解惑】在实践中如何发现和修正RNN训练过程中的数值不稳定?
云博士的AI课堂
大模型技术开发与实践哈佛博后带你玩转机器学习深度学习深度学习rnn人工智能tensorflowpytorch神经网络机器学习
在实践中发现和修正RNN训练过程中的数值不稳定目录引言与背景介绍原理解释代码说明与实现应用场景与案例分析实验设计与结果分析性能分析与技术对比常见问题与解决方案创新性与差异性说明局限性与挑战未来建议和进一步研究扩展阅读与资源推荐图示与交互性内容语言风格与通俗化表达互动交流1.引言与背景介绍循环神经网络(RNN)在处理序列数据时表现出色,但训练过程中常面临梯度消失和梯度爆炸问题,导致数值不稳定。当网络
- 【深度学习实战】当前三个最佳图像分类模型的代码详解
云博士的AI课堂
大模型技术开发与实践哈佛博后带你玩转机器学习深度学习深度学习人工智能分类模型机器学习TransformerEfficientNetConvNeXt
下面给出三个在当前图像分类任务中精度表现突出的模型示例,分别基于SwinTransformer、EfficientNet与ConvNeXt。每个模型均包含:训练代码(使用PyTorch)从预训练权重开始微调(也可注释掉预训练选项,从头训练)数据集目录结构:└──dataset_root├──buy#第一类图像└──nobuy#第二类图像随机拆分:80%训练,20%验证每个Epoch输出一次loss
- 第35周—————糖尿病预测模型优化探索
目录目录前言1.检查GPU2.查看数据编辑3.划分数据集4.创建模型与编译训练5.编译及训练模型6.结果可视化7.总结前言本文为365天深度学习训练营中的学习记录博客原作者:K同学啊1.检查GPUimporttorch.nnasnnimporttorch.nn.functionalasFimporttorchvision,torch#设置硬件设备,如果有GPU则使用,没有则使用cpudevice=
- 详解LLMOps,将DevOps用于大语言模型开发
大家好,在机器学习领域,随着技术的不断发展,将大型语言模型(LLMs)集成到商业产品中已成为一种趋势,同时也带来了许多挑战。为了有效应对这些挑战,数据科学家们转向了一种新型的DevOps实践LLM-OPS,专为大型语言模型的开发和维护而设计。本文将介绍LLM-OPS的核心思想,并分析这一策略如何帮助数据科学家更高效地运用DevOps的优秀实践,从而在语言模型的开发和部署过程中,提升工作效率和成果的
- 搜广推校招面经九十一
美团机器学习/数据挖掘算法工程师_二面一、介绍一下ESMM模型,是否有进行过函数推导传统的转化率建模方式:只用发生点击(click=1)的样本来训练CVR模型。CVR定义如下:CVR=P(y=1∣x,z=1)CVR=P(y=1|x,z=1)CVR=P(y=1∣x,z=1)y=1表示用户发生了转化(如购买)z=1表示用户点击了广告这样做的问题:样本选择偏差(SampleSelectionBias,S
- Java常用排序算法/程序员必须掌握的8大排序算法
cugfy
java
分类:
1)插入排序(直接插入排序、希尔排序)
2)交换排序(冒泡排序、快速排序)
3)选择排序(直接选择排序、堆排序)
4)归并排序
5)分配排序(基数排序)
所需辅助空间最多:归并排序
所需辅助空间最少:堆排序
平均速度最快:快速排序
不稳定:快速排序,希尔排序,堆排序。
先来看看8种排序之间的关系:
1.直接插入排序
(1
- 【Spark102】Spark存储模块BlockManager剖析
bit1129
manager
Spark围绕着BlockManager构建了存储模块,包括RDD,Shuffle,Broadcast的存储都使用了BlockManager。而BlockManager在实现上是一个针对每个应用的Master/Executor结构,即Driver上BlockManager充当了Master角色,而各个Slave上(具体到应用范围,就是Executor)的BlockManager充当了Slave角色
- linux 查看端口被占用情况详解
daizj
linux端口占用netstatlsof
经常在启动一个程序会碰到端口被占用,这里讲一下怎么查看端口是否被占用,及哪个程序占用,怎么Kill掉已占用端口的程序
1、lsof -i:port
port为端口号
[root@slave /data/spark-1.4.0-bin-cdh4]# lsof -i:8080
COMMAND PID USER FD TY
- Hosts文件使用
周凡杨
hostslocahost
一切都要从localhost说起,经常在tomcat容器起动后,访问页面时输入http://localhost:8088/index.jsp,大家都知道localhost代表本机地址,如果本机IP是10.10.134.21,那就相当于http://10.10.134.21:8088/index.jsp,有时候也会看到http: 127.0.0.1:
- java excel工具
g21121
Java excel
直接上代码,一看就懂,利用的是jxl:
import java.io.File;
import java.io.IOException;
import jxl.Cell;
import jxl.Sheet;
import jxl.Workbook;
import jxl.read.biff.BiffException;
import jxl.write.Label;
import
- web报表工具finereport常用函数的用法总结(数组函数)
老A不折腾
finereportweb报表函数总结
ADD2ARRAY
ADDARRAY(array,insertArray, start):在数组第start个位置插入insertArray中的所有元素,再返回该数组。
示例:
ADDARRAY([3,4, 1, 5, 7], [23, 43, 22], 3)返回[3, 4, 23, 43, 22, 1, 5, 7].
ADDARRAY([3,4, 1, 5, 7], "测试&q
- 游戏服务器网络带宽负载计算
墙头上一根草
服务器
家庭所安装的4M,8M宽带。其中M是指,Mbits/S
其中要提前说明的是:
8bits = 1Byte
即8位等于1字节。我们硬盘大小50G。意思是50*1024M字节,约为 50000多字节。但是网宽是以“位”为单位的,所以,8Mbits就是1M字节。是容积体积的单位。
8Mbits/s后面的S是秒。8Mbits/s意思是 每秒8M位,即每秒1M字节。
我是在计算我们网络流量时想到的
- 我的spring学习笔记2-IoC(反向控制 依赖注入)
aijuans
Spring 3 系列
IoC(反向控制 依赖注入)这是Spring提出来了,这也是Spring一大特色。这里我不用多说,我们看Spring教程就可以了解。当然我们不用Spring也可以用IoC,下面我将介绍不用Spring的IoC。
IoC不是框架,她是java的技术,如今大多数轻量级的容器都会用到IoC技术。这里我就用一个例子来说明:
如:程序中有 Mysql.calss 、Oracle.class 、SqlSe
- 高性能mysql 之 选择存储引擎(一)
annan211
mysqlInnoDBMySQL引擎存储引擎
1 没有特殊情况,应尽可能使用InnoDB存储引擎。 原因:InnoDB 和 MYIsAM 是mysql 最常用、使用最普遍的存储引擎。其中InnoDB是最重要、最广泛的存储引擎。她 被设计用来处理大量的短期事务。短期事务大部分情况下是正常提交的,很少有回滚的情况。InnoDB的性能和自动崩溃 恢复特性使得她在非事务型存储的需求中也非常流行,除非有非常
- UDP网络编程
百合不是茶
UDP编程局域网组播
UDP是基于无连接的,不可靠的传输 与TCP/IP相反
UDP实现私聊,发送方式客户端,接受方式服务器
package netUDP_sc;
import java.net.DatagramPacket;
import java.net.DatagramSocket;
import java.net.Ine
- JQuery对象的val()方法执行结果分析
bijian1013
JavaScriptjsjquery
JavaScript中,如果id对应的标签不存在(同理JAVA中,如果对象不存在),则调用它的方法会报错或抛异常。在实际开发中,发现JQuery在id对应的标签不存在时,调其val()方法不会报错,结果是undefined。
- http请求测试实例(采用json-lib解析)
bijian1013
jsonhttp
由于fastjson只支持JDK1.5版本,因些对于JDK1.4的项目,可以采用json-lib来解析JSON数据。如下是http请求的另外一种写法,仅供参考。
package com;
import java.util.HashMap;
import java.util.Map;
import
- 【RPC框架Hessian四】Hessian与Spring集成
bit1129
hessian
在【RPC框架Hessian二】Hessian 对象序列化和反序列化一文中介绍了基于Hessian的RPC服务的实现步骤,在那里使用Hessian提供的API完成基于Hessian的RPC服务开发和客户端调用,本文使用Spring对Hessian的集成来实现Hessian的RPC调用。
定义模型、接口和服务器端代码
|---Model
&nb
- 【Mahout三】基于Mahout CBayes算法的20newsgroup流程分析
bit1129
Mahout
1.Mahout环境搭建
1.下载Mahout
http://mirror.bit.edu.cn/apache/mahout/0.10.0/mahout-distribution-0.10.0.tar.gz
2.解压Mahout
3. 配置环境变量
vim /etc/profile
export HADOOP_HOME=/home
- nginx负载tomcat遇非80时的转发问题
ronin47
nginx负载后端容器是tomcat(其它容器如WAS,JBOSS暂没发现这个问题)非80端口,遇到跳转异常问题。解决的思路是:$host:port
详细如下:
该问题是最先发现的,由于之前对nginx不是特别的熟悉所以该问题是个入门级别的:
? 1 2 3 4 5
- java-17-在一个字符串中找到第一个只出现一次的字符
bylijinnan
java
public class FirstShowOnlyOnceElement {
/**Q17.在一个字符串中找到第一个只出现一次的字符。如输入abaccdeff,则输出b
* 1.int[] count:count[i]表示i对应字符出现的次数
* 2.将26个英文字母映射:a-z <--> 0-25
* 3.假设全部字母都是小写
*/
pu
- mongoDB 复制集
开窍的石头
mongodb
mongo的复制集就像mysql的主从数据库,当你往其中的主复制集(primary)写数据的时候,副复制集(secondary)会自动同步主复制集(Primary)的数据,当主复制集挂掉以后其中的一个副复制集会自动成为主复制集。提供服务器的可用性。和防止当机问题
mo
- [宇宙与天文]宇宙时代的经济学
comsci
经济
宇宙尺度的交通工具一般都体型巨大,造价高昂。。。。。
在宇宙中进行航行,近程采用反作用力类型的发动机,需要消耗少量矿石燃料,中远程航行要采用量子或者聚变反应堆发动机,进行超空间跳跃,要消耗大量高纯度水晶体能源
以目前地球上国家的经济发展水平来讲,
- Git忽略文件
Cwind
git
有很多文件不必使用git管理。例如Eclipse或其他IDE生成的项目文件,编译生成的各种目标或临时文件等。使用git status时,会在Untracked files里面看到这些文件列表,在一次需要添加的文件比较多时(使用git add . / git add -u),会把这些所有的未跟踪文件添加进索引。
==== ==== ==== 一些牢骚
- MySQL连接数据库的必须配置
dashuaifu
mysql连接数据库配置
MySQL连接数据库的必须配置
1.driverClass:com.mysql.jdbc.Driver
2.jdbcUrl:jdbc:mysql://localhost:3306/dbname
3.user:username
4.password:password
其中1是驱动名;2是url,这里的‘dbna
- 一生要养成的60个习惯
dcj3sjt126com
习惯
一生要养成的60个习惯
第1篇 让你更受大家欢迎的习惯
1 守时,不准时赴约,让别人等,会失去很多机会。
如何做到:
①该起床时就起床,
②养成任何事情都提前15分钟的习惯。
③带本可以随时阅读的书,如果早了就拿出来读读。
④有条理,生活没条理最容易耽误时间。
⑤提前计划:将重要和不重要的事情岔开。
⑥今天就准备好明天要穿的衣服。
⑦按时睡觉,这会让按时起床更容易。
2 注重
- [介绍]Yii 是什么
dcj3sjt126com
PHPyii2
Yii 是一个高性能,基于组件的 PHP 框架,用于快速开发现代 Web 应用程序。名字 Yii (读作 易)在中文里有“极致简单与不断演变”两重含义,也可看作 Yes It Is! 的缩写。
Yii 最适合做什么?
Yii 是一个通用的 Web 编程框架,即可以用于开发各种用 PHP 构建的 Web 应用。因为基于组件的框架结构和设计精巧的缓存支持,它特别适合开发大型应
- Linux SSH常用总结
eksliang
linux sshSSHD
转载请出自出处:http://eksliang.iteye.com/blog/2186931 一、连接到远程主机
格式:
ssh name@remoteserver
例如:
ssh
[email protected]
二、连接到远程主机指定的端口
格式:
ssh name@remoteserver -p 22
例如:
ssh i
- 快速上传头像到服务端工具类FaceUtil
gundumw100
android
快速迭代用
import java.io.DataOutputStream;
import java.io.File;
import java.io.FileInputStream;
import java.io.FileNotFoundException;
import java.io.FileOutputStream;
import java.io.IOExceptio
- jQuery入门之怎么使用
ini
JavaScripthtmljqueryWebcss
jQuery的强大我何问起(个人主页:hovertree.com)就不用多说了,那么怎么使用jQuery呢?
首先,下载jquery。下载地址:http://hovertree.com/hvtart/bjae/b8627323101a4994.htm,一个是压缩版本,一个是未压缩版本,如果在开发测试阶段,可以使用未压缩版本,实际应用一般使用压缩版本(min)。然后就在页面上引用。
- 带filter的hbase查询优化
kane_xie
查询优化hbaseRandomRowFilter
问题描述
hbase scan数据缓慢,server端出现LeaseException。hbase写入缓慢。
问题原因
直接原因是: hbase client端每次和regionserver交互的时候,都会在服务器端生成一个Lease,Lease的有效期由参数hbase.regionserver.lease.period确定。如果hbase scan需
- java设计模式-单例模式
men4661273
java单例枚举反射IOC
单例模式1,饿汉模式
//饿汉式单例类.在类初始化时,已经自行实例化
public class Singleton1 {
//私有的默认构造函数
private Singleton1() {}
//已经自行实例化
private static final Singleton1 singl
- mongodb 查询某一天所有信息的3种方法,根据日期查询
qiaolevip
每天进步一点点学习永无止境mongodb纵观千象
// mongodb的查询真让人难以琢磨,就查询单天信息,都需要花费一番功夫才行。
// 第一种方式:
coll.aggregate([
{$project:{sendDate: {$substr: ['$sendTime', 0, 10]}, sendTime: 1, content:1}},
{$match:{sendDate: '2015-
- 二维数组转换成JSON
tangqi609567707
java二维数组json
原文出处:http://blog.csdn.net/springsen/article/details/7833596
public class Demo {
public static void main(String[] args) { String[][] blogL
- erlang supervisor
wudixiaotie
erlang
定义supervisor时,如果是监控celuesimple_one_for_one则删除children的时候就用supervisor:terminate_child (SupModuleName, ChildPid),如果shutdown策略选择的是brutal_kill,那么supervisor会调用exit(ChildPid, kill),这样的话如果Child的behavior是gen_