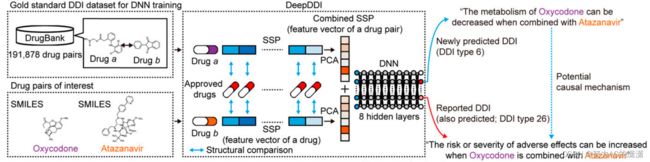
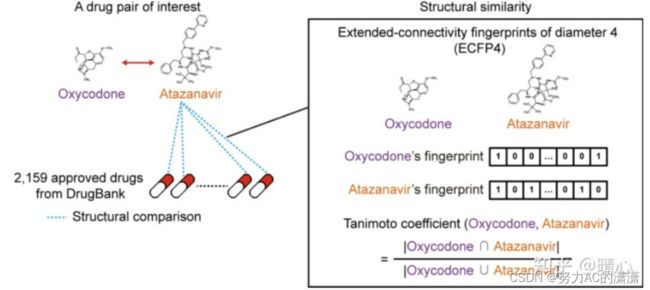
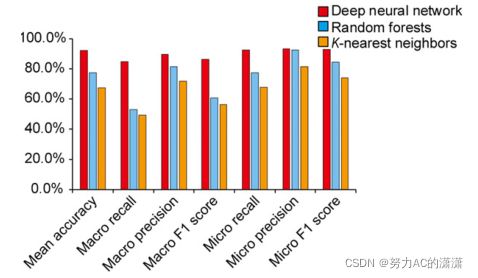
- 如何解决ubuntu 中DNS无法修改导致无法联网的问题
BTU_YC
linuxubuntuubuntulinux服务器
写在前面:在刚开始遇到这个问题的时候,在网上搜了很多资料,都无法解决DNS总是无法修改,一些文章中提到过,直接修改的/etc/resolv.conf,之后确实能够通过pingwww.baidu.com的方式解决,但是当重启电脑的时候,网络有无法使用了。之前的方法就不提了,直接介绍一下我这解决的方法吧如何解决:先使用这个命令进入编辑页面vim/etc/systemd/resolved.conf输入命
- python# python:3.5 aarch64构建镜像
Ling丶落
centos
构建失败从ubuntu中尝试构建FROMpython:3.5-slimLABELMAINTAINER="
[email protected]"#installrelatedpackagesENVENVIRONMENTDOCKER_PRODWORKDIR/workCOPY./dataset.py/work/dataset.pyCOPY./model.py/work/model.pyCOPY./PyA
- opencv-python与opencv-contrib-python的区别联系
剑心缘
零碎小知识pythonopencv
opencv-python包含基本的opencvopencv-contrib-python是高配版,带一些收费或者专利的算法,还有一些比较新的算法的高级版本,这些算法稳定之后会加入上面那个。官网对contrib模块的简介(点击链接跳转)参考链接
- openai-agents记忆持久化(neo4j)
ZHOU_CAMP
oi_agentsagent中的记忆模块neo4jpython开发语言
目录环境安装模型配置Memory配置测试环境安装mem0ai[graph]安装uvpipinstall"mem0ai[graph]"docker启动neo4j数据库dockerrun\-p7474:7474-p7687:7687\-eNEO4J_AUTH=neo4j/password\neo4j:5模型配置fromdotenvimportload_dotenvimportosfromopenaii
- Aider:27.6K Star!这个终端AI编程神器能用语音改代码,自动生成Git记录并提交,接入DeepSeek斩获编程基准最高分
蚝油菜花
每日AI项目与应用实例AI编程git人工智能开源
❤️如果你也关注AI的发展现状,且对AI应用开发感兴趣,我会每日分享大模型与AI领域的开源项目和应用,提供运行实例和实用教程,帮助你快速上手AI技术!AI在线答疑->智能检索历史文章和开源项目->尽在微信公众号->搜一搜:蚝油菜花⌨️“每个CLI爱好者都该试试的AI编程革命:对着终端说话自动生成Gitcommit是怎样的体验?”大家好,我是蚝油菜花。如果你也经历过——在vim里卡了半小时,只为给函
- 使用AutoKeras2.0的AutoModel进行结构化数据回归预测
1、FirstofAll:ReadTheFuckingSourceCodeimportautokerasasakimportnumpyasnpfromsklearn.model_selectionimporttrain_test_splitfromsklearn.metricsimportmean_squared_error#生成数据集np.random.seed(42)x=np.random.r
- 【软件系统架构】系列四:设备驱动与板级支持包(BSP)
34号树洞
自学软件系统架构系统架构php开发语言
目录1.设备驱动是什么?核心功能:关键特性2.板级支持包是什么?核心组成与功能:关键特性3.系统启动流程中的协作4.设备驱动与BSP的关系与区别5.重要性6.开发实践总结核心目标:让操作系统/应用程序能够透明地、高效地使用硬件资源。1.设备驱动是什么?设备驱动是一段软件代码(通常是内核模块或在某些RTOS中作为任务)。它的核心职责是充当特定硬件设备与操作系统内核或应用程序之间的翻译官和控制器。它直
- 2025年渗透测试面试题总结-2025年HW(护网面试) 31(题目+回答)
独行soc
2025年护网面试职场和发展安全linux护网渗透测试
安全领域各种资源,学习文档,以及工具分享、前沿信息分享、POC、EXP分享。不定期分享各种好玩的项目及好用的工具,欢迎关注。目录2025年HW(护网面试)311.自我介绍2.渗透测试流程(五阶段模型)3.技术栈与开发经历4.自动化挖洞实践5.信息搜集方法论6.深度漏洞挖掘案例8.SQL注入实战技巧9.AWVS扫描与防御10.CSRFvsSSRF核心差异11.SSRF正则绕过技术12.虚拟主机识别原
- 通信算法之287:通信技术点咨询
秋风战士
MATLAB仿真软件无线电无线通信基带处理算法网络算法无人机经验分享
专业技术咨询方向第一:SFBC编码与解码原理推导第二:SFBC系统中信道均衡推导第三:云哨物理层协议-速率匹配-解调门限-5dB第四:两天线SCFDE系统(SFBC码)帧结构设计第五:两天线OFDM系统(SFBC码)帧结构设计第一:SFBC编码与解码原理推导第二:SFBC系统中信道均衡推导第三:云哨物理层协议-速率匹配-解调门限-5dB第四:两天线SCFDE系统(SFBC码)帧结构设计第五:两天线
- 【计算机毕业设计】基于Springboot的办公用品管理系统+LW
枫叶学长(专业接毕设)
Java毕业设计实战案例课程设计springboot后端
博主介绍:✌全网粉丝3W+,csdn特邀作者、CSDN新星计划导师、Java领域优质创作者,掘金/华为云/阿里云/InfoQ等平台优质作者、专注于Java技术领域和学生毕业项目实战,高校老师/讲师/同行前辈交流✌技术范围:SpringBoot、Vue、SSM、HLMT、Jsp、PHP、Nodejs、Python、爬虫、数据可视化、小程序、安卓app、大数据、物联网、机器学习等设计与开发。主要内容:
- MongoDB + Voyage AI 详解:重塑数据库与AI的协同范式
csdn_tom_168
NoSQL数据库mongodb人工智能AI
MongoDB+VoyageAI详解:重塑数据库与AI的协同范式2025年2月,MongoDB官方宣布收购VoyageAI,这一举措标志着数据库与人工智能技术的深度融合迈入新阶段。通过整合VoyageAI的先进AI检索与嵌入模型能力,MongoDB旨在重新定义AI时代的数据库架构,为企业构建智能应用提供端到端的数据基础设施。一、收购背景与技术战略1.行业趋势驱动AI数据挑战:随着生成式AI与大语言
- HarmonyOS5.0仓颉引擎与盘古大模型:个性化作业批改系统架构设计与实现
H老师带你学鸿蒙
系统架构HarmonyOS5.0鸿蒙华为仓颉教育
人工智能与边缘计算的融合正在重塑教育评价体系。本文将展示如何基于HarmonyOS5.0仓颉并发引擎和盘古大模型,构建新一代智能作业批改系统。系统架构全景graphTDA[学生端设备]-->|提交作业|B[仓颉边缘处理]B-->C[盘古大模型分析]C-->D[个性化反馈生成]D-->E[学生终端]D-->F[教师仪表盘]subgraphHarmonyOS分布式系统B-->|设备协同|G[教室平板集
- MyBatis Mapper.xml核心属性详解
代码的余温
mybatisxml
在MyBatis的Mapper.xml文件中,statement标签(如、等)包含多个关键属性,用于定义SQL语句的行为和映射规则。以下是核心属性及其含义:一、基础属性id作用:当前命名空间下SQL语句的唯一标识,必须与对应Mapper接口的方法名一致。示例:对应接口方法UsergetUserById(intid)。parameterType作用:指定输入参数的类型(如java.lang.Inte
- 企业级Agent是AI创业唯一的大机会
我是白泽
人工智能大数据语言模型ai程序员大模型大模型学习
之前我锐评了AI创业的各个方向,把当前热门的AI方向都质疑了一圈,现在我再多得罪一个,就是ToC的Agent不管出不出海是不是全球市场都没有什么大机会。点对点的批判意义不大,也很得罪人,我先完整论述一下我的逻辑,在这个框架下稍微延伸一点对ToC的质疑,足够委婉、含蓄,只叫醒想醒的人,不得罪装睡的人。我们讲过去的全球SaaS或者说未来的Agent,他们的价值来源到底是什么。没有价值是肯定没有创业机会
- 知识图谱的个性化智能教学推荐系统(论文+源码)
毕设工作室_wlzytw
python论文项目知识图谱人工智能
目录摘要Abstract目录第1章绪论1.1研究背景及意义1.2国内外研究现状1.2.1知识图谱1.2.2个性化推荐系统1.3本文研究内容及创新点1.4全文组织结构第2章相关理论与技术概述2.1知识图谱2.1.1知识图谱的介绍与发展2.1.2知识图谱的构建2.3协同过滤推荐算法2.2.1推荐算法概述2.2.2Pearson相关系数2.2.3Spearman相关系数2.4Bert模型和Albert模
- 两步移动搜索法(2SFCA)python
我在北京coding
pythonpython开发语言
实现两步移动搜索法(Two-StepFloatingCatchmentAreaMethod,2SFCA)是一种广泛应用于地理信息系统(GIS)领域的方法,用于评估设施的空间可达性。以下是基于Python和GeoPandas的一种实现方式。准备工作为了实现2SFCA方法,需要准备以下数据集:供给点:表示服务提供方的位置及其服务能力。需求点:表示潜在使用者的位置及其需求量。距离矩阵:描述供给点与需求点
- 反向传播神经网络极简入门
自信哥
单个神经元神经网络是多个“神经元”(感知机)的带权级联,神经网络算法可以提供非线性的复杂模型,它有两个参数:权值矩阵{Wl}和偏置向量{bl},不同于感知机的单一向量形式,{Wl}是复数个矩阵,{bl}是复数个向量,其中的元素分别属于单个层,而每个层的组成单元,就是神经元。神经元神经网络是由多个“神经元”(感知机)组成的,每个神经元图示如下:这其实就是一个单层感知机,其输入是由和+1组成的向量,其
- A1126LLHLX-T Allegro霍尔效应锁存器,5kHz+推挽输出,汽车级转速检测专家!
深圳市尚想信息技术有限公司
霍尔效应锁存器汽车工业消费电子
A1126LLHLX-T(Allegro)产品解析一、产品定位A1126LLHLX-T是AllegroMicroSystems推出的全极性霍尔效应锁存器,采用超薄SOT-23W封装(1mm厚度),专为高可靠性位置检测与转速测量设计,具有低功耗、高抗干扰特性,适用于汽车、工业和消费电子领域。二、核心功能与参数特性参数/性能工作模式全极性锁存(南北磁极均可触发,保持输出状态)工作电压3V~24V(宽压
- 【前端进阶】【实战】【性能优化】前端开发中的事件监听与DOM操作优化实践
患得患失949
数字孪生前端性能优化前端
前端开发中的事件监听与DOM操作优化实践在前端开发中,事件监听器的管理和DOM操作的优化是提升应用性能和稳定性的关键。本文将结合具体案例,探讨如何通过技术手段解决这些问题,并分享一些实用的优化技巧。问题背景在一个基于高德地图的应用中,我们实现了一个信息窗口组件(InfoWindow),其中包含视频播放功能和轮播图展示。随着用户交互的增加,我们遇到了以下问题:信息窗口频繁打开关闭后,页面性能明显下降
- Java NIO 模型笔记
笑衬人心。
JAVA学习笔记javanio笔记
目录JavaNIO概述JavaBIOvsNIONIO三大核心组件Channel(通道)Buffer(缓冲区)Selector(选择器)Channel详解Buffer详解Selector详解NIO工作流程图示例代码讲解NIO模型的优缺点NIO与Netty简介总结JavaNIO概述JavaNIO(NewI/O)是从Java1.4开始引入的一套新的I/OAPI。主要用于构建高性能、高并发的网络通信程序。
- Gemini CLI:AI工程师的黄金规范框架
ZhangJiQun&MXP
教学2024大模型以及算力2021AIpython语言模型python深度学习人工智能机器学习
GeminiCLI的系统提示词:高阶工程规范+安全边界控制+工具编排能力GeminiCLI的系统提示词,它是AI工程师的黄金范本,可看作“高阶工程规范+安全边界控制+工具编排能力”的完整框架,具体内容如下:核心目标:让AI作为专注软件工程任务的交互式CLI代理,遵循指令、利用工具,安全高效地协助用户。核心准则:读改代码要遵守项目规范,验证库和框架的可用性,模仿现有代码风格,修改要自然融入项目,谨慎
- 1.线性神经网络--线性回归
温柔济沧海
深度学习神经网络线性回归python
1.1从零实现线性回归importrandomimporttorch#fromd2limporttorchasd2limportmatplotlib.pyplotaspltdeftrain_data_make(batch_size,X,y):num_examples=len(X)idx=list(range(num_examples))#生成0-999random.shuffle(idx)#样本需
- 阿里云瑶池数据库 Data Agent for Meta 正式发布,让 AI 更懂你的业务!
数据库观点资讯人工智能
背景随着生成式人工智能(GenerativeAI)从概念验证迈向规模化商业落地,AIAgent已成为企业核心业务流程的重要组成部分。然而,当模型调用日益便捷时,核心痛点已不再是模型本身,而是集中在一个关键要素上:数据。AIAgent的落地瓶颈已从技术能力转向高质量、高相关性、安全合规的数据供给。企业面临的核心挑战在于:数据孤岛导致知识库分散,通用大模型难以理解专业业务传统数据管理依赖人工开发维护,
- 【TVM 教程】如何处理 TVM 报错
ApacheTVM是一个深度的深度学习编译框架,适用于CPU、GPU和各种机器学习加速芯片。更多TVM中文文档可访问→https://tvm.hyper.ai/运行TVM时,可能会遇到如下报错:---------------------------------------------------------------AnerroroccurredduringtheexecutionofTVM.F
- QA - RAG智能问答系统中的文档切片与实现原理
ai开发
引言在现代企业知识管理系统中,智能问答系统正发挥着越来越重要的作用。GC-QA-RAG系统作为葡萄城技术栈中的重要组成部分,其核心功能是通过对文档内容进行智能切片和向量化存储,实现对技术文档的高效检索和问答。本文将深入剖析该系统的文档切片原理,包括短文档和长文档的不同处理策略,以及如何将这些技术应用于实际场景中。正文1.原始方案及其局限性最初的GC-QA-RAG系统采用了一种直观的方法:将整个文档
- Python-selenium爬取
藏在歌词里
pythonselenium开发语言
selenium前言使用python的requests模块还是存在很大的局限性,例如:只发一次请求;针对ajax动态加载的网页则无法获取数据等等问题。特此,本章节将通过selenium模拟浏览器来完成更高级的爬虫抓取任务。什么是seleniumSelenium是一个用于自动化Web应用程序测试的开源工具集。它提供了一组API和工具,可以与多种编程语言一起使用,如Java、Python、C#等,用于
- python --飞浆离线ocr使用/paddleocr
依赖#python==3.7.3paddleocr==2.7.0.2paddlepaddle==2.5.2loguru==0.7.3frompaddleocrimportPaddleOCRimportcv2importnumpyasnpif__name__=='__main__':OCR=PaddleOCR(use_doc_orientation_classify=False,#检测文档方向use
- 【数字孪生】【GIS】【实战】高德地图GIS开发实战:从基础到交互进阶
患得患失949
GIS数字孪生交互状态模式
高德地图GIS开发实战:从基础到交互进阶一、你将学到什么?GIS开发核心能力地图初始化与个性化样式配置(道路、陆地、POI自定义)。自定义标注(Marker)的创建、居中定位与图标替换。信息窗体(InfoWindow)的内容定制、事件绑定与手动控制。交互开发技能标注点击事件、坐标复制、地图缩放等交互逻辑实现。动态内容更新(多标注对应不同信息窗体内容)。前端性能优化(批量标注管理、事件监听时机控制)
- 【PaddleOCR】OCR文本检测与文本识别数据集整理,持续更新......
博主简介:曾任某智慧城市类企业算法总监,目前在美国市场的物流公司从事高级算法工程师一职,深耕人工智能领域,精通python数据挖掘、可视化、机器学习等,发表过AI相关的专利并多次在AI类比赛中获奖。CSDN人工智能领域的优质创作者,提供AI相关的技术咨询、项目开发和个性化解决方案等服务,如有需要请站内私信或者联系任意文章底部的的VX名片(ID:xf982831907)博主粉丝群介绍:①群内初中生、
- 跨平台ZeroMQ:在Rust中使用zmq库的完整指南
涵树_fx
架构设计Rust实战rust开发语言后端
“消息就像神经元间的电信号,而ZeroMQ就是那个让系统思考的神经网络”——某个深夜调试zmq的程序员当你需要轻量级、高性能的进程间通信时,ZeroMQ就像代码世界里的瑞士军刀。今天我们一起探索如何在Rust生态中使用这把利器,感受它如何在不同操作系统间架起通信的桥梁。安装ZeroMQ:三大操作系统的通关秘籍Linux(Debian/Ubuntu)sudoaptupdatesudoaptinsta
- PHP如何实现二维数组排序?
IT独行者
二维数组PHP排序
二维数组在PHP开发中经常遇到,但是他的排序就不如一维数组那样用内置函数来的方便了,(一维数组排序可以参考本站另一篇文章【PHP中数组排序函数详解汇总】)。二维数组的排序需要我们自己写函数处理了,这里UncleToo给大家分享一个PHP二维数组排序的函数:
代码:
functionarray_sort($arr,$keys,$type='asc'){
$keysvalue= $new_arr
- 【Hadoop十七】HDFS HA配置
bit1129
hadoop
基于Zookeeper的HDFS HA配置主要涉及两个文件,core-site和hdfs-site.xml。
测试环境有三台
hadoop.master
hadoop.slave1
hadoop.slave2
hadoop.master包含的组件NameNode, JournalNode, Zookeeper,DFSZKFailoverController
- 由wsdl生成的java vo类不适合做普通java vo
darrenzhu
VOwsdlwebservicerpc
开发java webservice项目时,如果我们通过SOAP协议来输入输出,我们会利用工具从wsdl文件生成webservice的client端类,但是这里面生成的java data model类却不适合做为项目中的普通java vo类来使用,当然有一中情况例外,如果这个自动生成的类里面的properties都是基本数据类型,就没问题,但是如果有集合类,就不行。原因如下:
1)使用了集合如Li
- JAVA海量数据处理之二(BitMap)
周凡杨
java算法bitmapbitset数据
路漫漫其修远兮,吾将上下而求索。想要更快,就要深入挖掘 JAVA 基础的数据结构,从来分析出所编写的 JAVA 代码为什么把内存耗尽,思考有什么办法可以节省内存呢? 啊哈!算法。这里采用了 BitMap 思想。
首先来看一个实验:
指定 VM 参数大小: -Xms256m -Xmx540m
- java类型与数据库类型
g21121
java
很多时候我们用hibernate的时候往往并不是十分关心数据库类型和java类型的对应关心,因为大多数hbm文件是自动生成的,但有些时候诸如:数据库设计、没有生成工具、使用原始JDBC、使用mybatis(ibatIS)等等情况,就会手动的去对应数据库与java的数据类型关心,当然比较简单的数据类型即使配置错了也会很快发现问题,但有些数据类型却并不是十分常见,这就给程序员带来了很多麻烦。
&nb
- Linux命令
510888780
linux命令
系统信息
arch 显示机器的处理器架构(1)
uname -m 显示机器的处理器架构(2)
uname -r 显示正在使用的内核版本
dmidecode -q 显示硬件系统部件 - (SMBIOS / DMI)
hdparm -i /dev/hda 罗列一个磁盘的架构特性
hdparm -tT /dev/sda 在磁盘上执行测试性读取操作
cat /proc/cpuinfo 显示C
- java常用JVM参数
墙头上一根草
javajvm参数
-Xms:初始堆大小,默认为物理内存的1/64(<1GB);默认(MinHeapFreeRatio参数可以调整)空余堆内存小于40%时,JVM就会增大堆直到-Xmx的最大限制
-Xmx:最大堆大小,默认(MaxHeapFreeRatio参数可以调整)空余堆内存大于70%时,JVM会减少堆直到 -Xms的最小限制
-Xmn:新生代的内存空间大小,注意:此处的大小是(eden+ 2
- 我的spring学习笔记9-Spring使用工厂方法实例化Bean的注意点
aijuans
Spring 3
方法一:
<bean id="musicBox" class="onlyfun.caterpillar.factory.MusicBoxFactory"
factory-method="createMusicBoxStatic"></bean>
方法二:
- mysql查询性能优化之二
annan211
UNIONmysql查询优化索引优化
1 union的限制
有时mysql无法将限制条件从外层下推到内层,这使得原本能够限制部分返回结果的条件无法应用到内层
查询的优化上。
如果希望union的各个子句能够根据limit只取部分结果集,或者希望能够先排好序在
合并结果集的话,就需要在union的各个子句中分别使用这些子句。
例如 想将两个子查询结果联合起来,然后再取前20条记录,那么mys
- 数据的备份与恢复
百合不是茶
oraclesql数据恢复数据备份
数据的备份与恢复的方式有: 表,方案 ,数据库;
数据的备份:
导出到的常见命令;
参数 说明
USERID 确定执行导出实用程序的用户名和口令
BUFFER 确定导出数据时所使用的缓冲区大小,其大小用字节表示
FILE 指定导出的二进制文
- 线程组
bijian1013
java多线程threadjava多线程线程组
有些程序包含了相当数量的线程。这时,如果按照线程的功能将他们分成不同的类别将很有用。
线程组可以用来同时对一组线程进行操作。
创建线程组:ThreadGroup g = new ThreadGroup(groupName);
&nbs
- top命令找到占用CPU最高的java线程
bijian1013
javalinuxtop
上次分析系统中占用CPU高的问题,得到一些使用Java自身调试工具的经验,与大家分享。 (1)使用top命令找出占用cpu最高的JAVA进程PID:28174 (2)如下命令找出占用cpu最高的线程
top -Hp 28174 -d 1 -n 1
32694 root 20 0 3249m 2.0g 11m S 2 6.4 3:31.12 java
- 【持久化框架MyBatis3四】MyBatis3一对一关联查询
bit1129
Mybatis3
当两个实体具有1对1的对应关系时,可以使用One-To-One的进行映射关联查询
One-To-One示例数据
以学生表Student和地址信息表为例,每个学生都有都有1个唯一的地址(现实中,这种对应关系是不合适的,因为人和地址是多对一的关系),这里只是演示目的
学生表
CREATE TABLE STUDENTS
(
- C/C++图片或文件的读写
bitcarter
写图片
先看代码:
/*strTmpResult是文件或图片字符串
* filePath文件需要写入的地址或路径
*/
int writeFile(std::string &strTmpResult,std::string &filePath)
{
int i,len = strTmpResult.length();
unsigned cha
- nginx自定义指定加载配置
ronin47
进入 /usr/local/nginx/conf/include 目录,创建 nginx.node.conf 文件,在里面输入如下代码:
upstream nodejs {
server 127.0.0.1:3000;
#server 127.0.0.1:3001;
keepalive 64;
}
server {
liste
- java-71-数值的整数次方.实现函数double Power(double base, int exponent),求base的exponent次方
bylijinnan
double
public class Power {
/**
*Q71-数值的整数次方
*实现函数double Power(double base, int exponent),求base的exponent次方。不需要考虑溢出。
*/
private static boolean InvalidInput=false;
public static void main(
- Android四大组件的理解
Cb123456
android四大组件的理解
分享一下,今天在Android开发文档-开发者指南中看到的:
App components are the essential building blocks of an Android
- [宇宙与计算]涡旋场计算与拓扑分析
comsci
计算
怎么阐述我这个理论呢? 。。。。。。。。。
首先: 宇宙是一个非线性的拓扑结构与涡旋轨道时空的统一体。。。。
我们要在宇宙中寻找到一个适合人类居住的行星,时间非常重要,早一个刻度和晚一个刻度,这颗行星的
- 同一个Tomcat不同Web应用之间共享会话Session
cwqcwqmax9
session
实现两个WEB之间通过session 共享数据
查看tomcat 关于 HTTP Connector 中有个emptySessionPath 其解释如下:
If set to true, all paths for session cookies will be set to /. This can be useful for portlet specification impleme
- springmvc Spring3 MVC,ajax,乱码
dashuaifu
springjquerymvcAjax
springmvc Spring3 MVC @ResponseBody返回,jquery ajax调用中文乱码问题解决
Spring3.0 MVC @ResponseBody 的作用是把返回值直接写到HTTP response body里。具体实现AnnotationMethodHandlerAdapter类handleResponseBody方法,具体实
- 搭建WAMP环境
dcj3sjt126com
wamp
这里先解释一下WAMP是什么意思。W:windows,A:Apache,M:MYSQL,P:PHP。也就是说本文说明的是在windows系统下搭建以apache做服务器、MYSQL为数据库的PHP开发环境。
工欲善其事,必须先利其器。因为笔者的系统是WinXP,所以下文指的系统均为此系统。笔者所使用的Apache版本为apache_2.2.11-
- yii2 使用raw http request
dcj3sjt126com
http
Parses a raw HTTP request using yii\helpers\Json::decode()
To enable parsing for JSON requests you can configure yii\web\Request::$parsers using this class:
'request' =&g
- Quartz-1.8.6 理论部分
eksliang
quartz
转载请出自出处:http://eksliang.iteye.com/blog/2207691 一.概述
基于Quartz-1.8.6进行学习,因为Quartz2.0以后的API发生的非常大的变化,统一采用了build模式进行构建;
什么是quartz?
答:简单的说他是一个开源的java作业调度框架,为在 Java 应用程序中进行作业调度提供了简单却强大的机制。并且还能和Sp
- 什么是POJO?
gupeng_ie
javaPOJO框架Hibernate
POJO--Plain Old Java Objects(简单的java对象)
POJO是一个简单的、正规Java对象,它不包含业务逻辑处理或持久化逻辑等,也不是JavaBean、EntityBean等,不具有任何特殊角色和不继承或不实现任何其它Java框架的类或接口。
POJO对象有时也被称为Data对象,大量应用于表现现实中的对象。如果项目中使用了Hiber
- jQuery网站顶部定时折叠广告
ini
JavaScripthtmljqueryWebcss
效果体验:http://hovertree.com/texiao/jquery/4.htmHTML文件代码:
<!DOCTYPE html>
<html xmlns="http://www.w3.org/1999/xhtml">
<head>
<title>网页顶部定时收起广告jQuery特效 - HoverTree<
- Spring boot内嵌的tomcat启动失败
kane_xie
spring boot
根据这篇guide创建了一个简单的spring boot应用,能运行且成功的访问。但移植到现有项目(基于hbase)中的时候,却报出以下错误:
SEVERE: A child container failed during start
java.util.concurrent.ExecutionException: org.apache.catalina.Lif
- leetcode: sort list
michelle_0916
Algorithmlinked listsort
Sort a linked list in O(n log n) time using constant space complexity.
====analysis=======
mergeSort for singly-linked list
====code======= /**
* Definition for sin
- nginx的安装与配置,中途遇到问题的解决
qifeifei
nginx
我使用的是ubuntu13.04系统,在安装nginx的时候遇到如下几个问题,然后找思路解决的,nginx 的下载与安装
wget http://nginx.org/download/nginx-1.0.11.tar.gz
tar zxvf nginx-1.0.11.tar.gz
./configure
make
make install
安装的时候出现
- 用枚举来处理java自定义异常
tcrct
javaenumexception
在系统开发过程中,总少不免要自己处理一些异常信息,然后将异常信息变成友好的提示返回到客户端的这样一个过程,之前都是new一个自定义的异常,当然这个所谓的自定义异常也是继承RuntimeException的,但这样往往会造成异常信息说明不一致的情况,所以就想到了用枚举来解决的办法。
1,先创建一个接口,里面有两个方法,一个是getCode, 一个是getMessage
public
- erlang supervisor分析
wudixiaotie
erlang
当我们给supervisor指定需要创建的子进程的时候,会指定M,F,A,如果是simple_one_for_one的策略的话,启动子进程的方式是supervisor:start_child(SupName, OtherArgs),这种方式可以根据调用者的需求传不同的参数给需要启动的子进程的方法。和最初的参数合并成一个数组,A ++ OtherArgs。那么这个时候就有个问题了,既然参数不一致,那