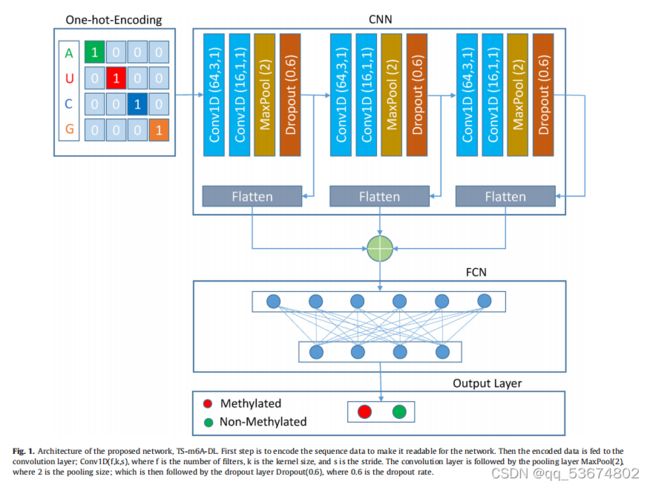
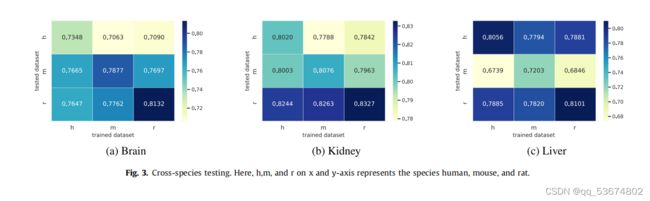
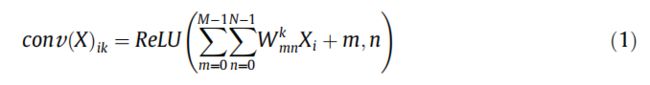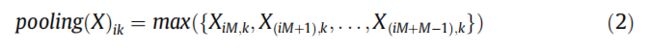- 机器学习模型监控警报系统设计:Prometheus+Evidently 实战教程
大熊计算机
机器学习prometheus人工智能
1.系统架构设计:从数据采集到智能告警(1)监控系统核心组件交互图预测请求监控指标告警规则通知渠道预测结果质量报告时序数据模型服务PrometheusExporterPrometheusServerAlertmanager邮件/Slack/WebhookEvidently服务可视化仪表盘图解:系统采用双引擎架构,Prometheus负责基础监控指标采集与告警触发,Evidently执行深度模型分析
- C++学习笔记.2
Lowjin_
C++c++学习笔记
类和对象封装语法:class关键字{访问权限属性行为}#includeusingnamespacestd;constdoublepi=3.14;//设计一个圆类classcircle{//访问权限//公共权限public://属性intr;//行为doublec(){return2*pi*r;}};intmain(){//通过圆类创建具体的圆(对象)circlec1;c1.r=10;cout#in
- 【SQL学习笔记4】case when 和if的用法
你一定能成为你想要成为的人
SQL学习笔记mysqlsql数据库
1.case用法--用法一:casewhen条件1then字段取值1when条件2then字段取值2when条件3then字段取值3else字段取值4--如果上述全部不满足,则执行end--用法二:case字段名when取值1then字段取值1when取值2then字段取值2when取值3then字段取值3else字段取值4--如果上述全部不满足,则执行end2.if用法if(条件,取值1,取值2
- 创建没有 TPM 和安全启动的 Windows 11 可启动 USB 驱动器
创建没有TPM和安全启动的Windows11可启动USB驱动器如果你使用的笔记本电脑或台式机系统不符合Windows11的系统要求,即没有安全启动和TPM2.0;那么这里有一个解决方案,可以创建一个Windows11的可启动USB驱动器,但会移除TPM和安全启动的要求。微软对安装Win11的用户设定了某些限制,这些用户使用的计算机没有TPM和安全启动功能。不过,既然凡事都有解决办法,这个问题也不例
- Python个人学习基础笔记-3.爬虫(1)
孜宸润泽
python学习笔记
一.爬虫的定义爬虫(crawler/spider)是模拟浏览器行为,按照编写规则,自动接收网页信息的工具。通常而言爬虫首先从初始URL集选择URL,向目标网页发起请求,获取网页的HTML源码,然后将获取的数据进行解析过滤,保存我们所需要的标题、内容等,最后提取新的URL加入待爬序列。爬虫常见所需要的库包括Request库、BeautifulSoup4库、Scrapy库和Selenium库等。二.R
- 和李沐老师学深度学习--2.数据操作部分代码实现(学习笔记)
大家对代码有不懂地方都可以上网去查找,最好是有一定的数据分析基础比较容易理解,李沐老师课程视频链接我放在这里了大家有不懂都可以观看课程进行学习04数据操作+数据预处理【动手学深度学习v2】_哔哩哔哩_bilibili深度学习课程电子书:大家可以使用翻译插件观看书的内容Preface—DiveintoDeepLearning1.0.3documentation深度学习github项目:https:/
- 一文教你学会使用 ts 泛型;ts 泛型常用知识点
GGhhccc
javascript开发语言前端typescript
文章目录1.泛型是什么?为什么要用泛型?2.泛型如何使用泛型类泛型约束3.泛型部分实用工具类型ExcludeExtractOmitPick4.结语最近回头复习了一下ts泛型的知识,做一些笔记的总结分享~1.泛型是什么?为什么要用泛型?引用官网的例子,此时有一个需求:我们要定义一个函数,他会返回任何传入他的值。这个情况下,我们如果已知他的数据类型(假定是number),就可以写出以下代码:funct
- ts学习笔记七:泛型
//泛型的用处在于当我们调用的时候确定类型,而不是一开始就写好类型,类型不确定,只有在执行的时候才能确定//1.单个泛型声明的时候需要用(times:number,value:T):Array{//根据对应参数的类型给T赋值//letresult=[];//for(leti=0;i{//[key:number]:T//}//interfaceICreateArray{//interface后面的类
- TS中的泛型 (学习笔记)
小码龙~
TS学习笔记前端typescript
文章目录前言一、泛型是什么?二、泛型的分类三、泛型的基本使用3.1函数中的泛型使用3.2接口中的泛型使用(运用广泛)3.2类型别名中的泛型使用(运用广泛)3.2类中的泛型使用总结前言泛型的基本使用一、泛型是什么?泛型(Generics)是指在定义函数、接⼝或类的时候,不预先指定具体的类型,⽽在使⽤的时候再指定类型的⼀种特性,简单来说泛型其实就是类型参数,在定义的时候定义形参(类型变量),使⽤的时候
- TS 函数泛型和泛型约束
邱志刚
TS前端
仅供参考,自己学习记笔记。//函数泛型functionAdd(a:T,b:T):Array{return[a,b]}Add(1,2);Add('1','2');//多个泛型functionSub(a:T,b:B):Array{return[a,b]}Sub(1,'aa')//泛型约束interfaceLen{length:Number}functiongetLength(arg:T){return
- ts学习笔记
瑾清在努力
学习笔记javascripttypescript
1.介绍ts是js的超集,他融合了其他语言的优势,将js带到了一个新的高度js,es,ts的关系:ECMAScript是JavaScript的标准,TypeScript是JavaScript的超集2.为什么使用ts?1.发现问题js---运行后报错ts---运行之前可检查出错误(静态类型检查)2.非异常故障错别字,未调用函数,基本逻辑错误constuser={name:'小明',age:26}us
- 使用LangChain构建智能应用:从入门到实战
afTFODguAKBF
langchainpython
引言在当今的人工智能时代,构建智能应用程序已经成为越来越多开发者的目标。LangChain是一个强大的工具,可以帮助我们快速开发基于大型语言模型(LLM)的应用。本篇文章将带你了解如何从零开始使用LangChain,构建一个简单的LLM应用程序,并逐步探索更复杂的功能。主要内容构建简单的LLM应用使用LangChain,我们可以快速构建一个简单的LLM应用程序。接下来,我将带你一步步实现。什么是L
- 端侧开发详解 初赛收官盛宴 | 2025高通边缘智能创新应用大赛第九场公开课来袭!
阿加犀智能
人工智能智能硬件
各位开发者、技术爱好者,2025高通边缘智能创新应用大赛即将迎来初赛阶段的最后一堂重磅公开课!诚邀大家于7月3日(星期四)晚8点,准时收看由瑞莎的嵌入式开发工程师张子烽(Morgan)带来的专题分享,共同探索端侧智能应用开发的创新技术路径。聚焦前沿平台掌握端侧智能开发流程本次课程将聚焦基于瑞莎DragonQ6A开发板的端侧人工智能应用开发。该开发板搭载高通跃龙™QCS6490平台(由阿加犀提供开发
- TS泛型笔记
红中马喽
笔记
1.泛型基础概念定义:泛型是TypeScript中允许创建可复用组件的特性,这些组件可以支持多种数据类型,而非单一特定类型。核心优势:代码复用性:同一组件可处理不同类型数据类型安全:在编译阶段捕获类型错误灵活性:保持代码的灵活性同时提供强类型支持泛型函数//基础泛型函数语法functionidentity(arg:T):T{returnarg;}//使用方式constresult=identity
- 目标跟踪领域经典论文解析
♢.*
目标跟踪人工智能计算机视觉
亲爱的小伙伴们,在求知的漫漫旅途中,若你对深度学习的奥秘、JAVA、PYTHON与SAP的奇妙世界,亦或是读研论文的撰写攻略有所探寻,那不妨给我一个小小的关注吧。我会精心筹备,在未来的日子里不定期地为大家呈上这些领域的知识宝藏与实用经验分享。每一个点赞,都如同春日里的一缕阳光,给予我满满的动力与温暖,让我们在学习成长的道路上相伴而行,共同进步✨。期待你的关注与点赞哟!目标跟踪是计算机视觉领域的一个
- hmc7044时钟芯片调试笔记
So_shine
Linux驱动总结分享linux内核驱动时钟芯片
目录前言一、依赖文档、工具二、运行linux内核驱动的平台1、代码、文件列表2、适配、编译3、调试三、无os的mcu平台1、代码、文件列表2、适配、编译3、调试前言本笔记基于运行linux操作系统的SOC芯片平台、linux内核版本linux5.10.xxx和无操作系统的mcu平台记录调试;一、依赖文档、工具文档名说明获取方式hmc7044.pdf数据手册adi官网或者国内采芯网GUI配置工具通过
- vue的侦听器及怎么侦听数组--笔记
小番茄炒鸡蛋
vue.jsjavascript前端
作用侦听属性响应数据的变化,当数据发生改变的时候会立即执行对应的函数letvm=newVue({el:"#test",data:{entry:""},watch:{entry(){console.log("侦听到了");}}})这里我同过侦听器和v-model指令一起用可以更直观的体现他的作用(这也是常用搭配)。原理:当input输入内容后,因为v-model指令的绑定,此时entry属性值会随之
- 鸿蒙开发之埋点方案:高效追踪用户行为
niu某某
移动开发鸿蒙开发HarmonyOSharmonyos鸿蒙开发移动开发组件化模块化ArkUI
往期推文全新看点(文中附带最新·鸿蒙全栈学习笔记)✒️鸿蒙应用开发与鸿蒙系统开发哪个更有前景?✒️嵌入式开发适不适合做鸿蒙南向开发?看完这篇你就了解了~✒️对于大前端开发来说,转鸿蒙开发究竟是福还是祸?✒️鸿蒙岗位需求突增!移动端、PC端、IoT到底该怎么选?✒️记录一场鸿蒙开发岗位面试经历~✒️持续更新中……概述埋点是指将信息采集程序和原本的功能代码结合起来,针对特定用户行为收集、处理和发送一些
- SpringBoot生态全景图:从SpringCloud到云原生技术栈演进
fanxbl957
Webspringbootspringcloud云原生
博主介绍:Java、Python、js全栈开发“多面手”,精通多种编程语言和技术,痴迷于人工智能领域。秉持着对技术的热爱与执着,持续探索创新,愿在此分享交流和学习,与大家共进步。DeepSeek-行业融合之万象视界(附实战案例详解100+)全栈开发环境搭建运行攻略:多语言一站式指南(环境搭建+运行+调试+发布+保姆级详解)感兴趣的可以先收藏起来,希望帮助更多的人SpringBoot生态全景图:从S
- 燕山大学软件用户界面设计考题
能运行就算成功
经验分享
2024年考题,考前完全不知道考什么,趁着现在还记得,造福下后辈。全部是简答。1.描述下实用性和它的三个维度2.写出五个功能可见性的例子3.关键性模型Keystroke-LevelModel(KLM)字母的意思4.undo四个设计原则(笔记和翻译根本没有,看到时已经懵了)5.GUI三种设计方式6.瀑布模型为什么不适合ui设计后面是大题,跟写实验报告差不多,这次是个预定家政服务的题,写信息点描述中心
- 燕大《Python机器学习》实验报告:探索机器学习的奥秘
温冰礼
燕大《Python机器学习》实验报告:探索机器学习的奥秘【下载地址】燕大Python机器学习实验报告下载这份实验报告是燕山大学软件工程专业的学生在进行机器学习实验时所编写的,内容详实,结构清晰,可以直接下载使用。报告中的实验数据和代码均经过验证,确保下载后可以直接应用于实际项目或作为学习参考项目地址:https://gitcode.com/Open-source-documentation-tut
- Microsoft VBA Excel VBA学习笔记——双重筛选+复制数值1.0
偷心伊普西隆
VBA学习和实践microsoftexcel
问题场景CountryProductCLASS1CLASS2CLASS3CLASS4CLASS5CLASS6…USApple0.3641416030.8918210610.0591451990.7320110290.0509636560.222464259…USBanana0.2300833330.4027262180.1548836670.2988904860.7802326210.028592
- SQL Server的个人学习笔记
萌尛喵
sql学习数据库
1.基础SQLServer是由Microsoft开发和销售的关系数据库管理系统或RDBMS。SQLServer建立于SOL之上,是一种用于关系数据交互的标准编程语言。2.组件SQLServer主要由数据库引擎和SQLOS两个组件组成。①数据库引擎SQLServer的核心组件是数据库引擎。数据库引擎由处理查询的关系引擎和管理数据库文件、页面、索引等的存储组成。数据库引擎也创建并执行数据库对象,如存储
- SQLserver数据库学习笔记
溪衡
学习
小记1:1.newid()我觉得是一个生成唯一键的好方法,不用自增控制主键,可以用这个试试,注意不做处理的话,需要36位。例如:在数据库中直接使用语句selectnewid()2.nolock按我的理解是“不上锁的”,所谓的脏读,大多用的都是这个东西,据说可以提高查询速度。3.go批处理语句,将前面的代码作为一批处理。4.内连接与简单多表在数据量少的时候查询速度差距并不明显。5.删除和更新数据时,
- (转)优秀的 python 机器学习库
patrick75
python机器学习python机器学习
优秀的python机器学习库IntroductionThereisnodoubtthatneuralnetworks,andmachinelearningingeneral,hasbeenoneofthehottesttopicsintechthepastfewyearsorso.It’seasytoseewhywithallofthereallyinterestinguse-casestheys
- DAY 10 机器学习建模与评估
心落薄荷糖
Python训练营机器学习人工智能
知识点:1.数据集的划分2.机器学习模型建模的三行代码3.机器学习模型分类问题的评估今日代码比较多,但是难度不大,仔细看看示例代码,好好理解下这几个评估指标。作业:尝试对心脏病数据集采用机器学习模型建模和评估#一、导入库importpandasaspdimportpandasaspd#用于数据处理和分析,可处理表格数据。importnumpyasnp#用于数值计算,提供了高效的数组操作。impor
- SQL学习笔记1
1.数据库1、什么是数据库数据库(DB)即用于存放数据的服务器,如MySQL等软件是数据库管理系统(DBMS),用于管理存放在数据库中的数据,SQL是用于操作DBMS的标准语言。2、数据库的类型数据库分为关系型数据库和非关系型数据库;关系型数据库是指用建立在关系模型上互相关联的二维表组成的数据库,MySQL是用于管理关系型数据库的数据库管理系统2.MySQL启动与连接1、MySQL启动安装好MyS
- 【图像处理入门】12. 综合项目与进阶:超分辨率、医学分割与工业检测
小米玄戒Andrew
图像处理:从入门到专家图像处理人工智能深度学习算法python计算机视觉CV
摘要本周将聚焦三个高价值的综合项目,打通传统算法与深度学习的技术壁垒。通过图像超分辨率重建对比传统方法与深度学习方案,掌握医学图像分割的U-Net实现,设计工业缺陷检测的完整流水线。每个项目均包含原理解析、代码实现与性能优化,帮助读者从“技术应用”迈向“系统设计”。一、项目1:图像超分辨率重建(从模糊到清晰的跨越)1.技术背景与核心指标超分辨率(SR)是通过算法将低分辨率(LR)图像恢复为高分辨率
- Python机器学习元学习库higher
音程
机器学习人工智能python机器学习
higher是一个用于元学习(Meta-Learning)和高阶导数(Higher-ordergradients)的Python库,专为PyTorch设计。它扩展了PyTorch的自动微分机制,使得在训练过程中可以动态地计算参数的梯度更新,并把这些更新过程纳入到更高阶的梯度计算中。一、主要用途higher主要用于以下场景:元学习(Meta-Learning)比如MAML(Model-Agnosti
- Learning PostgresSQL读书笔记: 第8章 Triggers and Rules
dingdingfish
PostgresSQLpostgresqldatabasearchitecturetutorial
本章将讨论以下内容:•探索PostgreSQL中的规则•管理PostgreSQL中的触发器•事件触发器探索PostgreSQL中的规则文档中的这段话阐述了rule和trigger的区别:PostgreSQL规则系统允许定义在数据库表中插入、更新或删除时执行的替代操作。粗略地说,当对给定表执行给定命令时,规则会执行其他命令。或者,INSTEAD规则可以用另一个命令替换给定命令,或者导致命令根本不执行
- java线程的无限循环和退出
3213213333332132
java
最近想写一个游戏,然后碰到有关线程的问题,网上查了好多资料都没满足。
突然想起了前段时间看的有关线程的视频,于是信手拈来写了一个线程的代码片段。
希望帮助刚学java线程的童鞋
package thread;
import java.text.SimpleDateFormat;
import java.util.Calendar;
import java.util.Date
- tomcat 容器
BlueSkator
tomcatWebservlet
Tomcat的组成部分 1、server
A Server element represents the entire Catalina servlet container. (Singleton) 2、service
service包括多个connector以及一个engine,其职责为处理由connector获得的客户请求。
3、connector
一个connector
- php递归,静态变量,匿名函数使用
dcj3sjt126com
PHP递归函数匿名函数静态变量引用传参
<!doctype html>
<html lang="en">
<head>
<meta charset="utf-8">
<title>Current To-Do List</title>
</head>
<body>
- 属性颜色字体变化
周华华
JavaScript
function changSize(className){
var diva=byId("fot")
diva.className=className;
}
</script>
<style type="text/css">
.max{
background: #900;
color:#039;
- 将properties内容放置到map中
g21121
properties
代码比较简单:
private static Map<Object, Object> map;
private static Properties p;
static {
//读取properties文件
InputStream is = XXX.class.getClassLoader().getResourceAsStream("xxx.properti
- [简单]拼接字符串
53873039oycg
字符串
工作中遇到需要从Map里面取值拼接字符串的情况,自己写了个,不是很好,欢迎提出更优雅的写法,代码如下:
import java.util.HashMap;
import java.uti
- Struts2学习
云端月影
最近开始关注struts2的新特性,从这个版本开始,Struts开始使用convention-plugin代替codebehind-plugin来实现struts的零配置。
配置文件精简了,的确是简便了开发过程,但是,我们熟悉的配置突然disappear了,真是一下很不适应。跟着潮流走吧,看看该怎样来搞定convention-plugin。
使用Convention插件,你需要将其JAR文件放
- Java新手入门的30个基本概念二
aijuans
java新手java 入门
基本概念: 1.OOP中唯一关系的是对象的接口是什么,就像计算机的销售商她不管电源内部结构是怎样的,他只关系能否给你提供电就行了,也就是只要知道can or not而不是how and why.所有的程序是由一定的属性和行为对象组成的,不同的对象的访问通过函数调用来完成,对象间所有的交流都是通过方法调用,通过对封装对象数据,很大限度上提高复用率。 2.OOP中最重要的思想是类,类是模板是蓝图,
- jedis 简单使用
antlove
javarediscachecommandjedis
jedis.RedisOperationCollection.java
package jedis;
import org.apache.log4j.Logger;
import redis.clients.jedis.Jedis;
import java.util.List;
import java.util.Map;
import java.util.Set;
pub
- PL/SQL的函数和包体的基础
百合不是茶
PL/SQL编程函数包体显示包的具体数据包
由于明天举要上课,所以刚刚将代码敲了一遍PL/SQL的函数和包体的实现(单例模式过几天好好的总结下再发出来);以便明天能更好的学习PL/SQL的循环,今天太累了,所以早点睡觉,明天继续PL/SQL总有一天我会将你永远的记载在心里,,,
函数;
函数:PL/SQL中的函数相当于java中的方法;函数有返回值
定义函数的
--输入姓名找到该姓名的年薪
create or re
- Mockito(二)--实例篇
bijian1013
持续集成mockito单元测试
学习了基本知识后,就可以实战了,Mockito的实际使用还是比较麻烦的。因为在实际使用中,最常遇到的就是需要模拟第三方类库的行为。
比如现在有一个类FTPFileTransfer,实现了向FTP传输文件的功能。这个类中使用了a
- 精通Oracle10编程SQL(7)编写控制结构
bijian1013
oracle数据库plsql
/*
*编写控制结构
*/
--条件分支语句
--简单条件判断
DECLARE
v_sal NUMBER(6,2);
BEGIN
select sal into v_sal from emp
where lower(ename)=lower('&name');
if v_sal<2000 then
update emp set
- 【Log4j二】Log4j属性文件配置详解
bit1129
log4j
如下是一个log4j.properties的配置
log4j.rootCategory=INFO, stdout , R
log4j.appender.stdout=org.apache.log4j.ConsoleAppender
log4j.appender.stdout.layout=org.apache.log4j.PatternLayout
log4j.appe
- java集合排序笔记
白糖_
java
public class CollectionDemo implements Serializable,Comparable<CollectionDemo>{
private static final long serialVersionUID = -2958090810811192128L;
private int id;
private String nam
- java导致linux负载过高的定位方法
ronin47
定位java进程ID
可以使用top或ps -ef |grep java
![图片描述][1]
根据进程ID找到最消耗资源的java pid
比如第一步找到的进程ID为5431
执行
top -p 5431 -H
![图片描述][2]
打印java栈信息
$ jstack -l 5431 > 5431.log
在栈信息中定位具体问题
将消耗资源的Java PID转
- 给定能随机生成整数1到5的函数,写出能随机生成整数1到7的函数
bylijinnan
函数
import java.util.ArrayList;
import java.util.List;
import java.util.Random;
public class RandNFromRand5 {
/**
题目:给定能随机生成整数1到5的函数,写出能随机生成整数1到7的函数。
解法1:
f(k) = (x0-1)*5^0+(x1-
- PL/SQL Developer保存布局
Kai_Ge
近日由于项目需要,数据库从DB2迁移到ORCAL,因此数据库连接客户端选择了PL/SQL Developer。由于软件运用不熟悉,造成了很多麻烦,最主要的就是进入后,左边列表有很多选项,自己删除了一些选项卡,布局很满意了,下次进入后又恢复了以前的布局,很是苦恼。在众多PL/SQL Developer使用技巧中找到如下这段:
&n
- [未来战士计划]超能查派[剧透,慎入]
comsci
计划
非常好看,超能查派,这部电影......为我们这些热爱人工智能的工程技术人员提供一些参考意见和思想........
虽然电影里面的人物形象不是非常的可爱....但是非常的贴近现实生活....
&nbs
- Google Map API V2
dai_lm
google map
以后如果要开发包含google map的程序就更麻烦咯
http://www.cnblogs.com/mengdd/archive/2013/01/01/2841390.html
找到篇不错的文章,大家可以参考一下
http://blog.sina.com.cn/s/blog_c2839d410101jahv.html
1. 创建Android工程
由于v2的key需要G
- java数据计算层的几种解决方法2
datamachine
javasql集算器
2、SQL
SQL/SP/JDBC在这里属于一类,这是老牌的数据计算层,性能和灵活性是它的优势。但随着新情况的不断出现,单纯用SQL已经难以满足需求,比如: JAVA开发规模的扩大,数据量的剧增,复杂计算问题的涌现。虽然SQL得高分的指标不多,但都是权重最高的。
成熟度:5星。最成熟的。
- Linux下Telnet的安装与运行
dcj3sjt126com
linuxtelnet
Linux下Telnet的安装与运行 linux默认是使用SSH服务的 而不安装telnet服务 如果要使用telnet 就必须先安装相应的软件包 即使安装了软件包 默认的设置telnet 服务也是不运行的 需要手工进行设置 如果是redhat9,则在第三张光盘中找到 telnet-server-0.17-25.i386.rpm
- PHP中钩子函数的实现与认识
dcj3sjt126com
PHP
假如有这么一段程序:
function fun(){
fun1();
fun2();
}
首先程序执行完fun1()之后执行fun2()然后fun()结束。
但是,假如我们想对函数做一些变化。比如说,fun是一个解析函数,我们希望后期可以提供丰富的解析函数,而究竟用哪个函数解析,我们希望在配置文件中配置。这个时候就可以发挥钩子的力量了。
我们可以在fu
- EOS中的WorkSpace密码修改
蕃薯耀
修改WorkSpace密码
EOS中BPS的WorkSpace密码修改
>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>
蕃薯耀 201
- SpringMVC4零配置--SpringSecurity相关配置【SpringSecurityConfig】
hanqunfeng
SpringSecurity
SpringSecurity的配置相对来说有些复杂,如果是完整的bean配置,则需要配置大量的bean,所以xml配置时使用了命名空间来简化配置,同样,spring为我们提供了一个抽象类WebSecurityConfigurerAdapter和一个注解@EnableWebMvcSecurity,达到同样减少bean配置的目的,如下:
applicationContex
- ie 9 kendo ui中ajax跨域的问题
jackyrong
AJAX跨域
这两天遇到个问题,kendo ui的datagrid,根据json去读取数据,然后前端通过kendo ui的datagrid去渲染,但很奇怪的是,在ie 10,ie 11,chrome,firefox等浏览器中,同样的程序,
浏览起来是没问题的,但把应用放到公网上的一台服务器,
却发现如下情况:
1) ie 9下,不能出现任何数据,但用IE 9浏览器浏览本机的应用,却没任何问题
- 不要让别人笑你不能成为程序员
lampcy
编程程序员
在经历六个月的编程集训之后,我刚刚完成了我的第一次一对一的编码评估。但是事情并没有如我所想的那般顺利。
说实话,我感觉我的脑细胞像被轰炸过一样。
手慢慢地离开键盘,心里很压抑。不禁默默祈祷:一切都会进展顺利的,对吧?至少有些地方我的回答应该是没有遗漏的,是不是?
难道我选择编程真的是一个巨大的错误吗——我真的永远也成不了程序员吗?
我需要一点点安慰。在自我怀疑,不安全感和脆弱等等像龙卷风一
- 马皇后的贤德
nannan408
马皇后不怕朱元璋的坏脾气,并敢理直气壮地吹耳边风。众所周知,朱元璋不喜欢女人干政,他认为“后妃虽母仪天下,然不可使干政事”,因为“宠之太过,则骄恣犯分,上下失序”,因此还特地命人纂述《女诫》,以示警诫。但马皇后是个例外。
有一次,马皇后问朱元璋道:“如今天下老百姓安居乐业了吗?”朱元璋不高兴地回答:“这不是你应该问的。”马皇后振振有词地回敬道:“陛下是天下之父,
- 选择某个属性值最大的那条记录(不仅仅包含指定属性,而是想要什么属性都可以)
Rainbow702
sqlgroup by最大值max最大的那条记录
好久好久不写SQL了,技能退化严重啊!!!
直入主题:
比如我有一张表,file_info,
它有两个属性(但实际不只,我这里只是作说明用):
file_code, file_version
同一个code可能对应多个version
现在,我想针对每一个code,取得它相关的记录中,version 值 最大的那条记录,
SQL如下:
select
*
- VBScript脚本语言
tntxia
VBScript
VBScript 是基于VB的脚本语言。主要用于Asp和Excel的编程。
VB家族语言简介
Visual Basic 6.0
源于BASIC语言。
由微软公司开发的包含协助开发环境的事
- java中枚举类型的使用
xiao1zhao2
javaenum枚举1.5新特性
枚举类型是j2se在1.5引入的新的类型,通过关键字enum来定义,常用来存储一些常量.
1.定义一个简单的枚举类型
public enum Sex {
MAN,
WOMAN
}
枚举类型本质是类,编译此段代码会生成.class文件.通过Sex.MAN来访问Sex中的成员,其返回值是Sex类型.
2.常用方法
静态的values()方