7. 吴恩达机器学习-PCA
1. PCA理论原理
1. 降维与PCA
降维:将数据由原来的 n 个特征缩减为 k 个特征(可能从 n 个中直接选取 k 个,也能根据这 n 个重新组合成 k 个)。可起到数据压缩的作用(因而也就存在数据丢失)。
PCA:即主成分分析法,属于降维的一种方法。其主要思想是:根据原始的 n 个特征(也就是 n 维),重新组合出 k 个特征,且这 k 个特征能最大量度地涵盖原始的数据信息(虽然会导致信息丢失,但所丢失信息可忽略不计)。有一个结论:**当某一维的方差越大时,其所包含的信息量也越大;反之则反。**所以,PCA 的主要工作就是:重构出 k 个特征,使其所包含的信息量最大。
以下是两个例子:
第一幅图:将平面上(二维)的点映射到一条直线或向量上(一维),其丢失的信息量就是:每个点到直线上的距离。因为降维之后,就认为所有点都在直线上了。

第二幅图将空间上投影到一个平面上。注意:这两个例子都选取了与原始数据尽可能 “靠近” 的直线或平面,使得其保存下来的信息量最大。

2. 算法过程
2. 求出特征的协方差矩阵
∑ = 1 m X T X \sum=\frac{1}{m}X^TX ∑=m1XTX
3. 求出协方差矩阵的特征值及特征向量,这里可直接调用函数库

其中, S S S 为对角矩阵,其对角线上的数就是协方差矩阵的特征值,而 U U U 就是协方差矩阵的特征向量。而 U U U 的前 k 列就是我们要求的新特征(用于代替原来的 n 个特征,起到数据压缩的作用)。所以,假设原始的数据特征为 x(n维),经过变换后变为 z(k 维),则有如下公式: [ U , S , V ] = s v d ( S i g m a ) ; U r e d u c e = U ( : , 1 : k ) ; z = U r e d u c e ′ × x ; [U, S, V]=svd(Sigma); \\ Ureduce = U(:,1:k); \\ z = Ureduce'\times x; [U,S,V]=svd(Sigma);Ureduce=U(:,1:k);z=Ureduce′×x;
3. 算法原理
从上面 PCA 的实现步骤可以发现,它的关键步骤便是求出样本协方差矩阵 C 的特征向量矩阵。可是,为什么要这么做呢?
上文提到过,PCA 变换其实就是一种降维技术。
什么是降维?
降维就是指通过矩阵乘法运算后,把原来的矩阵维度减少。维数减少了,虽然可以大大减少算法的计算量,但是若对基矩阵 P 选择不当的话就很有可能会导致信息量的缺失。因此我们要选择哪 K 个基(这里还不知道是特征向量)才能保证降维后能最大程度保留原有的信息,是进行设计的主方向。
什么是信息呢?
根据信息论的定义,我们可以知道信息来源于未知。也就是说如果不同样本的同一维度的值差异特别大,那该维度带给我们的信息量就是极大的。转换成数学语言,也就是说某维度的方差越大,它的信息量越大。
举个例子:假如你有 3 个人的人脸特征数据,他们均有 3 个维度:眼睛、鼻子、嘴巴。如果他们鼻子这一维度的数据都是一样的,如下图中,三个人都是大鼻子(方差=0)。那么我们从鼻子这一维度获得的信息量就是为零,因为无法从该维度得知该人脸图像到底属于谁的。

那如果他们鼻子这一维度的数据各不相同,如下图中,三个人分别是大、中、小鼻子(方差很大)。那么我们从鼻子这一维度获得的信息量就很大了,甚至直接用这一个维度就可以识别出是谁的人脸图像。

综上,我们就可以很容易联想到 第一个优化目标:降维后各维度的方差尽可能大。
既然有了第一个,当然就会有第二个啦,我们的第二个优化目标便是保证不同维度之间的相关性为0(其实就是让基向量互相正交)。
因为如果某2个维度间存在相关性,就说明从一个维度的值可以推测出另一个维度的值。说明该维度中有一个是多余的,对我们识别特征帮助不大,那自然可以把其中一个维度舍去。
比如说:如果上面例子中嘴巴与鼻子这两个维度之间存在线性关系(相关性为1,完全相关):对于格式:(y)嘴巴、(x)鼻子,有y=x,x∈[大,中,小]。那么嘴巴这一维度在这里是不是就是冗余的?有它没它都一个样,完全可以通过鼻子的大小推测出嘴巴的大小,进而判断出人脸的身份。
综上所述:PCA 算法的优化目标就是:①降维后同一维度的方差最大 ②不同维度之间的相关性为0
根据线性代数,我们可以知道同一元素的协方差就表示该元素的方差,不同元素之间的协方差就表示它们的相关性。因此这两个优化目标可以用协方差矩阵来表示,如下图: C = [ C o v ( x 1 , x 1 ) C o v ( x 1 , x 2 ) … C o v ( x 1 , x k ) C o v ( x 2 , x 1 ) C o v ( x 2 , x 2 ) … C o v ( x 2 , x k ) ⋮ ⋮ ⋱ ⋮ C o v ( x k , x 1 ) C o v ( x k , x 2 ) … C o v ( x k , x k ) ] ⇒ C Y = [ δ 11 0 … 0 0 δ 22 … 0 ⋮ ⋮ ⋱ ⋮ 0 0 … δ k k ] C= \left[ \begin{matrix} Cov(x_1,x_1) & Cov(x_1,x_2) & \dots & Cov(x_1,x_k) \\ Cov(x_2,x_1) & Cov(x_2,x_2) & \dots & Cov(x_2,x_k) \\ \vdots & \vdots & \ddots & \vdots \\ Cov(x_k,x_1) & Cov(x_k,x_2) & \dots & Cov(x_k,x_k) \end{matrix} \right]\Rightarrow C_Y= \left[ \begin{matrix} \delta_{11} & 0 & \dots & 0 \\ 0 & \delta_{22} & \dots & 0 \\ \vdots & \vdots & \ddots & \vdots \\ 0 & 0 & \dots & \delta_{kk} \end{matrix} \right] C=⎣ ⎡Cov(x1,x1)Cov(x2,x1)⋮Cov(xk,x1)Cov(x1,x2)Cov(x2,x2)⋮Cov(xk,x2)……⋱…Cov(x1,xk)Cov(x2,xk)⋮Cov(xk,xk)⎦ ⎤⇒CY=⎣ ⎡δ110⋮00δ22⋮0……⋱…00⋮δkk⎦ ⎤ C o v ( x m , x k ) = 1 n − 1 ∑ i = 1 n ( x m i − x ‾ m ) ( x k i − x ‾ k ) Cov(x_m, x_k)=\frac{1}{n-1}\sum_{i=1}^{n}{(x_{mi}-\overline{x}_m)(x_{ki}-\overline{x}_k)} Cov(xm,xk)=n−11i=1∑n(xmi−xm)(xki−xk) 可以知道优化目标一可转换为令 C y C_y Cy 矩阵的对角线元素之和最大,即 m a x max max t r ( C y ) tr(C_y) tr(Cy)。知道了目标,接下来就好办了,我们要做的就是想尽一切办法去达到我们的优化目标。下面便是推导过程:
设 X X X 为 m × n m\times n m×n 的样本中心化矩阵, P P P 为 n × k n\times k n×k 的基矩阵(每一列为一个基向量),降维后的样本矩阵 Y = X P Y=XP Y=XP,则 Y Y Y 矩阵的协方差 C Y ′ = 1 m ( X P ) T ( X P ) = P T ( 1 m X T X ) P ⇒ C Y ′ = P T C P C'_Y=\frac{1}{m}(XP)^T(XP)=P^T(\frac{1}{m}X^TX)P\Rightarrow C'_Y=P^TCP CY′=m1(XP)T(XP)=PT(m1XTX)P⇒CY′=PTCP 由于 P P P 是正交的(正交能减少降维后不同维度间的相关性),所以 P T P = E P^TP=E PTP=E,因此我们的优化目标可以表示为: { m a x ( t r ( P T C P ) ) P T P = E \begin{cases} max(tr(P^TCP)) \\ P^TP=E \end{cases} {max(tr(PTCP))PTP=E 根据拉格朗日乘子法,可得: f ( P ) = t r ( P T C P ) + λ ( P T P − E ) f(P)=tr(P^TCP)+\lambda(P^TP-E) f(P)=tr(PTCP)+λ(PTP−E) 下面对 f ( P ) f(P) f(P) 求导,取其零点,就可以知道 P P P 是什么才符合我们的要求了。
矩阵的迹有如下重要性质: { ∂ t r ( A B ) ∂ A = B T t r ( U V ) = t r ( V U ) \begin{cases} \frac{\partial tr(AB)}{\partial A}=B^T \\ tr(UV)=tr(VU) \end{cases} {∂A∂tr(AB)=BTtr(UV)=tr(VU) ∂ f ∂ P = ∂ t r ( P T C P ) ∂ P + λ ∂ ( P T P ) ∂ P = ∂ t r ( P P T C ) ∂ P + λ P = ( P T C ) T + λ P = C T P + λ P = C P + λ P \frac{\partial f}{\partial P}=\frac{\partial tr(P^TCP)}{\partial P}+\lambda\frac{\partial (P^TP)}{\partial P}=\frac{\partial tr(PP^TC)}{\partial P}+\lambda P=(P^TC)^T+\lambda P=C^TP+\lambda P=CP+\lambda P ∂P∂f=∂P∂tr(PTCP)+λ∂P∂(PTP)=∂P∂tr(PPTC)+λP=(PTC)T+λP=CTP+λP=CP+λP 当 ∂ f ∂ P = 0 \frac{\partial f}{\partial P} = 0 ∂P∂f=0 时: C P + λ P = 0 ⇒ C P = ( − λ ) P CP+\lambda P=0\Rightarrow CP=(-\lambda)P CP+λP=0⇒CP=(−λ)P 可以看到,最终求得的结果满足特征向量的关系式,因此由 C C C 矩阵的特征向量所构成的基矩阵,就是我们要求的变换矩阵。由该矩阵降维得到的新样本矩阵可以最大程度保留原样本的信息。
至此,问题都已经解决了:信息量保存能力最大的基向量一定是样本矩阵 X X X 的协方差矩阵的特征向量,并且这个特征向量保存的信息量就是它对应的特征值的绝对值。这个推导过程就解释了为什么 PCA 算法要利用样本协方差的特征向量矩阵来降维。我觉得这正是理解 PCA 算法的关键点,只要理解了这一点,对 PCA 算法也就基本掌握了。
4. PCA之恢复
- 对人脸图像进行降维压缩的效果如下:

- 压缩后再进行恢复时,丢失的那部分数据找不回来了。恢复方式如下:

由于降维后的样本为: Z = U r e d u c e T × X Z = U_{reduce}^T\times X Z=UreduceT×X,也就是把左图中的二维数据转换到一维的数轴上去。因此要将一维数据恢复为原来的二维数据,其公式为: X a p p r o x = U r e d u c e × Z X_{approx}=U_{reduce}\times Z Xapprox=Ureduce×Z,也即图右的二维空间,但此时所有的点都落在了直线上。所以丢失的数据即为原始点与直线的距离。
5. 如何选取维数K
如果可能,k当然越小越好,k越小表明压缩的程度越高,但同时又要保证足够多的数据量。因此,选出最小的k,满足:

以下为其求解求解过程,并且我们可以直接调用函数库:


6. PCA的作用与适用场合
2. 代码实现
1. 2D PCA
import matplotlib.pyplot as plt
import seaborn as sns
sns.set(context="notebook", style="white")
import numpy as np
import pandas as pd
import scipy.io as sio
加载数据集
# 获取训练数据
def get_X(df):
ones = pd.DataFrame({'ones':np.ones(len(df))})
data = pd.concat([ones, df], axis=1)
return data.iloc[:, :-1].values # 将数据转换为数组形式
# 获取标签
def get_y(df):
return np.array(df.iloc[:, -1])
# 对特征进行归一化处理
def normalize_feature(df):
return df.apply(lambda column: (column - column.mean()) / column.std())
mat = sio.loadmat('../data/ex7data1.mat')
X = mat.get('X') # 获取训练数据,X.shape=(50,2)
# 对数据进行可视化展示
sns.lmplot('X1','X2',data=pd.DataFrame(X, columns=['X1', 'X2']), fit_reg=False)
plt.show()
# 连续画 n 张图片
def plot_n_image(X, n):
pic_size = int(np.sqrt(X.shape[1]))
grid_size = int(np.sqrt(n))
first_n_images = X[:n, :]
fig, ax_array = plt.subplots(nrows=grid_size, ncols=grid_size, sharey=True, sharex=True, figsize=(8,8))
for r in range(grid_size):
for c in range(grid_size):
ax_array[r,c].imshow(first_n_images[grid_size * r + c].reshape(pic_size, pic_size))
plt.xticks(np.array([])) # np.array()创建一个数组,plt.xticks()记录和设置x坐标
plt.yticks(np.array([]))
# 计算 X 的协方差矩阵
def covariance_matrix(X):
m = X.shape[0]
return (X.T @ X) / m
# 归一化处理
def normalize(X):
X_copy = X.copy()
m, n = X_copy.shape
for col in range(n):
X_copy[:, col] = (X_copy[:, col] - X_copy[:, col].mean()) / X_copy[:,col].std()
return X_copy
# pca 算法实现
def pca(X):
X_norm = normalize(X)
Sigma = covariance_matrix(X_norm)
# U:左奇异矩阵,列由 A@A.T 的特征向量组成,且特征向量为单位向量
# S:奇异值矩阵,对角线元素来源于 A@A.T 的特征值的平方根,按降序排列,值越大则越重要
# V:右奇异矩阵,列由 A.T@A 的特征向量组成,且特征向量为单位向量
U, S, V = np.linalg.svd(Sigma)
return U, S, V
# 将数据降到 k 维
def project_data(X, U, k):
m, n = X.shape
if k > n:
raise ValueError('k should be lower dimension of n')
return X @ U[:, :k]
# 恢复数据
def recover_data(Z, U):
m, n = Z.shape
if n >= U.shape[0]:
raise ValueError('Z dimension is >= U, you should recover from lower dimension to higher')
return Z @ U[:, :n].T
对数据进行归一化处理
X_norm = normalize(X)
sns.lmplot('X1', 'X2', data=pd.DataFrame(X_norm, columns=['X1', 'X2']), fit_reg=False)
plt.show()
Sigma = covariance_matrix(X_norm)
将数据映射到1维,并进行可视化展示
U, S, V = pca(X_norm)
# show top 10 projected data
Z = project_data(X_norm, U, 1)
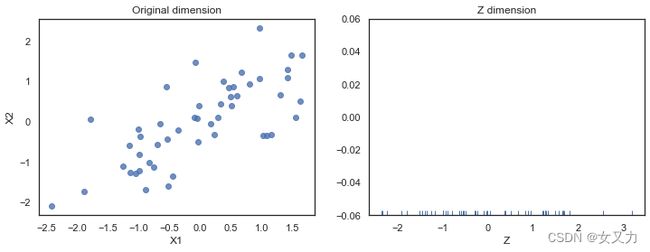
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(12, 4))
# 比较两个变量的关系是否符合线性回归,fit_reg=False表示不拟合直线
sns.regplot('X1', 'X2', data=pd.DataFrame(X_norm, columns=['X1', 'X2']), fit_reg=False, ax=ax1)
ax1.set_title('Original dimension')
# 绘制出一维数组中数据点实际的分布位置
sns.rugplot(Z.flatten(), ax=ax2)
ax2.set_xlabel('Z')
ax2.set_title('Z dimension')
plt.show()
X_recover = recover_data(Z, U)
fig, (ax1, ax2, ax3) = plt.subplots(ncols=3, figsize=(12, 4))
sns.rugplot(Z.flatten(), ax=ax1)
ax1.set_title('Z dimension')
ax1.set_xlabel('Z')
# 恢复数据必然会丢失某些信息
sns.regplot('X1', 'X2', data=pd.DataFrame(X_recover, columns=['X1', 'X2']), fit_reg=False, ax=ax2)
ax2.set_title("2D projection from Z")
# 画出实际数据,进行对比
sns.regplot('X1', 'X2', data=pd.DataFrame(X_norm, columns=['X1', 'X2']), fit_reg=False, ax=ax3)
ax3.set_title('Original dimension')
plt.show()
2. PCA 用于面部数据
import matplotlib.pyplot as plt
import seaborn as sns
sns.set(context="notebook", style="white")
import numpy as np
import pandas as pd
import scipy.io as sio
mat = sio.loadmat('../data/ex7faces.mat')
mat.keys() # ['__header__', '__version__', '__globals__', 'X']
# 由于人脸数据错误,这样做是为了将人脸数据反转90°
X = np.array([x.reshape((32, 32)).T.reshape(1024) for x in mat.get('X')])
X.shape # (5000, 1024)
plot_n_image(X, n=64)
plt.show()
U, _, _ = pca(X)
U.shape # (1024, 1024)
# 在主成分中没有看到人脸
plot_n_image(U, n=36)
plt.show()
# 降维后也没有人脸
Z = project_data(X, U, k=100)
plot_n_image(Z, n=64)
plt.show()
# 虽然失去了某些细节,但它们是十分相似的
X_recover = recover_data(Z, U)
plot_n_image(X_recover, n=64)
plt.show()

使用 sklearn 库的 PCA算法与我们设计的算法的效果进行对比
sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False)
# n_components:PCA算法中索要保留的主成分个数 n
# copy:是否在运行算法时,将原始数据复制一份,若为True,则运行PCA算法后院训练数据的值不会由任何改变
# whiten:白化,使得每个特征具有相同的方差
PCA 方法:
fit(X, y=None):用数据 X 来训练 PCA 模型。返回调用 fit 方法的对象本身
fit_transform(X):用X来训练 PCA 模型,同时返回降维后的数据
inverse_transform():将降维后的数据转换成原始数据
transform(X):将数据X转换成降维后的数据。当模型训练好后,对于新输入的数据,都可以用transform方法来降维。
from sklearn.decomposition import PCA
sk_pca = PCA(n_components=100)
Z = sk_pca.fit_transform(X)
Z.shape # (5000, 100)
plot_n_image(Z, 64)
plt.show()
X_recover = sk_pca.inverse_transform(Z)
X_recover.shape # (5000, 1024)
plot_n_image(X_recover, n=64)
plt.show()