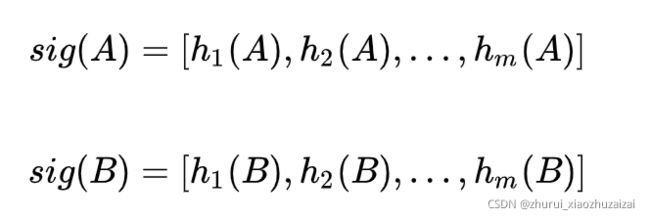LSH局部敏感哈希
1.简介
局部敏感哈希(Locality Sensitive Hashing,LSH)主要是为了处理高维度数据的查询和匹配等操作。
相似度的计算有多种方式:欧氏距离、余弦相似度或者Jaccard相似度,不管以何种计算方式,在数据维度较小时,都可以用naive的方式直接遍历每一个pair去计算。
但当数据维度增大到一定程度时,计算复杂度就开始飙升了
【文本相似性计算】minHash和LSH算法
大规模数据的相似度计算:LSH算法
LSH算法分两种:SimHash和MinHash。
simhash的原理是减少搜索空间,用汉明距离替代余弦距离
minHash的原理是降维。通过hash映射函数,将特征元素的个数降下来。
广泛应用的LSH算法:
1, 基于Stable Distribution投影方法 2, 基于随机超平面投影的方法; 3, SimHash; 4, Kernel LSH
2.Jaccard相似度
判断两个集合是否相等,一般使用称之为Jaccard相似度的算法(后面用Jac(S1,S2)来表示集合S1和S2的Jaccard相似度)。举个列子,集合X = {a,b,c},Y = {b,c,d}。那么Jac(X,Y) = 2 / 4 = 0.50。也就是说,结合X和Y有50%的元素相同。下面是形式的表述Jaccard相似度公式:
Jac(X,Y) = |X∩Y| / |X∪Y|
也就是两个结合交集的个数比上两个集合并集的个数。范围在[0,1]之间。
3.LSH之相似网页查找——Simhash
为了计算哈希后各向量之间的距离,这里使用汉明距离。
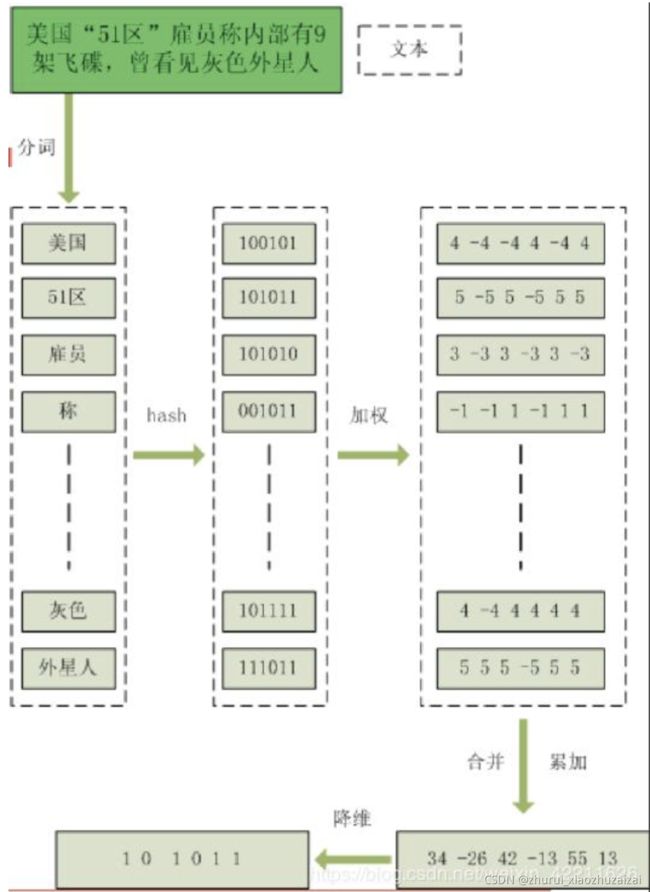
上面的图是这个算法的大致流程图,下面是具体过程:
假设当前文本文件为 “美国“51区”雇员称内部有9架飞机,曾看见灰色外星人”
(1)提取特征
使用jieba库函数,将上文本提取出特征分别为“美国”、“51区”、“雇员”、“称”、……,“灰色”、“外星人”等。
(2)Hash
然后使用各种好使(即指尽量减少碰撞又能表达原特征)的手段将提取出的特征进行hash,hash后的特征如上图的第二列所示。
(3)加权
为每一个特征赋予一个权重,这个权重可使用tf-idf统计词频(当前文件中该特征出现的次数/所有文件中该特征出现的总次数),显然,这个词频越大说明该特征越重要,相应地权重就越大。这样给每个hash后的特征乘上这个权重,得到第三列。
(4)合并
将得到的每个乘以相应权重的特征累加起来,各个数位各自相加。这样,得到最右下角的一个对源文件的特征表达。
(5)降维
对于上步骤中得到的特征表达,大于0的另其为1,小于0的另其为0,这样得到最终降维结果:[1,0,1,0,1,1] 。
思考一个问题:如果去掉特征“灰色”,这样的话对最后的结果会有影响吗?
分析下:特征“灰色”hash并加权后得[4,-4,4,4,4,4],如果去掉该特征,那么累加后特征变为[30,-22,38,-9,51,9],最后对应到降维的结果依然是[1,0,1,0,1,1]没变。
所以,只要那些权重很大(很重要)的特征才会影响到最后的结果。
4.min hash最小哈希
A和B向量中的非零值个数分别为 a和b,共同的非零值个数为 c,则Jaccard相似度可定义为 jaccard(A,B)=c/(a+b)
当a,b的值较大的话,计算Jaccard相似度的复杂度也是线性增长的,如何减小这个计算复杂度就是MinHash想要去解决的问题。简单来说,MinHash所做的事情就是:将向量A、B映射到一个低维空间,并且近似保持A、B之间的相似度。
要理解这个等式,可以考虑向量A,B每一行的取值可以分为三类:
A和B在这一行上的取值均为1
A和B在这一行上一个为1,一个为0
A和B在这一行上的取值均为0
对于稀疏向量而言,大部分行都是属于第3类,而这种情况对等式两边都没有影响。
假设第1类和第2类情况的数量分别为x和y,那么容易得到等式右边jaccard(a,b)=x/(x+y) 。
对于等式左边,如果permutation是随机的话,那么向量A,B从上往下找,遇到的第一个非零行的情况属于第一类的概率也应为jaccard(a,b)=x/(x+y) ,从而上面的等式成立。
假设我们对向量A,B做m次permutation(m一般为几百或更小,通常远小于原向量的长度n),每一次permutation得到MinHash值的映射记为hi(A) ,那么向量A,B就分别被转换为两个signature向量:
这样只要计算这两个signature向量MinHash值相等的比例,即可以估计原向量A,B的Jaccard相似度。
上面理解Min Hashing的方式虽然很直观,但是在计算上却是很难实现:当n很大时,做m次permutation的时间复杂度是很高的。
通常我们可以使用一个针对row index的哈希函数来达到permutation的效果,虽然可能会有哈希碰撞的情况产生,但是只要碰撞的概率不大,对估计的结果没有大的影响。
可以通过一些随机哈希函数来模拟行打乱的效果。
哈希函数的选择,可以参考Spark中Min Hashing算法的实现,这里将核心代码提取如下:
import org.apache.spark.mllib.linalg.SparseVector
import scala.util.Random
/**
* @param hashNum 签名向量的维度, hash函数的个数
*/
class MinHash(hashNum: Int) extends Serializable {
val HASH_PRIME=2038074743
val rand = new Random()
/**
* n个随机哈希函数的参数配置
*/
val randCoefs: Array[(Int, Int)] = Array.fill(hashNum) {
(1 + rand.nextInt(HASH_PRIME - 1), rand.nextInt(HASH_PRIME - 1))
}
def generateSignature(vector: SparseVector): Array[Int] = {
val indexes = vector.indices
val signatureVector = randCoefs.map {
case (a, b) =>
indexes.map(index => ((1 + index) * a + b) % HASH_PRIME).min
}
signatureVector
}
}
5.LSH – 局部敏感哈希
用于解决在高维空间中查找相似节点的问题。
如果直接在高维空间中进行线性查找,将面临维度灾难,效率低下,LSH的作用就是把原来高维空间上的点都映射到一个或多个hashtable的不同的位置上,这个位置术语上称作桶(buckets)。
它映射的原则是:原来在高维空间中就很接近的点,会以很大的概率被映射到同一个桶中。这样,如果再给你一个高维空间上的点,你只需要按照同样的方式也把这个点映射到一个桶中,而在同一个桶中点都是有很大概率在原来高维空间中是相似的,这样就可以直接对这个桶中的元素进行查找即可,大大的提高了查找的效率。
如何保证原来高维空间中相近的点以很大的概率被映射到同一个桶中:
LSH的做法是在原来的高维空间中随机均匀的画很多个平面,具体有多少个可以用一个参数k来表示。
高维空间中的每一点和这些平面就会有一个位置划分关系,比如点在平面上还是在平面下,分别对应1和0,这样每一个点就会形成一个长度为k的一个编码,被叫做汉明编码(hamingcode)
原来高维空间中很接近的点,它们对应的汉明编码也应该大致相同。
如果把每一个汉明编码看作是一个桶,这样就相当于把原始高维空间中的相近的点以一个很大的概率都映射到了同一个桶里面了。
这个概率具体有多大呢,这就和原始空间被划分的细致程度有关了,也就是平面的个数k,这个k越大,对应的所有可能的汉明编码数量也就是2k个,也就是桶的个数为2k个。
LSH的具体做法是在Min Hashing所得的signature向量的基础上,将每一个向量分为几段,称之为band
其基本想法是:如果两个向量的其中一个或多个band相同,那么这两个向量就可能相似度较高;相同的band数越多,其相似度高的可能性越大。
所以LSH的做法就是对各个用户的signature向量在每一个band上分别进行哈希分桶(如md5,sha1等),在任意一个band上被分到同一个桶内的DOC就互为candidate,这样只需要计算所有query相似度就可以找到最相似的了
另外, 需要注意的是,每一层的band只能和同一层的band相比,若hash值相同,则放入同一个哈希桶中。
6.常用的LSH增强的方法
1 使用多个独立的hashtable
每个hashtable由k个LSH hash function创建,每次选用k个LSH hash function(同属于一个LSH function family)就得到了一个hash table,重复多次,即可创建多个hash table.
多个的好处在于能够降低false negtive【不相同的被认为相同】
2 AND与操作
从同一个LSH function family中挑选出k个LSH hash function
H(X)=H(Y)有且仅当这k个Hi(X)=Hi(Y)都满足。也就是说只有当两个数据的这k个hash值都对应相同时,才会被投影到相同的桶内,只要有一个不满足就不会被投影到相同的桶内
能够降低false negtive
3 OR操作。
降低 false positive
4 AND 和OR的级联
AND then OR ,它是先要求每个band的所有对应元素必须都相同,再要求多个band中至少有一个相同。符合这两条,才能发生hash碰撞。
5 求模运算
new hash value = old hash value % N
6 随机投影
new hash value = h1r1 + h2r2 + … + hk*rk, 其中r1,r2,rk是一些随机数
7 XOR异或
new hash value = h1 XOR h2 XOR h3 … XOR hk
7.LSH分桶优化
下面我们对signature向量的分桶概率作一些数值上的分析,以便针对具体应用确定相应的向量分段参数。
假设我们将signature向量分为b个band,每个band的大小(也就是band内包含的行数)为r。
假设两个doc向量之间的Jaccard相似度为s,前面我们知道signature向量的任意一行相同的概率等于Jaccard相似度s,我们可以按照以下步骤计算两个query成为candidate的概率:
如果想要尽可能少的出现false negative,就需要选择b和r使得概率变化最陡的地方小于 Ssim。
例如假设我们认为s在0.5以上才属于相似doc,那么我们就要选择b和r使得S曲线的最陡处小于0.5(上图所示的b=100,r=4就是一个较好的选择),这样的话,s在0.5以上的“真正”的相似doc就会以很大的概率成为candidate。
如果想要保证计算速度较快,并且尽可能少出现false positive,那么最好选择b和r使得概率变化最陡的地方较大
例如下图所示的b=20,r=6。这样的话,s较小的两个doc就很难成为candidate,但同时也会有一些“潜在”的相似doc不会被划分到同一个桶内。(candidate是一部分质量较高的相似doc)
8.LSH开源实现
关于LSH开源工具库,有很多,这里推荐两个LSH开源工具包:LSHash和FALCONN, 分别对应于学习和应用场景。
8.1 LSHash
LSHash非常适合用来学习,里面实现的是最经典的LSH方法,并且还是单表哈希。哈希函数的系数采用随机的方式生成,具体代码如下:
def _generate_uniform_planes(self):
“”" Generate uniformly distributed hyperplanes and return it as a 2D
numpy array.
“”"
return np.random.randn(self.hash_size, self.input_dim)
hash_size为哈希函数的数目,即前面介绍的K。整个框架,不论是LSH的哈希函数的生成方式,还是LSH做查询,都极其的中规中矩,所以用来作为了解LSH的过程,再适合不过。如果要在实用中使用LSH,可以使用FALCONN。
8.2 FALCONN
FALCONN是经过极致优化的LSH,
其对应的论文为NIPS 2015 Practical and Optimal LSH for Angular Distance,
Piotr Indyk系作者之一(Piotr Indyk不知道是谁?E2LSH这个页面对于看过LSH的应该非常眼熟),
论文有些晦涩难懂,不过FALCONN工具包却是极其容易使用的,提供有C++使用的例子random_benchmark.cc以及Python的例子random_benchmark.py,
另外文档非常的详实,具体可参阅falconn Namespace Reference和falconn module。
下面将其Python例子和C++例子中初始化索引以及构建哈希表的部分提取出来,对其中的参数做一下简要的分析。
Python初始化与构建索引L127:
// Hyperplane hashing
params_hp = falconn.LSHConstructionParameters()
params_hp.dimension = d
params_hp.lsh_family = ‘hyperplane’
params_hp.distance_function = ‘negative_inner_product’
params_hp.storage_hash_table = ‘flat_hash_table’
params_hp.k = 19
params_hp.l = 10
params_hp.num_setup_threads = 0
params_hp.seed = seed ^ 833840234
print(‘Hyperplane hash\n’)
start = timeit.default_timer()
hp_table = falconn.LSHIndex(params_hp)
hp_table.setup(data)
hp_table.set_num_probes(2464)
C++初始化与构建索引L194:
// Hyperplane hashing
LSHConstructionParameters params_hp;
params_hp.dimension = d;
params_hp.lsh_family = LSHFamily::Hyperplane;
params_hp.distance_function = distance_function;
params_hp.storage_hash_table = storage_hash_table;
params_hp.k = 19;
params_hp.l = num_tables;
params_hp.num_setup_threads = num_setup_threads;
params_hp.seed = seed ^ 833840234;
cout << “Hyperplane hash” << endl;
Timer hp_construction;
unique_ptrhptable(
std::move(construct_table(data, params_hp)));
hptable->set_num_probes(2464);
可以看到,有3个很重要的参数,分别是k、l和set_num_probes,对应的具体意义前面已经解释,这里不再赘述。FALCONN的索引构建过程非常快,百万量级数据,维度如果是128维,其构建索引时间大概2-3min的样子,实时搜索可以做到几毫秒响应时间。
另外谈一下数据规模问题。对于小数据集和中型规模的数据集(几个million-几十个million), FALCONN和NMSLIB是一个非常不错的选择,如果对于大型规模数据集(几百个million以上),基于矢量量化的Faiss是一个明智的选择。对于这方面的讨论,可以参阅讨论benchmark。
当然,FALCONN还不是很完善,比如对于数据的动态增删目前还不支持,具体的讨论可以参见Add a dynamic LSH table。其实这不是FALCONN独有的问题,NMSLIB目前也不支持。一般而言,动态的增删在实际应用场合是一个基本的要求,但是我们应注意到,增删并不是毫无限制的,在增删频繁且持续了一段时间后,这时的数据分布已经不是我们原来建索引的数据分布形式了,我们应该重新构建索引。在这一点上,基于矢量量化的方法对数据的动态增删更友好。
通常而言,哈希向量量化方法比矢量量化方法,在召回率上要差一些。一个比较直观的理解是:哈希向量量化后在计算距离的时候,计算的是汉明距离,在向量量化比特位长度相同的条件下,汉明距离表示的距离集合是有限的,而矢量量化计算的距离是一个实数,意味着它构成的距离集合是无限的。
代码-
方法一:引用python包datasketch
安装:pip install datasketch
使用示例如下:
MinHash
from datasketch import MinHash
data1 = ['minhash', 'is', 'a', 'probabilistic', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'datasets']
data2 = ['minhash', 'is', 'a', 'probability', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'documents']
m1, m2 = MinHash(), MinHash()
for d in data1:
m1.update(d.encode('utf8'))
for d in data2:
m2.update(d.encode('utf8'))
print("Estimated Jaccard for data1 and data2 is", m1.jaccard(m2))
s1 = set(data1)
s2 = set(data2)
actual_jaccard = float(len(s1.intersection(s2)))/float(len(s1.union(s2)))
print("Actual Jaccard for data1 and data2 is", actual_jaccard)
MinHash LSH
from datasketch import MinHash, MinHashLSH
set1 = set(['minhash', 'is', 'a', 'probabilistic', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'datasets'])
set2 = set(['minhash', 'is', 'a', 'probability', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'documents'])
set3 = set(['minhash', 'is', 'probability', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'documents'])
m1 = MinHash(num_perm=128)
m2 = MinHash(num_perm=128)
m3 = MinHash(num_perm=128)
for d in set1:
m1.update(d.encode('utf8'))
for d in set2:
m2.update(d.encode('utf8'))
for d in set3:
m3.update(d.encode('utf8'))
# Create LSH index
lsh = MinHashLSH(threshold=0.5, num_perm=128)
lsh.insert("m2", m2)
lsh.insert("m3", m3)
result = lsh.query(m1)
print("Approximate neighbours with Jaccard similarity > 0.5", result)
MinHash LSH Forest——局部敏感随机投影森林
from datasketch import MinHashLSHForest, MinHash
data1 = ['minhash', 'is', 'a', 'probabilistic', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'datasets']
data2 = ['minhash', 'is', 'a', 'probability', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'documents']
data3 = ['minhash', 'is', 'probability', 'data', 'structure', 'for',
'estimating', 'the', 'similarity', 'between', 'documents']
# Create MinHash objects
m1 = MinHash(num_perm=128)
m2 = MinHash(num_perm=128)
m3 = MinHash(num_perm=128)
for d in data1:
m1.update(d.encode('utf8'))
for d in data2:
m2.update(d.encode('utf8'))
for d in data3:
m3.update(d.encode('utf8'))
# Create a MinHash LSH Forest with the same num_perm parameter
forest = MinHashLSHForest(num_perm=128)
# Add m2 and m3 into the index
forest.add("m2", m2)
forest.add("m3", m3)
# IMPORTANT: must call index() otherwise the keys won't be searchable
forest.index()
# Check for membership using the key
print("m2" in forest)
print("m3" in forest)
# Using m1 as the query, retrieve top 2 keys that have the higest Jaccard
result = forest.query(m1, 2)
print("Top 2 candidates", result)
方法二
minHash源码实现如下:
from random import randint, seed, choice, random
import string
import sys
import itertools
def generate_random_docs(n_docs, max_doc_length, n_similar_docs):
for i in range(n_docs):
if n_similar_docs > 0 and i % 10 == 0 and i > 0:
permuted_doc = list(lastDoc)
permuted_doc[randint(0,len(permuted_doc))] = choice('1234567890')
n_similar_docs -= 1
yield ''.join(permuted_doc)
else:
lastDoc = ''.join(choice('aaeioutgrb ') for _ in range(randint(int(max_doc_length*.75), max_doc_length)))
yield lastDoc
def generate_shingles(doc, shingle_size):
shingles = set([])
for i in range(len(doc)-shingle_size+1):
shingles.add(doc[i:i+shingle_size])
return shingles
def get_minhash(shingles, n_hashes, random_strings):
minhash_row = []
for i in range(n_hashes):
minhash = sys.maxsize
for shingle in shingles:
hash_candidate = abs(hash(shingle + random_strings[i]))
if hash_candidate < minhash:
minhash = hash_candidate
minhash_row.append(minhash)
return minhash_row
def get_band_hashes(minhash_row, band_size):
band_hashes = []
for i in range(len(minhash_row)):
if i % band_size == 0:
if i > 0:
band_hashes.append(band_hash)
band_hash = 0
band_hash += hash(minhash_row[i])
return band_hashes
def get_similar_docs(docs, n_hashes=400, band_size=7, shingle_size=3, collectIndexes=True):
hash_bands = {}
random_strings = [str(random()) for _ in range(n_hashes)]
docNum = 0
for doc in docs:
shingles = generate_shingles(doc, shingle_size)
minhash_row = get_minhash(shingles, n_hashes, random_strings)
band_hashes = get_band_hashes(minhash_row, band_size)
docMember = docNum if collectIndexes else doc
for i in range(len(band_hashes)):
if i not in hash_bands:
hash_bands[i] = {}
if band_hashes[i] not in hash_bands[i]:
hash_bands[i][band_hashes[i]] = [docMember]
else:
hash_bands[i][band_hashes[i]].append(docMember)
docNum += 1
similar_docs = set()
for i in hash_bands:
for hash_num in hash_bands[i]:
if len(hash_bands[i][hash_num]) > 1:
for pair in itertools.combinations(hash_bands[i][hash_num], r=2):
similar_docs.add(pair)
return similar_docs
if __name__ == '__main__':
n_hashes = 200
band_size = 7
shingle_size = 3
n_docs = 1000
max_doc_length = 40
n_similar_docs = 10
seed(42)
docs = generate_random_docs(n_docs, max_doc_length, n_similar_docs)
similar_docs = get_similar_docs(docs, n_hashes, band_size, shingle_size, collectIndexes=False)
print(similar_docs)
r = float(n_hashes/band_size)
similarity = (1/r)**(1/float(band_size))
print("similarity: %f" % similarity)
print("# Similar Pairs: %d" % len(similar_docs))
if len(similar_docs) == n_similar_docs:
print("Test Passed: All similar pairs found.")
else:
print("Test Failed.")
参考:
https://blog.csdn.net/weixin_43461341/article/details/105603825
https://zhuanlan.zhihu.com/p/46164294