- 临床研究论文 COX 比例风险回归模型
- Fit Proportional Hazards Regression Model
- 如果如下内容有错误,请留言,谢谢,感激不尽!
1 写在前面
- 本文需要的R包:"survival" 、"tableone"、"broom"、"forestplot"和 "survminer".
- 本代码使用survival自带的lung数据集,lung数据集中出现的变量解释:

2 安装拓展包
install.packages("survival")install.packages("survminer")install.packages("forestplot")install.packages("tableone")install.packages("broom")# 不需要的包可以不安装
3 调用拓展包
library(survival) # 生存分析需要library(survminer) # 作图使用ggplot画library(forestplot) # 画森林图library(tableone) library(broom) # 后面tidy函数需要data("lung") head(lung) # 预览 lung 数据集信息

4 Kaplan-Meier曲线
4.1 统计分析
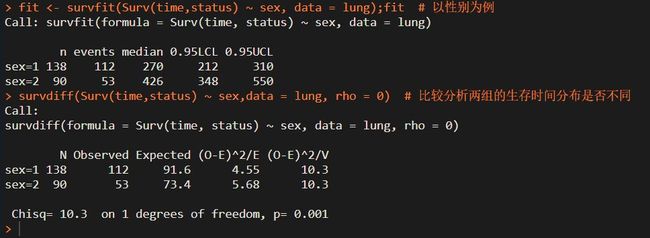
fit survdiff(Surv(time,status) ~ sex,data = lung, rho = 0) # 比较分析两组的生存时间分布是否不同# rho = 0 表示使用long-rank检验或者Mantel-Haenszel 检验)
4.2 ggsurvplot绘图及美化
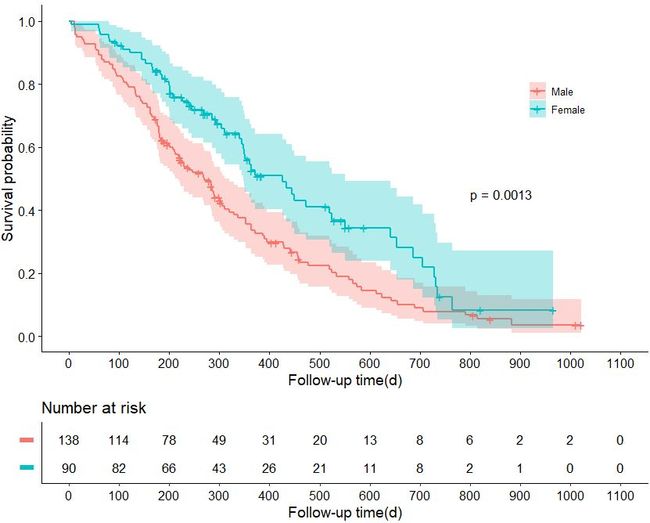
ggsurvplot(fit, data = lung, pval = TRUE, # 显示P值,默认P值检验为 log-rank test. pval.coord = c(800,0.45), #自定义P值在图中的坐标(x,y),可自行调整 conf.int = TRUE, # 图中输出95%CI risk.table = TRUE, # 输出risk table,“FALSE”为不显示; risk.table.height = 0.25, # 调整 risk table的高度,默认为0.25 risk.table.y.text = FALSE, # 不显示risk table分组文字,用颜色替代 risk.table.y.text.col=TRUE, # risk table文字颜色 censor.shape = "+", #可删除 surv_median.line = "hv", #输出中位生存期 xlim = c(0,1100), break.x.by = 100, # 自定义X轴范围和间距,自行调整 ylim = c(0,1), break.y.by = 0.2, # 自定义y轴范围和间距,自行调整 xlab = "Follow-up time(d)", # 定义 X 轴标签 font.x = c(14,"black"), # x轴字体,颜色 font.y = c(14,"black"), # y轴字体,颜色 font.tickslab = c(12, "plain", "black"), # 标签字体,样式,颜色 legend = c(0.85,0.75), # 图例坐标位置(x,y),X,Y轴值都在0-1之间 legend.title = "", # 图例标题,在双引号中输入文字 legend.labs = c("Male", "Female")) # 数据框中 sex 分组
生成KM曲线:
5 单因素Cox回归分析
5.1 单个变量进行单因素Cox回归分析
res.cox1 summary(res.cox1) # 生成完整的结果报告

- 最下面 likehood ration test , wald test, logrank test三种检验方法的p值,p值小于0.05, 说明回归方程是有统计学意义的。然后看自变量的p值,最后查看自变量的coef等指标,coef就是偏回归系数,exp(coef)就是HR。
5.2 批量进行单因素Cox生存分析
covariates univ_formulas function(x) as.formula(paste('Surv(time, status)~', x)))univ_formulas

univ_models univ_models # 输出每个单因素COX回归分析结果
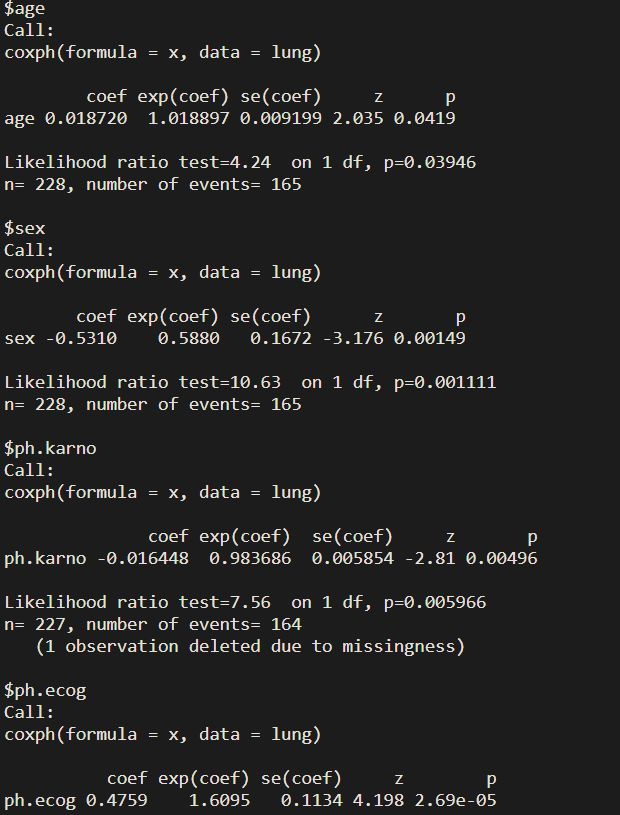
提取出分析结果: 创建提取批量分析结果的函数,提取分析结果,转换为数据框输出 注意是列表元素的提取,exp(coef)指的是HR,beta值是系数
univ_results function(x){ x p.value wald.test beta HR HR.confint.lower HR.confint.upper HR HR.confint.lower, "-", HR.confint.upper, ")") res names(res) return(res) #return(exp(cbind(coef(x),confint(x)))) })class(univ_results)str(univ_results)

res as.data.frame(res)
提取结果:

6 多因素Cox回归分析
6.1 纳入筛选后的变量进入 Cox 回归模型
res.cox summary(res.cox) # 查看con回归模型结果
结果:

- 结果解释:先看最下面likehood ration test , wald test, logrank test三种检验方法的p值,p值小于0.05, 说明回归方程是有统计学意义的。然后看每个自变量的p值,最后查看自变量的coef等指标,coef就是偏回归系数,exp(coef)就是 HR。
6.2 导出数据
table1 printToggle = TRUE, quote = FALSE, ciFun = confint)table2table3write.csv(table3,file="table3.csv") # 数据导出
导出数据为csv格式,可以用excel打开

6.3 构建Cox模型后检验模型是不是等比例模型
res.cox1
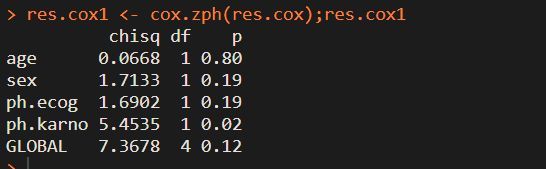
- 检验结果中 ph.karno 具有显著性(p<0.05),说明 ph.karno 这一自变量不符合“等比性”要求,这一回归模型不是等比例模型。
6.4 使用ggcoxzph函数还可以将其进行可视化
tempggcoxzph(temp)
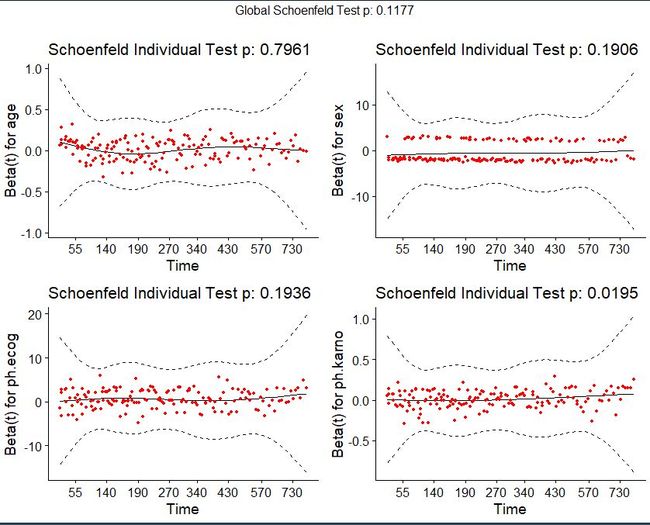
嗯,这张图片我也没看懂表达啥意思
6.5 当某一因素不符合要求时,将此因素分层分析
cox3 res.cox2 res.cox2 # 查看结果
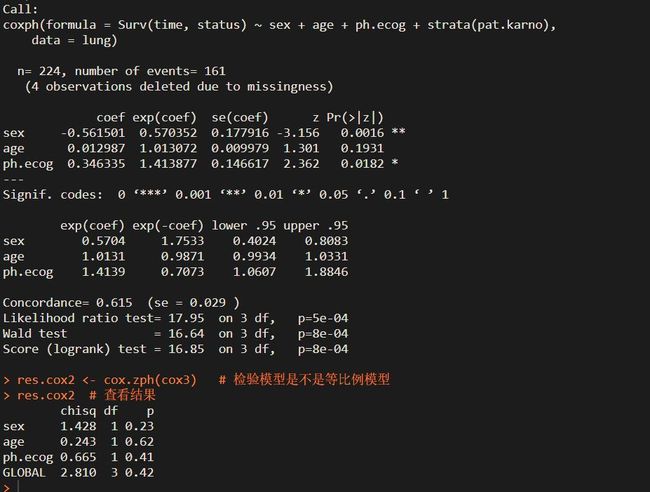
- 可以看到 P 值都>0.01,说明该模型能够通过PH检验。
7 森林图怎么画
ggforest(res.cox, data = lung, main="hazard ratio", cpositions=c(0.02,0.22,0.4), fontsize=0.8, refLabel="reference", noDigits=2)
森林图:

森林图参数调整的不多,下次在画好看点。
End

往期: R语言统计与绘图:KM曲线绘制 R语言统计与绘图:KM生存曲线+risk table 代码+注释 R语言统计与绘图:临床论文中基线特征表1怎么做?















 往期: R语言统计与绘图:KM曲线绘制 R语言统计与绘图:KM生存曲线+risk table 代码+注释 R语言统计与绘图:临床论文中基线特征表1怎么做?
往期: R语言统计与绘图:KM曲线绘制 R语言统计与绘图:KM生存曲线+risk table 代码+注释 R语言统计与绘图:临床论文中基线特征表1怎么做?