安装ribotree-利用蒙特卡洛搜索树的RNA设计工具
目录
- 1 安装依赖
-
- 1.1 安装arnie
- 1.2 安装DegScore
- 1.3 安装RiboGraphViz
- 1.4 安装biopython
- 1.5 安装Eternafold
- 1.6 安装linearfold和linearpartition
- 2 测试依赖
- 3 运行ribotree
- 4 附录
1 安装依赖
1.1 安装arnie
arnie用于管理各种RNA热动力学折叠的软件,下载连接:https://github.com/DasLab/arnie
(1)将文件夹名字arnie-marster改为arnie,~/.bashrc中添加以下行:
export PYTHONPATH=$PYTHONPATH:/opt/ribotree_1.1.7/arnie
(2)修改arnie/example_arnie_file.txt,需要对vienna_2,contrafold_2,eternafold,linearfold,linearpartition进行定义,下图先给出完整的配置,未安装的设置为None。
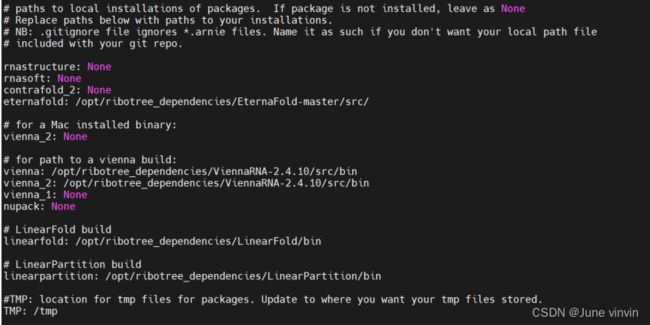
注意这里添加vienna,不然后续运行examples选择< -package vinenna >会出错。
(3)同时需要对这个文件的作为环境变量写入~/.bashrc中:
export ARNIEFILE=“opt/ribotree_1.1.7/arnie/example_arnie_file.txt”
1.2 安装DegScore
下载链接:https://github.com/eternagame/DegScore
将文件夹重命名为DegScore,~/.bashrc中添加以下行:
export PYTHONPATH=$PYTHONPATH:/opt/ribotree_dependencies/DegScore
1.3 安装RiboGraphViz
RiboGraphViz主要是对RNA结构进行可视化,下载链接:https://github.com/DasLab/RiboGraphViz
pip install -r requirements.txt
python setup.py install
#在jupyter notebook中如果要看到可视化的话可以安装pygraphviz包
conda install —channel conda-forge pygraphviz
1.4 安装biopython
pip install bio -i https://pypi.tuna.tsinghua.edu.cn/simple
1.5 安装Eternafold
https://eternagame.org/software
cd EternaFold-master/src
make
需要把对应目录添加到arnie/example_arnie_file.txt中,如下:
eternafold: /opt/ribotree_dependencies/EternaFold-master/src/
1.6 安装linearfold和linearpartition
git clone https://github.com/LinearFold/LinearFold.git
cd LinearFold
make
git reset --hard 260c6bbb9bf8cc84b807fa7633b9cb731e639884
cd ..
git clone https://github.com/LinearFold/LinearPartition.git
cd LinearPartition
make
git reset --hard 694b70a4d842af543d3f135af212fcb14a668bfd
需要把对应目录添加到arnie/example_arnie_file.txt中,如下:
# LinearFold build
linearfold: /opt/ribotree_dependencies/LinearFold/bin
# LinearPartition build
linearpartition: /opt/ribotree_dependencies/LinearPartition/bin
为eternafold和linearfold&linearpartition建立连接(注意修改EternaFold路径)
cd LinearFold
git apply --whitespace=fix /opt/ribotree_dependencies/EternaFold-master/LinearFold-E.patch
make
cd ..
cd LinearPartition
git apply --whitespace=fix /opt/ribotree_dependencies/EternaFold-master/LinearPartition-E.patch
make
cd ..
2 测试依赖
python test_dependencies.py
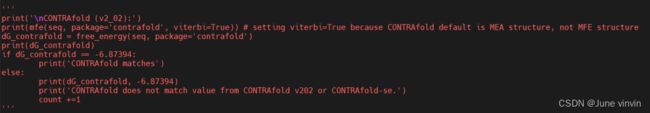
(1)注释关于contrafold的test,原因是:这个软件太老了,make都不行,连网页版都运行不了,这个软件也不会去用它。
![]()
3 运行ribotree
(1)如下图所示,选择对应的ribotree的代码进行下载,本文这里选择的是1.1.7的版本。https://eternagame.org/software
(2)安装scipy:pip install scipy -i https://pypi.tuna.tsinghua.edu.cn/simple
(3)运行:python ribotree.py --file input/example.txt
输出2个文件:
example.2023xxxx-xxxxxx-xxxxx.RUNNING_BEST.txt
包含每X次迭代收集一次最佳序列的序列,由“步长”参数确定,计算所有度量。
example.2023xxxx-xxxx-xxxxx.FULL.txt
包含蒙特卡洛运行中接受的所有序列以及损失函数值。
可以修改input/example.txt中的**-package**,支持vinenna和eternafold
4 附录
依赖目录:
---opt
---ribotree_1.1.7
---arnie
---example_arnie_file.txt
---...
---input
---ribotree.py
---test_dependencies.py
---...
---ribotree_dependencies
---DegScore
---EternaFold-master
---LinearFold
---LinearPartition
---RiboGraphViz-master
---ViennaRNA-2.4.10
参考链接:https://zhuanlan.zhihu.com/p/514096955
