利用 bioconda 管理生物信息软件
1 了解 conda,anaconda,miniconda,bioconda
1.1 conda
conda是一个软件模块管理工具,也是一个可执行命令,其核心功能是包管理与环境管理,可以用来管理Python, R, Ruby, Lua, Scala, Java, JavaScript, C/ C++, FORTRAN等语言的模块。在python中使用比较多,有点类似于pip工具。
conda的用途:
- 快速安装、运行和升级包及其依赖项
- 在计算机中便捷地创建、保存、加载和切换环境
1.2 anaconda
anaconda是一个开源的Python发行版本,包含了conda、Python等180多个科学包及其依赖项。
Anaconda具有如下特点:
- 开源
- 安装过程简单
- 高性能使用Python和R语言
- 免费的社区支持
其特点的实现主要基于Anaconda拥有的:
- conda包
- 环境管理器
- 1,000+开源库
1.3 miniconda
通过以上对anaconda的介绍,我们了解到anaconda的数据包和安装包都很大,但我们用不到那么多,那么,miniconda登场!前者安装包较大,包含了较多默认安装包,而miniconda属于精简版本,可按照自己需求进行安装,推荐安装miniconda。
1.4 bioconda
bioconda是一个管理生物信息软件的工具软件,它基于anaconda可以进行生物软件的搜索、下载、安装、升级、删除等操作,可以将 bioconda 当成生物信息软件的AppStore。
bioconda的使用首先需要安装miniconda,在安装好miniconda之后,添加相应的生物信息软件相关的channel之后,就是bioconda了。换言之,bioconda就是一种生物定制版本的conda。bioconda是目前已知最好的生物软件管理工具之一,目前已经支持超过 7000 多款生物软件的安装。
bioconda的优点:
- 自动安装依赖,解决软件依赖问题,实现生物软件一键安装,再也不用体会源代码编译之苦了;
- 除了生信类软件,R 语言、Python语言软件包都可以进行安装;
- 方便进行管理和升级,软件版本可选择,同一个软件可以选择安装任意的版本;
- 使用普通用户进行安装,不需要管理员帮忙安装依赖软件;
- 创建环境,例如需要使用Python 3,但默认是Python 2.7,可以创建一个Python 3环境;
- 所有软件安装在同一目录下,方便进行管理和迁移。
2 bioconda的安装
2.1 miniconda的安装
## 安装miniconda
curl -O https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh # 下载miniconda安装包(官网即可获得对应系统的installer)
sh Miniconda3-latest-Linux-x86_64.sh # 运行安装脚本进行安装
source ~/.bashrc # 刷新配置
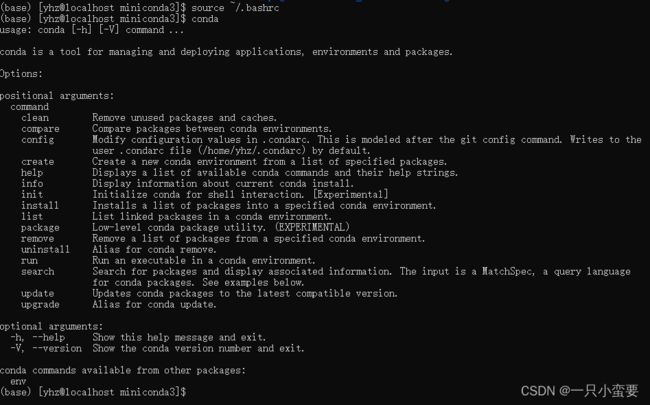
conda # 输入conda命令,弹出帮助信息,则说明安装成功
2.2 添加channel
刚才安装的只是miniconda,conda默认的channel是用来管理python模块的,必须添加bioconda这个channel才能下载和安装生物软件。
- 自用小tips
channel就是软件包存在的位置(类似于其余语言的CPAN, CRAN, Bioconductor),conda install yyy 之所以能成功的前提,是软件 yyy 已经在某个channel里。
## 添加channel
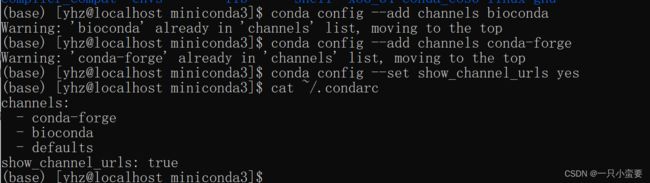
conda config --add channels bioconda
conda config --add channels conda-forge # conda-forge包含许多bioconda中尚不存在的包
conda config --set show_channel_urls yes # 显示软件来源于哪个channel
镜像
eg. 清华镜像channel: https://mirrors.tuna.tsinghua.edu.cn/anaconda/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
conda config --set show_channel_urls yes
- 小tips
3 利用bioconda管理软件
3.1 哪些软件可以用conda安装
- 网站查询
https://anaconda.org/
https://bioconda.github.io/ conda search xxx # 搜索软件是否存在

- 关键词搜索
搜索引擎直接xxx conda
3.2 软件管理
## 利用bioconda管理软件
conda activate rnaseq # 启动环境
conda install -y fastqc=0.11.7 # 安装软件,默认安装最新版本,-y 表示后面选项均选yes,即跳过确认步骤
conda install bwa samtools=1.9 bcftools fastp -y # 也可多个软件一起安装
- 小tips
安装时找不到这个包咋整?- 首先确认conda是否可以安装所需软件
- 确认是不是粗心大意打错名称
- 确认所需软件在conda中的名称
sratoolkit —— sra-tools
conda list # 查看当前环境所安装的软件
conda list fast* # 查看符合正则表达式的软件
conda list -n rnaseq # 查看指定环境下的软件
conda remove fastqc # 卸载软件
conda remove -n rnaseq fastqc # 卸载特定环境下的特定软件,如不指定-n参数,则需进入该环境之后才能进行卸载操作
conda update bwa # 更新软件
conda update conda # 升级conda本身
3.3 配置环境
3.3.1 why?
- conda会改变原来设置好的环境
- 不同软件的依赖会互相冲突
A软件依赖Python 2.7,B软件依赖Python 3.8 - 方便项目管理
基因组、转录组、Chip-seq …
- 小tips
base环境里装包的时候要了解这个包对原生环境会造成什么影响(如果它在所有小环境都可以用的话,或许可以考虑装在base环境下,新手谨慎)。
3.3.2 创建独立环境
## 创建独立环境
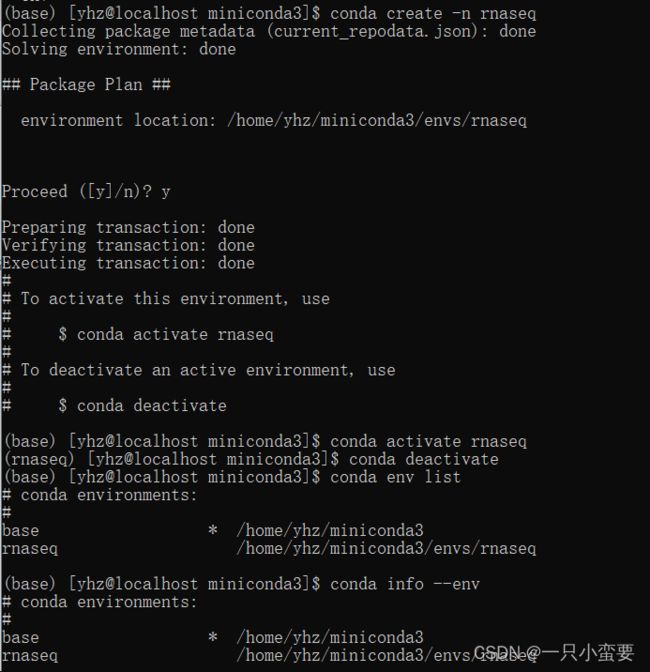
conda create -n rnaseq # 创建名为rnaseq的conda环境,-n 表示指定环境名称
conda activate rnaseq # 启动rnaseq环境
conda deactivate # 退出rnaseq环境
conda env list # 列出现有独立环境
conda info --env
conda remove -n rnaseq --all # 删除已创建环境及安装的包
conda create -n test1 bwa=0.7.16 blast=2.7.1 # 也可直接创建所需环境
conda create -n Python2.7
conda create -n py27 --clone Python2.7 # 环境重命名
conda remove -n Python2.7 --all
3.4 conda常用命令小结
3.4.1 环境管理
conda env list
conda create -n xxx
conda activate xxx
conda deactivate
3.4.2 软件管理
conda search xxx
conda install xxx
conda remove xxx
conda update xxx
conda list
4 conda大tips
4.1 mamba的使用
如果你曾遇到以下问题:
conda下载软件速度慢?
一直耗费时间处理环境问题?
环境问题处理失败?
那么,mamba绝对可以让你的软件安装过程变得过分丝滑!

mamba并没有完全重新把conda重新写一遍,而是加速了其中的一些线速度的步骤。
- 对
conda install进行改造,使其可以多线程并行下载 - 增加了一些新功能
4.1.1 mamba的安装与使用
首先切换至base环境,确保在base环境下安装mamba。
- 小tips
装在base环境中的包在其他小环境中可以调用,无需在每个环境下都安装一次。
## mamba的安装与使用
conda activate base # 切换至base环境
conda install mamba -c conda-forge -y # 安装mamba
除了启动环境外(conda activate xxx ),其他的conda命令都可以用mamba来代替,即直接将 conda 替换为 mamba 即可。
conda activate rnaseq
mamba search samtools # 搜索软件
mamba repoquery search samtools # 这个更快
mamba install samtools # 安装软件
4.1.2 使用mamba查看依赖关系
mamba可以很方便地查看安装软件之间的依赖关系。
## 查看依赖关系
mamba repoquery depends -t fastqc # 我依赖谁
mamba repoquery whoneeds -t python # 谁依赖我
4.2 版本控制和迁移
作用:
- 发文章的时候提供版本号;
- 更换服务器、分享的时候保持软件版本不变。
## 用 conda list 的 export 功能
conda list # 查看当前环境所安装的软件
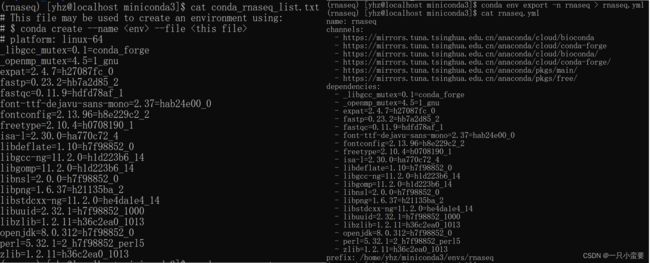
conda list -n rnaseq --export > conda_rnaseq_list.txt # 导出环境中安装的包的列表(软件名=版本号=build)
conda create -n rna -file conda_rnaseq_list.txt # 安装导出的信息
## 用 conda env 的 export 功能 —— yml文件提供的信息更全面
conda env export -n rnaseq > rnaseq.yml # 导出想要导出的环境
conda env create -f rnaseq.yml # 根据导出的yml文件创建环境
conda env update -f rnaseq.yml # 根据导出的yml文件更新环境
4.3 将软件安装到指定位置
## 将软件安装到指定位置
mkdir -p ~/biosoft/samtools # 创建相关文件夹
conda install -p ~/biosoft/samtools samtools # 使用-p参数指定安装位置
4.4 按照位置启动小环境
## 按照位置启动小环境
conda activate /home/yhz/miniconda3/envs/rnaseq # 直接指定到安装位置即可
4.5 为特定环境设置特殊频道
## 为特定环境设置特殊的channel
conda config --env --add channels genomedk
参考资料
https://www.jianshu.com/p/0534043b4471
https://www.bilibili.com/video/BV1hA411W7aC?spm_id_from=333.999.0.0
https://www.bilibili.com/video/BV1Yy4y117SX?p=15
https://www.jianshu.com/p/62f155eb6ac5