机器学习的敲门砖:kNN算法(下
plt.scatter(X2[:,0],X2[:,1])
plt.show()
计算其均值/方差
np.mean(X2[:,0])
np.std(X2[:,1])
1.4 Sklearn中的归一化
首先我们来看一个在实际使用归一化时的一个小陷阱。
我们在建模时要将数据集划分为训练数据集&测试数据集。
训练数据集进行归一化处理,需要计算出训练数据集的均值mean_train和方差std_train。
问题是:我们在对测试数据集进行归一化时,要计算测试数据的均值和方差么?
答案是否定的。在对测试数据集进行归一化时,仍然要使用训练数据集的均值train_mean和方差std_train。这是因为测试数据是模拟的真实环境,真实环境中可能无法得到均值和方差,对数据进行归一化。只能够使用公式(x_test - mean_train) / std_train
因此我们要保存训练数据集中得到的均值和方差。
在sklearn中专门的用来数据归一化的方法:StandardScaler。
下面我们加载鸢尾花数据集
import numpy as npfrom sklearn import datasetsfrom sklearn.model_selection import train_test_split
iris = datasets.load_iris()
X = iris.data
y = iris.target
X_train,X_test,y_train,y_test = train_test_split(iris.data,iris.target,test_size=0.2,random_state=666)
使用数据归一化的方法:
from sklearn.preprocessing import StandardScaler
standardScaler = StandardScaler()# 归一化的过程跟训练模型一样standardScaler.fit(X_train)
standardScaler.mean_
standardScaler.scale_ # 表述数据分布范围的变量,替代std_# 使用transformX_train_standard = standardScaler.transform(X_train)
X_test_standard = standardScaler.transform(X_test)
如此就能输出归一化后的数据了。
1.5 自己实现均值方差归一化
同样地,我们仿照sklearn的风格,可以自己实现一下均值方差归一化的方法。
我们在之前的工程中创建processing.py:
import numpy as npclass StandardScaler:
def init(self):
self.mean_ = None
self.scale_ = None
def fit(self, X):
“”“根据训练数据集X获得数据的均值和方差”“”
assert X.ndim == 2, “The dimension of X must be 2”
求出每个列的均值
self.mean_ = np.array([np.mean(X[:,i] for i in range(X.shape[1]))])
self.scale_ = np.array([np.std(X[:, i] for i in range(X.shape[1]))]) return self def tranform(self, X):
“”“将X根据StandardScaler进行均值方差归一化处理”“”
assert X.ndim == 2, “The dimension of X must be 2”
assert self.mean_ is not None and self.scale_ is not None, \ “must fit before transform”
assert X.shape[1] == len(self.mean_), \ “the feature number of X must be equal to mean_ and std_”
创建一个空的浮点型矩阵,大小和X相同
resX = np.empty(shape=X.shape, dtype=float) # 对于每一列(维度)都计算
for col in range(X.shape[1]):
resX[:,col] = (X[:,col] - self.mean_[col]) / self.scale_[col] return resX
0x02 kNN优缺点
KNN的主要优点有:
-
理论成熟,思想简单,既可以用来做分类也可以用来做回归
-
天然解决多分类问题,也可用于回归问题
-
和朴素贝叶斯之类的算法比, 《一线大厂Java面试题解析+后端开发学习笔记+最新架构讲解视频+实战项目源码讲义》无偿开源 威信搜索公众号【编程进阶路】 对数据没有假设,准确度高,对异常点不敏感
-
由于KNN方法主要靠周围有限的邻近的样本,而不是靠判别类域的方法来确定所属类别的,因此对于类域的交叉或重叠较多的待分样本集来说,KNN方法较其他方法更为适合
KNN的主要缺点有:
- 计算量大,效率低。
即使优化算法,效率也不高。
-
高度数据相关,样本不平衡的时候,对稀有类别的预测准确率低
-
相比决策树模型,KNN模型可解释性不强
-
维数灾难:
随着维度的增加,“看似相近”的两个点之间的距离越来越大,而knn非常依赖距离
| 维数 | 点到点 | 距离 |
| — | — | — |
| 1维 | 0到1的距离 | 1 |
| 2维 | (0,0)到(1,1)的距离 | 1.414 |
| 3维 | (0,0,0)到(1,1,1)的距离 | 1.73 |
| 64维 | (0,0,…0)到(1,1,…1) | 8 |
| 10000维 | (0,0,…0)到(1,1,…1) | 100 |
大家感觉一万维貌似很多,但实际上就是100*100像素的黑白灰图片。
以上就是关于kNN算法的总结。
你是不是以为这一篇就两节内容就结束了?没想到吧!下面还有一波干货:kNN优化之KD树。
3 KD树
K近邻法的重要步骤是对所有的实例点进行快速k近邻搜索。如果采用线性扫描(linear scan),要计算输入点与每一个点的距离,时间复杂度非常高。因此在查询操作是,使用kd树。
3.1 kd树的原理
kd树是一种对k维空间中的实例点进行存储以便对其进行快速检索的树形数据结构,且kd树是一种二叉树,表示对k维空间的一个划分。
k-d tree是每个节点均为k维样本点的二叉树,其上的每个样本点代表一个超平面,该超平面垂直于当前划分维度的坐标轴,并在该维度上将空间划分为两部分,一部分在其左子树,另一部分在其右子树。即若当前节点的划分维度为d,其左子树上所有点在d维的坐标值均小于当前值,右子树上所有点在d维的坐标值均大于等于当前值,本定义对其任意子节点均成立。
3.2 kd树的构建
常规的k-d tree的构建过程为:
-
循环依序取数据点的各维度来作为切分维度,
-
取数据点在该维度的中值作为切分超平面,
-
将中值左侧的数据点挂在其左子树,将中值右侧的数据点挂在其右子树,
-
递归处理其子树,直至所有数据点挂载完毕。
对于构建过程,有两个优化点:
-
选择切分维度:根据数据点在各维度上的分布情况,方差越大,分布越分散,从方差大的维度开始切分,有较好的切分效果和平衡性。
-
确定中值点:预先对原始数据点在所有维度进行一次排序,存储下来,然后在后续的中值选择中,无须每次都对其子集进行排序,提升了性能。也可以从原始数据点中随机选择固定数目的点,然后对其进行排序,每次从这些样本点中取中值,来作为分割超平面。该方式在实践中被证明可以取得很好性能及很好的平衡性。
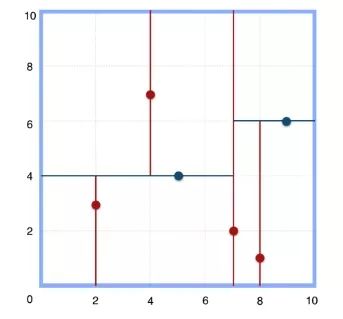
例子:
- 构建根节点时,此时的切分维度为x,如上点集合在x维从小到大排序为(2,3),(4,7),(5,4),(7,2),(8,1),(9,6);
其中值为(7,2)。
(注:
2,4,5,7,8,9在数学中的中值为(5 + 7)/2=6,但因该算法的中值需在点集合之内,所以本文中值计算用的是len(points)//2=3, points[3]=(7,2))
-
(2,3),(4,7),(5,4)挂在(7,2)节点的左子树,(8,1),(9,6)挂在(7,2)节点的右子树。
-
构建(7,2)节点的左子树时,点集合(2,3),(4,7),(5,4)此时的切分维度为y,中值为(5,4)作为分割平面,(2,3)挂在其左子树,(4,7)挂在其右子树。
-
构建(7,2)节点的右子树时,点集合(8,1),(9,6)此时的切分维度也为y,中值为(9,6)作为分割平面,(8,1)挂在其左子树。
至此k-d tree构建完成。
上述的构建过程结合下图可以看出,构建一个k-d tree即是将一个二维平面逐步划分的过程。
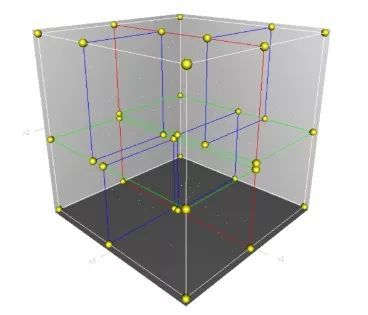
需要注意的是,对于每次切分,都是循环顺序选择维度的,二维是:x->y->x…;三维则是:x->y->z->x…。
下面从三维空间来看一下k-d tree的构建及空间划分过程。首先,边框为红色的竖直平面将整个空间划分为两部分,此两部分又分别被边框为绿色的水平平面划分为上下两部分。最后此4个子空间又分别被边框为蓝色的竖直平面分割为两部分,变为8个子空间,此8个子空间即为叶子节点。
points为实例点集合,depth深度,为用来确定取维度的参数def kd_tree(points, depth):
if 0 == len(points): return None
指定切分维度,len(points[0])是数据的实际维度,这样计算可以保证循环
cutting_dim = depth % len(points[0]) # 切分点初始化
medium_index = len(points) # 对所有的实例点按照指定维度进行排序,itemgetter用于获取对象哪些维度上的数据,参数为需要获取的数据在对象中的序号
points.sort(key=itemgetter(cutting_dim))
将该维度的中值点作为根节点
node = Node(points[medium_index])
对于左子树,重复构建(depth+1)
node.left = kd_tree(points[:medium_index], depth + 1)
对于右子树,重复构建(depth+1)
node.right = kd_tree(points[medium_index + 1:], depth + 1)