机器学习 基础理论 学习笔记 (8)特征选择(feature selection)(一)
0.说明
本文也许比较乱,请看目录再食用。
后续会 出文 机器学习 基础理论 学习笔记 (8)特征选择(feature selection)(二) 将 分类问题 和 回归问题 分开总结。
以及或将出文 机器学习 基础理论 学习笔记 (8)特征选择(feature selection)(三) 将sklearn上面实现的特征选择API和方法 总结。
1.特征选择
特征选择是特征工程里的一个重要问题,其目标是寻找最优特征子集。特征选择能剔除不相关(irrelevant)或冗余(redundant )的特征,从而达到减少特征个数,提高模型精确度,减少运行时间的目的。另一方面,选取出真正相关的特征简化模型,协助理解数据产生的过程。并且常能听到“数据和特征决定了机器学习的上限,而模型和算法只是逼近这个上限而已”,由此可见其重要性,但是它几乎很少出现于机器学习书本里面的某一章,然而在机器学习方面的成功很大程度上在于如果使用特征工程。
2.做特征选择的原因
考虑特征选择,是因为机器学习经常面临过拟合的问题。 过拟合的表现是模型参数太贴合训练集数据,模型在训练集上效果很好而在测试集上表现不好,也就是在高方差。简言之模型的泛化能力差。
即使用原始数据集的话:
- 耗时:特征个数越多,分析特征、训练模型所需的时间就越长。
- 过拟合:特征个数越多,容易引起“维度灾难”,模型也会越复杂,其推广能力会下降。
- 共线性:单因子对目标的作用被稀释,解释力下降
3.特征选择的一般过程
- 生成子集:搜索特征子集,为评价函数提供特征子集;
- 评价函数:评价特征子集的好坏;
- 停止准则:与评价函数相关,一般是阈值,评价函数达到一定标准后就可停止搜索;
- 验证过程:在验证数据集上验证选出来的特征子集的有效性;
4. 如何做特征选择
(1)方差特征选择法
过滤特征选择法有一种方法不需要度量特征 x_i 和类别标签 y 的信息量。这种方法(ANOVA)先要计算各个特征的方差,然后根据阈值,选择方差大于阈值的特征。
方差选择法用于特征选择的本质:
- 移除低方差特征 : 是指移除那些方差低于某个阈值,即特征值变动幅度小于某个范围的特征,这一部分特征的区分度较差,我们进行移除
- 考虑有值数据中的占比,异常数据的占比,正常范围数据过少的数据也可以移除。
假设我们有一个特征是由0和1组成的数据集,利用Removing features with low variance方法移除那些在整个数据集中特征值为0或者为1的比例超过p(同一类样本所占的比例)的特征。0 1 组成的数据集满足伯努利( Bernoulli )分布,因此其特征变量的方差为:p(1-p)。
在Removing features with low variance方法中,将剔除方差低于p(1-p)的特征。在Pyhon的sklearn模块中,具有该方法的实现,具体使用如下:
from sklearn.feature_selection import VarianceThreshold #导入python的相关模块
X=[[0,0,1],[0,1,0],[1,0,0],[0,1,1],[0,1,0],[0,1,1]]#其中包含6个样本,每个样本包含3个特征。
sel=VarianceThreshold(threshold=(0.8*(1-0.8)))#表示剔除特征的方差大于阈值的特征Removing features with low variance
sel.fit_transform(X)#返回的结果为选择的特征矩阵
print(sel.fit_transform(X))#
------------------------------------
或者案例:
# 方差法
from sklearn.feature_selection import VarianceThreshold
X = [[0, 2, 0, 3], [0, 1, 4, 3], [0, 1, 1, 3]]
selector = VarianceThreshold()
X_new = selector.fit_transform(X)
#
print('X_new:\n',selector.fit_transform(X))
print('get_params:\n',selector.get_params())
print('get_support:\n',selector.get_support())
print('inverse_transform:\n',selector.inverse_transform(X_new))
结果:
X_new:
[[2 0]
[1 4]
[1 1]]
get_params:
{'threshold': 0.0}
get_support:
[False True True False]
inverse_transform:
[[0 2 0 0]
[0 1 4 0]
[0 1 1 0]]
(2)相关性
单变量特征选择:单变量特征是基于单一变量和目标y之间的关系,通过计算某个能够度量特征重要性的指标,然后选出重要性Top的K个特征。但是这种方式有一个缺点就是忽略了特征组合的情况。
① 皮尔森相关系数
皮尔森相关系数是一种最简单的,能帮助理解特征和响应变量之间关系的方法,衡量的是变量之间的线性相关性,结果的取值区间为[-1,1] , -1 表示完全的负相关(这个变量下降,那个就会上升), +1 表示完全的正相关, 0 表示没有线性相关性。
公式:

Pearson相关系数的一个明显缺陷是,作为特征排序机制,他只对线性关系敏感。如果关系是非线性的,即便两个变量具有一一对应的关系,Pearson相关性也可能会接近 0 。
import numpy as np
from scipy.stats import pearsonr
np.random.seed(0)
size = 300
x = np.random.normal(0, 1, size)
print("Lower noise:", pearsonr(x, x + np.random.normal(0, 1, size)))
print("Higher noise:", pearsonr(x, x + np.random.normal(0, 10, size)))
from sklearn.feature_selection import SelectKBest
# 选择K个最好的特征,返回选择特征后的数据
# 第一个参数为计算评估特征是否好的函数,该函数输入特征矩阵和目标向量,输出二元组(评分,P值)的数组,数组第i项为第i个特征的评分和P值。在此定义为计算相关系数
# 参数k为选择的特征个数
SelectKBest(lambda X, Y: array(map(lambda x:pearsonr(x, Y), X.T)).T, k=2).fit_transform(iris.data, iris.target)
② Fisher得分
Fisher Score为过滤式的特征选择算法,是一种衡量特征在两类之间分辨能力的方法。Fisher Score是特征选择的有效方法之一, 其主要思想是鉴别性能较强的特征表现为类内距离尽可能小, 类间距离尽可能大。
公式:

其中i代表第i个特征,即每一个特征都会有一个F-score。没有 + 和 - 的 x平均值 是所有该特征值的平均数,而(+),(-)则分别代表所有阳性样本和阴性样本的特征值(的平均数)。代表k是对于具体第i个特征的每个实例,分母的两个sigma可以理解为阳性样本与阴性样本的特征值的方差。F-score越大说明该特征的辨别能力越强。
Fisher得分的改进版:

代码实现1:https://github.com/jundongl/scikit-feature/。
知识扩展:【自己点进去看】
③ 卡方检验
经典的卡方检验是检验类别型变量对类别型变量的相关性。假设自变量有N种取值,因变量有M种取值,考虑自变量等于i且因变量等于j的样本频数的观察值与期望的差距,构建统计量:

这个统计量的含义简而言之就是自变量对因变量的相关性。用sklearn中feature_selection库的SelectKBest类结合卡方检验来选择特征的代码如下:
from sklearn.datasets import load_iris
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import chi2
iris = load_iris()
X, y = iris.data, iris.target #iris数据集
#选择K个最好的特征,返回选择特征后的数据
X_new = SelectKBest(chi2, k=2).fit_transform(X, y)
sklearn中的卡方检验的两个选择函数实现的 异同:
* SelectKBest(score_func,k=)。保留评分最高得分的 K 个特征;
* SelectPercentile(score_func,percentile=)。保留最高得分的百分比特征;
④ 方差分析(ANOVA,或叫F检验)
知识补充:
sklearn.feature_selection 提供了3个接口:
- SelectKBest : 筛选k个相关性最高的特征,最常用。
- SelectPercentile : 筛选得分排名前k分位数的特征,即保留最高得分的百分比特征。
- GenericUnivariateSelect: 根据用户自定义评分函数进行筛选。
有3种常用的统计检验:
- f_classif:方差分析(ANOVA),适用于分类问题,特征是数值变量,目标是分类变量。
- chi2:差方分析,适用于分类问题,要求特征是计数或二元变量(正值)
- f_regression:适用于回归问题,特征和目标变量均为数值变量。
所有检验都返回一个分值和p-value,分值和p-value越高表示特征与目标变量的关联程度越高。
sklearn中的两个选择数据函数的实现和参数解析:
* SelectKBest(score_func,k=)。保留评分最高得分的 K 个特征;
* SelectPercentile(score_func,percentile=)。保留最高得分的百分比特征;
score_func的选择:
(1)对于回归:
f_regression:相关系数,计算每个变量与目标变量的相关系数,然后计算出F值和P值;基于线性回归分析来计算统计指标,给出各 特征的回归系数,系数比较大的特征更重要。
mutual_info_regression:互信息;计算X和y之间的互信息,以便度量相关程度,适用于回归问题。
(2)对于分类
chi2:卡方检验;计算各特征的卡方统计量,适用于分类问题。
f_classif:方差分析,计算方差分析(ANOVA)的F值 (组间均方 / 组内均方);根据方差分析(ANOVA)的原理,以F-分布为依据,利用平方和与自由度所计算的祖居与组内均方估计出F值,适用于分类问题。
mutual_info_classif:互信息,适用于分类问题。
注:chi2 , mutual_info_classif , mutual_info_regression 可以保持数据的稀疏性。
代码实现:
from sklearn.feature_selection import SelectKBest,f_classif
X=[
[1,2,3,4,5],
[5,4,3,2,1],
[3,3,3,3,3],
[1,1,1,1,1]
]
y=[0,1,0,1]
print('before transform:\n',X)
sel=SelectKBest(score_func=f_classif,k=3)
sel.fit(X,y) #计算统计指标,这里一定用到y
print('scores_:\n',sel.scores_)
print('pvalues_:',sel.pvalues_)
print('selected index:',sel.get_support(True))
print('after transform:\n',sel.transform(X))
⑤ 互信息熵
度量两个变量之间的相关性,互信息越大表明两个变量相关性越高;互信息为0,两个变量越独立。
代码实现:
from sklearn.feature_selection import SelectKBest,mutual_info_classif
X=[
[1,2,3,4,5],
[5,4,3,2,1],
[3,3,3,3,3],
[1,1,1,1,1]
]
y=[0,1,0,1]
print('before transform:\n',X)
sel=SelectKBest(score_func=mutual_info_classif,k=3)
sel.fit(X,y) #计算统计指标,这里一定用到y
print('scores_:\n',sel.scores_)
print('pvalues_:',sel.pvalues_)
print('selected index:',sel.get_support(True))
print('after transform:\n',sel.transform(X))
⑥ KL散度(也叫相对熵)
Kullback-Leibler Divergence,即K-L散度,又称相对熵,是一种量化两种概率分布P和Q之间差异的方式,又叫相对熵。在概率学和统计学上,我们经常会使用一种更简单的、近似的分布来替代观察数据或太复杂的分布。K-L散度能帮助我们度量使用一个分布来近似另一个分布时所损失的信息量。
根据计算公式
可以发现,P 和 Q 中元素的个数不用相等,只需要两个分布中的离散元素一致。
案例:
两个离散分布分布分别为 P 和 Q
P 的分布为:{1,1,2,2,3}
Q 的分布为:{1,1,1,1,1,2,3,3,3,3}
我们发现,虽然两个分布中元素个数不相同,P 的元素个数为 5,Q 的元素个数为 10。但里面的元素都有 “1”,“2”,“3” 这三个元素。
当 x = 1时,在 P 分布中,“1” 这个元素的个数为 2,故 P(x = 1) = 2525 = 0.4,在 Q 分布中,“1” 这个元素的个数为 5,故 Q(x = 1) = 510510 = 0.5
同理,
当 x = 2 时,P(x = 2) = 2525 = 0.4 ,Q(x = 2) = 110110 = 0.1
当 x = 3 时,P(x = 3) = 1515 = 0.2 ,Q(x = 3) = 410410 = 0.4
把上述概率带入公式:

代码实现:
import numpy as np
import scipy.stats
p=np.asarray([0.65,0.25,0.07,0.03])
q=np.array([0.6,0.25,0.1,0.05])
def KL_divergence(p,q):
return scipy.stats.entropy(p, q, base=2)
print(KL_divergence(p,q)) # 0.01693110139988926
print(KL_divergence(q,p)) # 0.019019266539324498
⑦距离相关系数
距离相关系数:研究两个变量之间的独立性,距离相关系数为0表示两个变量是独立的。克服了皮尔逊相关系数(Pearson)的弱点。pearson相关系数为0并不一定表示两个变量之间是独立的,也有可能是非线性相关的。
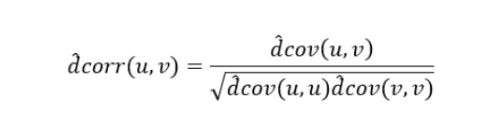
安装该实现包:pip install dcor
安装的时候可能会遇到 报错:ERROR: Cannot uninstall 'llvmlite'. It is a distutils installed project.
解决方法参考:https://blog.csdn.net/weixin_43535207/article/details/104385743
更多详细的介绍访问:https://dcor.readthedocs.io/en/latest/modules/dcor._dcor.html#b-distance-correlation
dcor的使用:
import numpy as np
a1=np.array([11,2,56,34])
b1=np.array([45,15,26,24])
dcor.distance_correlation(a1,b1)
输出:
0.6673874262718296
在网上有一个自定义距离相关系数函数的代码:https://gist.github.com/satra/aa3d19a12b74e9ab7941
其中numbapro没有这个包,把numbapro改为numba就好了。
from numba import jit, float32
(3)递归消除
递归消除:RFE, Recursive Features Elimination.
递归消除的核心思想是通过反复迭代,剔除没有预测意义的特征,与向后逐步回归非常相似,属于纯技术性的变量选择。
步骤:
- 使用所有特征先创建一个基准模型
- 剔除一个特征,创建新模型,记录新模型的预测精度
如果预测精度比基准模型高,剔除该特征
如果预测精度比基准模型低,保留该特征 - 重复第二步,直到预测精度保持稳定。
sklearn.feature_selection提供了两个接口:
- RFE: 可指定选择的特征数。
- RFECV: 根据k折交叉验证评分自动选择最优特征。
递归消除剔除了相关特征。
from sklearn.linear_model import LogisticRegression
from sklearn.feature_selection import RFECV
# 创建筛选器
selector = RFECV(
estimator=LogisticRegression(), # 由于这是分类问题,选择简单的逻辑回归
min_features_to_select=3, # 选择的最小特征数量
cv=5, # 交叉验证折数
scoring="accuracy", # 评估预测精度的指标
n_jobs=-1 # 使用所有CPU运算
)
# 拟合数据
results = selector.fit(X, y)
# 查看结果
# results.n_features_: 最终选择的特征数量
# results.support_: 布尔向量,True表示保留特征,False表示剔除特征
# results.ranking_: 特征等级,1表示最高优先级,即应该保留的特征
print("Number of selected features = %d" % results.n_features_)
print("Selected features: %s" % results.support_)
print("Feature ranking: %s" % results.ranking_)
Number of selected features = 5
Selected features: [ True True False False False True True True]
Feature ranking: [1 1 2 3 4 1 1 1]
--------------------------------------------------
调用results.transform()筛选最重要的特征。
X_new = results.transform(X)
X_new
array([[ 6. , 148. , 33.6 , 0.627, 50. ],
[ 1. , 85. , 26.6 , 0.351, 31. ],
[ 8. , 183. , 23.3 , 0.672, 32. ],
...,
[ 5. , 121. , 26.2 , 0.245, 30. ],
[ 1. , 126. , 30.1 , 0.349, 47. ],
[ 1. , 93. , 30.4 , 0.315, 23. ]])
----------------------------------
根据拟合结果自行筛选特征。
X_new = X.loc[:, results.support_]
X_new.head()
更多例子:
例子1:不采用交叉验证
# 属性:
# support_:选择的特征,布尔类型。
# ranking_:特征的排名位置。 选择的特征被指定为等级1。越差的特征,等级数越高。
# 不采用交叉验证
from sklearn.datasets import make_friedman1
from sklearn.feature_selection import RFE
from sklearn.svm import SVR
X, y = make_friedman1(n_samples=50, n_features=10, random_state=0)
# 可以选择不同的基模型
estimator = SVR(kernel="linear")
#参数estimator为基模型,参数n_features_to_select为选择的特征个数
selector = RFE(estimator, n_features_to_select=5)
selector = selector.fit(X, y)
print('selector.support_:\n',selector.support_)
print('selector.ranking_:\n',selector.ranking_)
print(selector)
例子2:采用交叉验证
# # 采用交叉验证,
from sklearn.datasets import make_friedman1
from sklearn.feature_selection import RFECV
from sklearn.svm import SVR
X, y = make_friedman1(n_samples=50, n_features=10, random_state=0)
estimator = SVR(kernel="linear")
# 参数:
# step: 如果大于或等于1,那么`step`对应于(整数)每次迭代时要删除的要素数。
# 如果在(0.0,1.0)内,那么`step`对应于百分比(向下舍入)要在每次迭代时删除的要素。
selector = RFECV(estimator, step=1, cv=6)
selector = selector.fit(X, y)
print('selector.support_:\n',selector.support_)
print('selector.ranking_:\n',selector.ranking_)
(4)特征重要性【待更新】
可以量化特征的重要程度,重要程度越高,对模型准确预测的贡献越大,反之贡献越低。要评估特征重要性,需要借助以树模型为基础的集成学习算法,最常用的是随机森林(RandomForest)。用随机森林拟合训练集后,可以从’feature_importances_'属性中获取特征重要性,用户可根据预先设定的阈值选择最重要的特征。
(5)降维 【待更新】
(6)其它【待更新】
5.参考
https://github.com/sladesha/Reflection_Summary
https://zhuanlan.zhihu.com/p/74198735
https://www.cnblogs.com/gczr/p/6802948.html
https://blog.csdn.net/qq_39923466/article/details/118809782
https://blog.csdn.net/sinat_23971513/article/details/103797927
https://www.cnblogs.com/nxf-rabbit75/p/11122415.html
https://zhuanlan.zhihu.com/p/91031244
https://www.jianshu.com/p/e858a6fffe0b
方差分析
sklearn实现特征选择
KL离散度
KL离散度2
距离相关系数

