R语言中ggplot2绘制热图,美化热图流程
ggplot2是一款强大的绘图R包,今天的笔记内容是学习使用ggplot2来绘制一幅热图,并进行美化调整。需要加载的包有两个:ggplot2、reshape2。
如果你也想跟着笔记一起学习,请访问链接下载脚本:
https://down.jewin.love/?f=/Rscript/
访问上面的网址即可下载使用,也可以直接在Rstudio中运行下面这一句代码,则会自动在当前工作路径下生成11副PDF图片结果,稍加修改输入数据就能生成其他的热图。
source("https://down.jewin.love/?f=/Rscript/heatmap.R")
接下来利用示例数据进行简单的流程介绍:
如何绘制一幅热图?
前期准备与数据创建
首先,需要安装并加载这两个R包。
install.packages("ggplot2")
install.packages("reshape2")
library(ggplot2)
library(reshape2)
创建示例数据,并输出数据查看是否正常。
> data <- c(1:6,6:1,6:1,1:6, (6:1)/10,(1:6)/10,(1:6)/10,(6:1)/10,1:6,6:1,6:1,1:6,6:1,1:6,1:6,6:1)
> data #查看当前数据
[1] 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0
[23] 5.0 6.0 0.6 0.5 0.4 0.3 0.2 0.1 0.1 0.2 0.3 0.4 0.5 0.6 0.1 0.2 0.3 0.4 0.5 0.6 0.6 0.5
[45] 0.4 0.3 0.2 0.1 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0 6.0 5.0 4.0 3.0 2.0 1.0
[67] 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0 1.0 2.0 3.0 4.0
[89] 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0
将输入的data数据转化成数据框格式,并更改列名和行名,查看前五行数据观察是否正常。
> data <- as.data.frame(matrix(data,ncol = 12,byrow = T)) #转化成数据框
> colnames(data) <- c("Zygote","2_cell","4_cell","8_cell","Morula","ICM","ESC","4 week PGC","7 week PGC","10 week PGC","17 week PGC", "OOcyte")
> rownames(data) <- paste("Gene",1:8,sep ="_")
> head(data,5) #输出前五行
Zygote 2_cell 4_cell 8_cell Morula ICM ESC 4 week PGC 7 week PGC 10 week PGC
Gene_1 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0
Gene_2 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0
Gene_3 0.6 0.5 0.4 0.3 0.2 0.1 0.1 0.2 0.3 0.4
Gene_4 0.1 0.2 0.3 0.4 0.5 0.6 0.6 0.5 0.4 0.3
Gene_5 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0
新建一列命名为id,内容为每行数据的行名,这样做的目的是为了后续将数据用melt函数转化为长类型(每一行的value数值变量只有一项,作图所需数据格式),查看数据前5行进行观察。
> data$id <- rownames(data) #新建一列(ID),内容为数据的行名
> data_m <- melt(data,id.vars=c("id")) #将原来的数据转化成长数据格式,并以ID列为主因素
> head(data_m,5) #查看转换后的结果,该数据将用于后续的流程
id variable value
1 Gene_1 Zygote 1.0
2 Gene_2 Zygote 6.0
3 Gene_3 Zygote 0.6
4 Gene_4 Zygote 0.1
5 Gene_5 Zygote 1.0
开始绘图
利用ggplot函数绘制热图,步骤:先初始化坐标轴(x= ,y= )然后设置填充类型和填充数据,在绘图函数后用+连接主题修改函数,可以在不断地修饰美化图片。
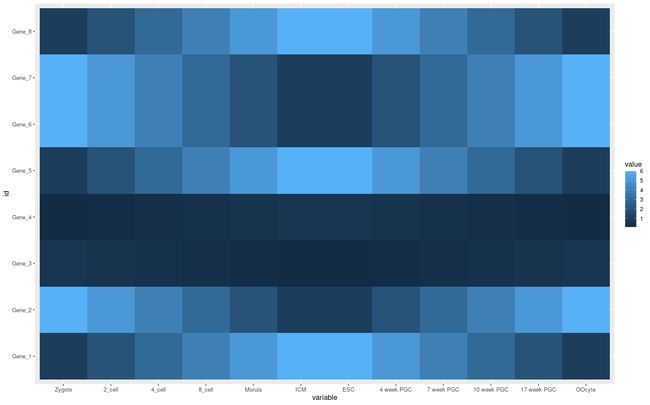
p <- ggplot(data_m,aes(x=variable,y=id)) #初始化,读入数据,设置x轴和y轴
p <- p + geom_tile(aes(fill=value))
#设置填充项为value值,绘制热图
p #查看生成的结果plot,在屏幕右下角处plot显示
保存图片
利用ggsave函数来保存生成的图片结果,这里演示将p保存到当前目录下,命名为my plot 1 .pdf,之后可以在相应目录下找到这张照片,无损矢量图方便修改。
ggsave(p,filename = "my plot 1.pdf",width = 10,
height = 15,units = "cm",colormodel="srgb")
#保存当前文件,生成的文件在当前工作目录下保存
刚刚那张图横轴的标签有的重叠到一起了,这样不好。那么如何对生成的热图进行后续调整呢?
美化热图
tips:切换显示主题与颜色
theme函数用于设置主题
axis.text.x表示x轴标签文本
element_text用来更改文本的角度(angle)、位置(hjust、vjust)
scale_fill_gradient表示用双色梯度方式填充显示。
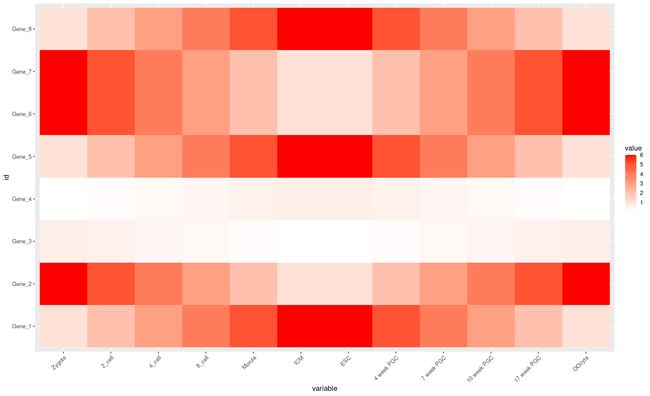
p <- p + theme(axis.text.x = element_text(
angle = 45,hjust = 1,vjust = 1)) #设置标签的显示方式
p <- p + scale_fill_gradient(
low="white",high = "red") #设置填充颜色
p #查看结果
切换显示类型与数据展示方式
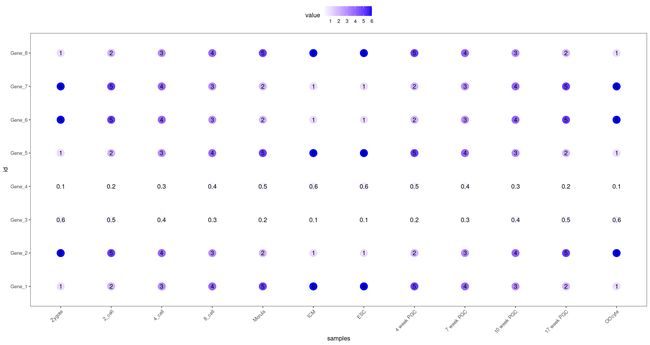
比如,我想在热图中同时展示两种颜色填充和数值,需要在上面的代码基础上略加修改,添加新的绘图参数。
p <- ggplot(data_m, #输入文件
aes(x=variable,y=id)) + #初始化坐标轴
xlab("samples") + #x轴标签
theme_bw() + #设置系统自带主题
theme(panel.grid.major = element_blank()) + #设置主项网格
theme(legend.key=element_blank()) + #去掉背景颜色
theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1)) + #设置坐标轴标签
theme(legend.position="top") + #设置图例的位置
geom_tile(aes(fill=value)) + #设置填充的值
scale_fill_gradient(low = "white", high = "red") + #设置颜色梯度
geom_point(aes(color=value), size=6) + #设置点的颜色和大小
geom_text(aes(label=value)) #设置文本显示数值
p #输出图片
试一试:重新更换一种主题样式,将红色填充去掉,并改为蓝色圆点加数值显示。
p <- ggplot(data_m,aes(x=variable,y=id)) +
xlab("samples") + theme_bw() +
theme(panel.grid.major = element_blank()) +
theme(legend.key = element_blank()) +
theme(axis.text.x = element_text(angle = 45,hjust = 1,vjust = 1)) +
theme(legend.position="top")+
geom_point(aes(color= value),size=6) +
scale_color_gradient(low = "white",high = "blue") +
geom_text(aes(label=value))
p
本节笔记到这里结束,感谢你的浏览,如果感觉有用欢迎转发分享,明天继续分享绘制热图过程中遇到数据的差异比较大时处理方法,对原始数据进行转化的技巧和应用。
参考引用:http://www.ehbio.com/Bioinfo_R_course/Rplots.html#ggplot2_heatmapmeihua
本文由mdnice多平台发布