R -- 二分类问题的分类+预测
brief
分类大致分为有监督分类和无监督分类,这里学习有监督分类。有监督分类一般包括逻辑回归、决策树、随机森林、支持向量机、神经网络等。
有监督学习基于一组包含预测变量值和输出变量值的样本单元。然后可以将全部数据分为一个训练数据集和一个验证数据集,其中训练数据集用于建立预测模型,验证数据集用于测试模型的准确性。
这里将通过 rpart、rpart.plot和party包实现决策树模型和可视化
通过randomForest包拟合随机森林
通过e1071包构造支持向量机
通过glm函数实现逻辑回归。
pkgs <- c("rpart", "rpart.plot", "party","randomForest", "e1071")
install.packages(pkgs, depend=TRUE)
数据准备
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url <- paste(loc, ds, sep="")
breast <- read.table(url, sep=",", header=FALSE, na.strings="?")
names(breast) <- c("ID", "clumpThickness", "sizeUniformity",
"shapeUniformity", "maginalAdhesion",
"singleEpithelialCellSize", "bareNuclei",
"blandChromatin", "normalNucleoli", "mitosis", "class")
df <- breast[-1]
df$class <- factor(df$class, levels=c(2,4),
labels=c("benign", "malignant"))
set.seed(1234)
train <- sample(nrow(df), 0.7*nrow(df))
df.train <- df[train,]
df.validate <- df[-train,]
table(df.train$class)
table(df.validate$class)
逻辑回归
逻辑回归(logistic regression)是广义线性模型的一种,可根据一组数值变量预测二元输出。R中的基本函数glm()函数可用于拟合逻辑回归模型。glm()函数自动将预测变量中的分类变量编码为相应的虚拟变量。本次的数据集预测变量都是数值向量,因此不必进行编码。代码如下:
fit.logit <- glm(class~., data=df.train, family=binomial())
summary(fit.logit)
-
预测变量的系数显著性检验表示有些变量系数很显著,有些不显著,这就是说有些预测变量对模型的解释度不够,可以舍弃。这里我们就不进行预测变量的优化了。
-
要注意的是logistic回归的结果变量的值是Y=1优势比,可以转换成概率 。
-
下面我们用上述的回归模型进行预测,此时的结果是对应的概率
# predict 默认输出对数概率,设置type = “response”返回概率值
prob <- predict(fit.logit, df.validate, type="response")# 进行预测
logit.pred <- factor(prob > .5, levels=c(FALSE, TRUE),
labels=c("benign", "malignant")) # 进行预测
logit.perf <- table(df.validate$class, logit.pred,
dnn=c("Actual", "Predicted")) #评估预测准确性
logit.perf
决策树
决策树试数据挖掘领域中常用的模型,其基本思想是对预测变量进行二元分离,从而构建一颗可用于预测新样本单元所属类别的树。
经典树
library(rpart)
set.seed(1234)
dtree <- rpart(class ~ ., data=df.train, method="class",
parms=list(split="information"))
print(dtree)
summary(dtree)
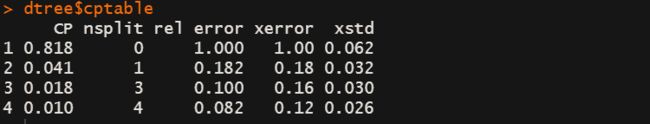
# 复杂度参数 cp 用于惩罚过大的树
# 树的大小也就是分支nsplit,有n个分支的树会有n+1个node
# rel error 栏即训练集中各种树对应的误差
# 交叉验证错误 xerror 即基于训练样本所得的10折交叉验证误差
# xstd 栏为交叉验证误差的标准差
dtree$cptable
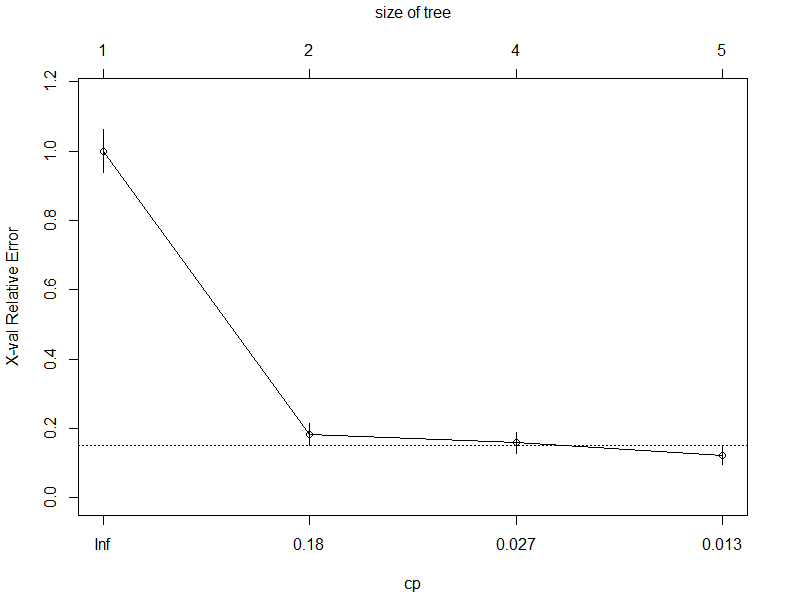
plotcp(dtree)
- dtree的总结

- 交叉验证的误差以及可视化
交叉验证误差 小于 最小的交叉验证误差一个标准误差范围内的树,就是最小的最优的树。
最小的xerror + 对应的xstd = 水平虚线,node=5时xerror 低于虚线,则nsplit=4最优。
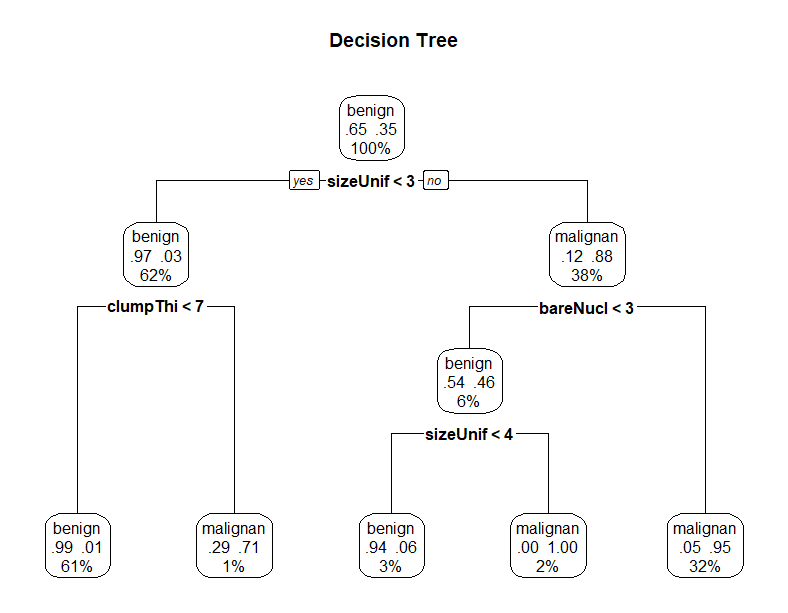
- 最终修剪和可视化
# 在完整树的的基础上,prune根据复杂度参数剪掉最不重要的枝,从而将树控制在理想范围内
dtree.pruned <- prune(dtree, cp=.01) # 剪枝
# rpart.plot包中的prp函数可以画出最终的决策树
library(rpart.plot)
prp(dtree.pruned, type = 2, extra = 104,
fallen.leaves = TRUE, main="Decision Tree")
- 进行预测
# 进行预测
dtree.pred <- predict(dtree.pruned, df.validate, type="class")
# 混淆矩阵
dtree.perf <- table(df.validate$class, dtree.pred,
dnn=c("Actual", "Predicted"))
dtree.perf
条件推断树


预测变量与响应变量的回归关系是否显著以及显著性大小为分割变量
library(party)
fit.ctree <- ctree(class~., data=df.train) # 推断决策
plot(fit.ctree, main="Conditional Inference Tree") #结果可视化
#预测
ctree.pred <- predict(fit.ctree, df.validate, type="response")
#混淆矩阵
ctree.perf <- table(df.validate$class, ctree.pred,
dnn=c("Actual", "Predicted"))
ctree.perf
随机森林
library(randomForest)
set.seed(1234)
fit.forest <- randomForest(class~., data=df.train,
na.action=na.roughfix,
importance=TRUE)
fit.forest
importance(fit.forest, type=2)
# predict
forest.pred <- predict(fit.forest, df.validate)
forest.perf <- table(df.validate$class, forest.pred,
dnn=c("Actual", "Predicted"))
forest.perf
支持向量机
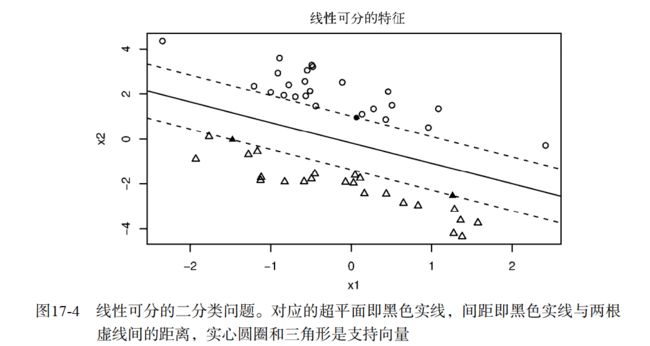
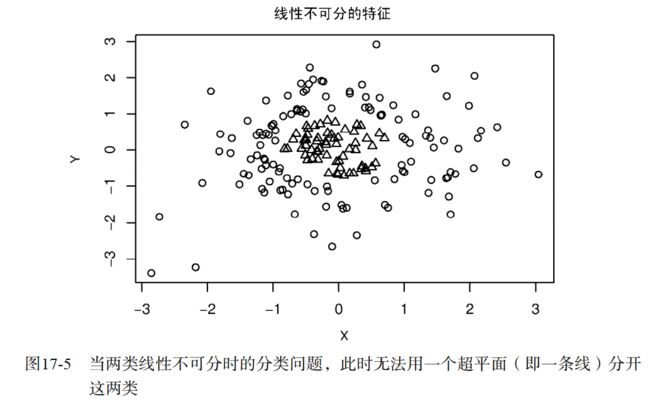
关于SVM应用两类线性不可分的分类问题时如何解决,我觉得这个的视频做的蛮通俗易懂的,链接。
kernlab包中的ksvm()和e1071包中的svm()都可以实现支持向量机算法,kernlab包中的ksvm()功能更强大,意味着参数更多学习成本更高,所以用e1071包中的svm()演示。
- 由于方差较大的预测变量对SVM的生成影响更大,svm()函数默认在生成模型前对每个变量标准化,使其均值为0,标准差为1.与随机森林算法不同的是,SVM在预测新样本单元时不允许有缺失值出现。
library(e1071)
set.seed(1234)
fit.svm <- svm(class~., data=df.train)
fit.svm
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf
# 利用tune.svm()
set.seed(1234)
tuned <- tune.svm(class~., data=df.train,
gamma=10^(-6:1),
cost=10^(-10:10))
tuned #根据返回的结果设置gamma值和cost值
fit.svm <- svm(class~., data=df.train, gamma=.01, cost=1)
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf