利用gatk进行calling-snp
calling-snp流程整理
1.对每个样品进行SNP Calling得到gvcf文件
## filter fastq file by fastp
使用fastp对原始数据进行过滤
$ fastp -i ./data/$1_1.fq.gz -I ./data/$1_2.fq.gz -o $1_1_clean.fa.gz -O $1_2_clean.fa.gz -q 5 -u 50 -n 10
参数 -q 5 -u 50 :read中含有的低质量(Q <= 5)碱基数超过该条read长度比例的 50% 时去除此read;-n :read中含有的N碱基的含量超过该条read长度比例的10% 时去除此reads
nohup fastp -i ./SRR12396010_1.fastq -o SRR12396010_1.clean.fastq -I SRR12396010_2.fastq -O SRR12396010_2.clean.fastq -q 5 -u 50 -n 10 &
-i, --in1 read1 input file name (string)
-o, --out1 read1 output file name (string [=])
-I, --in2 read2 input file name (string [=])
-O, --out2 read2 output file name (string [=])
问题:fastp过滤时,自己下载的数据是从ncbi下载的SRR项目的二进制文件
- 先将SRR1239601.1,命令mv SRR1239601.1 SRR1239601.sra
- 利用命令fastq-dump –split-3 ${i}.sra ,将.sra文件转换成为fastq格式
- 转换 .sra 文件成 .fastq/fasta 文件
- #single-end 单端测序
- .../fastq-dump DRR000003.sra # 结果生成DRR000003.fastq
- .../fastq-dump --fasta DRR000003.sra # 结果生成DRR000003.fastq
- #pair-end 双端测序
- .../fastq-dump --split-3 DRR002018.sra # 结果生成 DRR002018_1.fastq,DRR002018_2.fastq
## map reads by BWA-MEM
使用bwa mem将read比对到参考基因组
先建立索引:
$ bwa index ref.genome.fasta
$ bwa mem -R "@RG\tID:group_1\tLB:library_1\tPL:illumina\tSM:sample_1" /home/fengliying/data/oryza_AA/msu7.fasta /home/fengliying/data/oryza_AA/00.fastp/$1_1_clean.fa.gz /home/fengliying/data/oryza_AA/00.fastp/$1_2_clean.fa.gz | samtools view -@ 36 -S -b - -o $1.bam
自己的命令模式:
bwa mem -R "@RG\tID:group_1\tLB:library_1\tPL:illumina\tSM:sample_1" /home/huangguizhong/perl5/data_get/ref.japonica.genome.fasta /home/huangguizhong/perl5/data_get/SRR1239602_1.clean.fastq /home/huangguizhong/perl5/data_get/SRR1239602_2.clean.fastq > SRR1239602.sam
注意:上面的命令一次性跑,会报错[main_samview] fail to read the header from "-"。第一次是把bwa mem写错了“bwa men”。但是,命令改正后一次性跑还是报同样的错误。后面。将命令bwa mem和samtools view命令分开跑。另外,bwa mem这一步需要指定将结果输出到SRR1239601.sam
接着:#SAM转换为BAM
$ samtools view -bS out.sam >out.bam
出现了问题?
$ samtools view -@ 36 -S -b SRR1239601.sam > SRR1239601.pe3.bam
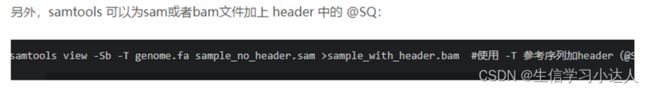
$ samtools view -Sb -T ref.japonica.genome.fasta SRR1239601.sam > SRR1239601_with_header.bam
-b 输出SAM格式
-o 输出文件,后接输出文件名
-@ INT 除了主线程[0]之外要使用的BAM压缩线程数。
-S 忽略了与以前的samtools版本的兼容性。以前,如果输入是SAM格式,则需要此选项,但现在通过检查输入的前几个字符自动检测到正确的格式。
参数 :默认参数
## sort BAM file
使用samtools将比对得到的bam文件进行排序
$ samtools sort -@ 64 -m 1G -O bam -o $1_sort.bam $1.bam
$samtools sort -@ 64 -m 1G -O bam -o /home/huangguizhong/perl5/data_get /SRR1239601.pe1_sort.bam /home/huangguizhong/perl5/data_get /SRR1239601.pe1.bam
参数 -@ 64 -m 1G :使用64个线程,每个线程可用1G内存
## mark PCR duplicates
对排序后的bam文件进行标识PCR重复,去除由PCR扩增所形成的duplicates
$ samtools rmdup $1_sort.bam $1_sort_rmdup.bam
参数:默认参数
## build BAM index
对去重后的bam文件构建索引
$ samtools index -@ 36 $1_sort_rmdup.bam
参数:默认参数
## generate GCVF by HaplotypeCaller
使用4.0版本GATK的HaplotypeCaller模块进行SNP calling,得到包含所有位点SNP信息的gvcf文件
$/home/fengliying/app/GATK/gatk-4.0.4.0/gatk HaplotypeCaller -R /home/fengliying/data/oryza_AA/msu7.fasta -I /home/fengliying/data/oryza_AA/01.bwa/$1_sort_rmdup.bam --genotyping-mode DISCOVERY -stand-call-conf 30 --emit-ref-confidence GVCF -O $1.g.vcf.gz
参数:主要是要 --emit-ref-confidence GVCF 这个参数,确保得到每个样品的gvcf 文件
- 将每个样品的gvcf进行合并和genotype,得到群体populations vcf文件
##joint calling
每个样品都进行snp calling得到gvcf文件后,将所有样品的gvcf文件进行合并得到总的群体gvcf文件
$gatk CombineGVCFs --reference /home/fengliying/data/oryza_AA/msu7.fasta --variant $1.g.vcf.gz --variant s2.g.vcf.gz --variant s3.g.vcf.gz (有多少个样品写多少个) --output ${pop}.g.vcf.gz
参数:默认参数
##genotype
对得到的群体gvcf文件进行genotype,得到最终的原始群体vcf文件
$gatk GenotypeGVCFs --reference ${reference} --variant ${pop}.g.vcf.gz --output ${pop}.raw.vcf.gz
参数:默认参数
- 对原始群体vcf文件进行进一步的过滤获得更准确的SNP信息
## select SNPs from raw vcf
因为得到的原始vcf文件中同时包含SNP和InDel位点,所以先把SNP位点提取出来
$gatk SelectVariants --select-type-to-include SNP --reference ${reference} --variant ${pop}.raw.vcf.gz --output ${pop}.raw.snp.vcf.gz
参数:默认参数
## perform hard filtering
然后对只包含SNP位点的vcf文件进行硬过滤
$ gatk VariantFiltration --variant ${pop}.raw.snp.vcf.gz --filter-expression "QD < 2.0 || FS > 60.0 || MQ < 40.0 || SOR > 3.0 || MQRankSum < -12.5 || ReadPosRankSum < -8.0" --filter-name "snp_filter" --genotype-filter-expression "DP < 2 || DP > 50" --genotype-filter-name "dp_fail" --output ${pop}.flag.snp.vcf.gz
参数:过滤参数为GATK官方推荐的参数
## selecting bi-allelic SNPs that pass the filtering
把符合过滤条件的SNP位点再提取出来,得到通过硬过滤的包含较高质量SNP位点的vcf文件
$gatk SelectVariants --exclude-filtered true --restrict-alleles-to BIALLELIC --reference ${reference} --variant ${pop}.flag.snp.vcf.gz --output ${pop}.hardfiltered.snp.vcf.gz
参数:默认参数
## perform population filtering
在进行后续的群体分析时,最好还需要在缺失位点和次等位基因频率上对SNP位点进行进一步过滤,这一步可以用vcftools
$ vcftools --gzvcf ${pop}.hardfiltered.snp.vcf.gz --max-missing 0.9 --maf 0.05 --recode --recode-INFO-all --stdout | bgzip -c > ${pop}.maffiltered.snp.vcf.gz
参数:--max-missing 0.9 --maf 0.05 保留位点缺失率小于10% (确保90%的样品包含该SNP)和次等位基因频率大于0.05的SNP位点
如果还需要InDel位点,可以按上面提取SNP的方法进行InDel位点的提取和过滤(过滤的参数跟SNP稍微不一样)
## select indels from raw vcf
gatk SelectVariants --select-type-to-include INDEL --reference ${reference} --variant ${pop}.raw.vcf.gz --output ${pop}.raw.indel.vcf.gz
## perform hard filtering
gatk VariantFiltration --variant ${pop}.raw.indel.vcf.gz --filter-expression "QD < 2.0 || FS > 200.0 || SOR > 10.0 || MQRankSum < -12.5 || ReadPosRankSum < -8.0" --filter-name "indel_filter" --output ${pop}.flag.indel.vcf.gz
## selecting indels that pass the filtering
gatk SelectVariants --exclude-filtered true --reference ${reference} --variant ${pop}.flag.indel.vcf.gz --output ${pop}.hardfiltered.indel.vcf.gz
好啦,今天就分享到这啦!