AI 对抗超级细菌:麦克马斯特大学利用深度学习发现新型抗生素 abaucin
内容一览:鲍曼不动杆菌是一种常见的医院获得性革兰氏阴性病原体,通常表现出多重耐药性。利用传统方法,发现抑制此菌的新型抗生素很困难。但利用机器学习可以快速探索化学空间,从而增加发现新型抗菌分子的可能性。近期,国际期刊《Nature Chemical Biology》上发布了一篇研究成果,研究人员发现 abaucin 能够有效地抑制鲍曼不动杆菌。
关键词:鲍曼不动杆菌 abaucin 深度学习
本文首发自 HyperAI 超神经微信公众平台~
鲍曼不动杆菌(学名:Acinetobacter baumannii,俗称:AB 菌)为不动杆菌属中最常见的一种革兰阴性杆菌,广泛存在于自然界的水及土壤中,也存在于正常人体皮肤、呼吸道、消化道和泌尿生殖道中。该菌常见于医院感染,也是水产养殖业动物的病原菌,通常会引起菌血症、肺炎、脑膜炎、腹膜炎、心内膜炎以及泌尿道和皮肤感染。
鲍曼不动杆菌对湿热、紫外线、化学消毒剂有较强的抵抗力,耐低温。在干燥的物体表面可以存活 25 天以上,常规消毒剂只能抑制其生长,不能杀灭,而且耐受肥皂,是医务人员手上、医疗器械、物体表面最常分离到的革兰阴性杆菌。由于抗生素的滥用,鲍曼不动杆菌产生多重抗药性,被世界卫生组织认定为世界上最危险的耐抗生素细菌之一。
近期,来自麦克马斯特大学 (McMaster University) 的 Gary Liu 以及来自麻省理工学院 (Massachusetts Institute of Technology) 的 Denise B. Catacutan 等研究人员利用深度学习筛选了大约 7,500 个分子,找出了抑制鲍曼不动杆菌的新型抗生素。目前,该研究已发布在《Nature Chemical Biology》期刊上,标题为「Deep learning-guided discovery of an antibiotic targeting Acinetobacter baumannii」。
该研究成果已发表在《Nature Chemical Biology》期刊上
论文地址:
Deep learning-guided discovery of an antibiotic targeting Acinetobacter baumannii | Nature Chemical Biology
实验过程
数据集
研究人员基于鲍曼不动杆菌 ATCC 17978 在 LB 培养基中的生长,对 7,684 个小分子进行了筛选。这些小分子包括 2,341 个非专利药物和 5,343 个合成化学物,都是由 Broad 研究所的高通量筛选子库提供的。根据传统的命中率分界线(即与整个数据集的平均生长抑制率相比低于一个标准差的分子),他们确定了 480 个分子为「活性」,而 7,204 个分子为「非活性」。
模型训练
为了增强模型结构,研究人员将分子表征与使用 RDCit 计算的 200 个额外的分子级特征连接起来。此外,为了进一步提高模型的性能,研究人员使用了一个由 10 个模型组成的集合,对 7,684 个分子的训练数据集(有 ~6.2% 的有效例子)进行模型建立和训练,并将模型应用于更新的药物再利用中心 (Drug Repurposing Hub)。研究人员把数据集随机分成 80% 的训练数据、10% 的验证数据和 10% 的测试数据。
图 1:模型训练数据
a. 7,684 个小分子的初步筛选数据,这些小分子在 50 μM 的 LB 培养基中抑制鲍曼不动杆菌 ATCC 17978 的生长。
b. 从预测集中选择用于验证的 240 个优先分子的排序生长抑制数据(上);预测得分最低的 240 个预测分子的排序生长抑制数据(中);训练数据集中未找到的具有最高预测分数的 240 个预测分子的排序生长抑制数据(底部)。水平虚线代表 50 μM 时 >80% 生长抑制的严格命中截止值。
c. 鲍曼不动杆菌在 LB 培养基中被 abaucin(蓝色)和 serdemetan(红色)抑制生长。
d. 用不同浓度的 abaucin 处理 6 小时后鲍曼不动杆菌细胞的生长动力。
代码获取:
GitHub - chemprop/chemprop: Message Passing Neural Networks for Molecule Property Prediction
小鼠模型实验
研究人员对 6 至 8 周大的雌性 C57BL/6N 小鼠进行了预处理,在实验开始前四天和前一天分别按体重注射了 150 mg/kg 及 100 mg/kg 的环磷酰胺,使小鼠中性粒细胞减少。在第 0 天,用异氟醚对小鼠进行麻醉,并以 0.1 mg/kg 的剂量对小鼠进行腹腔注射丁丙诺啡作为止痛剂。研究人员用大约 30-35 片高压胶布在小鼠背上留下 2 平方厘米的擦伤,通过胶布剥离到表皮基底层,随即使用 ~6.5 x 106 CFU A. baumannii ATCC 17978 感染小鼠。小鼠 (n = 5/6) 在感染后 1、2、3、4、6、10、21 和 24 小时用 ~10-20 μl 带有 abaucin 的 Glaxal Base (实验组) 或 DMSO (对照组) 治疗。
实验结果
研究人员在机器学习的帮助下发现了一种名为 abaucin 的抗菌化合物,它具有针对鲍曼不动杆菌的窄谱活性,能够通过干扰 LolE 的机制来干扰类脂蛋白的转运过程。下图显示了深度学习引导发现 abaucin 的过程。
图 2:机器学习引导发现 abaucin
a. 筛选的 ~7,500 个分子(蓝色);用该生长抑制数据集训练的定向信息传递的深度神经网络,并在药物再利用中心进行预测(红色);能够抑制鲍曼不动杆菌的新分子(紫色)。
b. 7,684 个小分子集合在 50 μM 时对鲍曼不动杆菌 ATCC 17978 的生长抑制。
c. 训练后的模型对药物再利用中心内的分子进行的预测分数排序。预测分数大于 0.2 的分子为初步候选分子。
d. A t-SNE 图显示了训练数据集(蓝色)、预测集(红色)和 abaucin(黄色)之间的化学关系。
e. abaucin 在 LB 培养基中对鲍曼不动杆菌的生长抑制。
f. abaucin 在营养丰富的条件下以不同浓度培养 1.5 小时(蓝色)、3 小时(蓝绿色)、4.5 小时(绿色)和 6 小时(紫色)后对鲍曼不动杆菌的杀伤力。
g. abaucin 在营养不足的条件下培养 1.5 小时(蓝色)、3 小时(蓝绿色)、4.5 小时(绿色)和 6 小时(紫色)后对鲍曼不动杆菌的杀伤力。
如图 2 所示,研究人员筛选了大约 7,500 个分子,利用这个生长抑制数据集训练了一个神经网络,并对能够抑制鲍曼不动杆菌活性的新型分子进行了预测。总的来说,这些数据表明,abaucin 通过抑制一种在生长和分裂期间活性最大的生物过程而发挥其抗菌效力,与大多数已知的抗生素一致。
此外,为了测试 abaucin 的体内功效,研究人员在小鼠的背面建立了伤口感染。
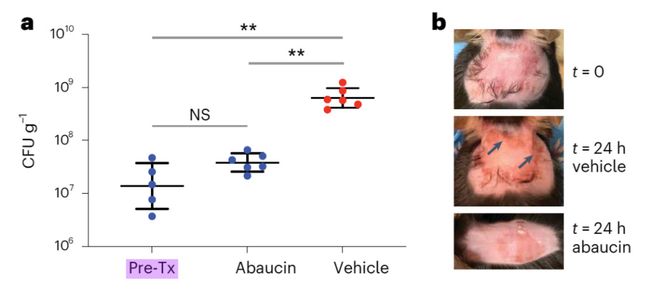
图 3:abaucin 可以抑制伤口模型中的鲍曼不动杆菌感染
a. 在背部伤口感染模型中,小鼠感染鲍曼不动杆菌 ATCC 17978 (~6.5 x 106 CFU)。感染 1 小时后,用 DMSO(红色;n = 6)或 4% abaucin(蓝色;n = 6)治疗小鼠 24 小时以上。纵轴是感染后 25 小时伤口组织的细菌负荷。Pre-Tx 代表初始治疗时的细菌负荷 (n = 5)。黑线代表平均值 ±s.d。NS 表示无统计学意义;** P < 0.005 使用未配对的两侧 t 检验和 Welch 校正(Pre-Tx 与 4% abaucin 相比,P= 0.0704;Pre-Tx 与 DMSO 相比,P= 0.0034;DMSO 与 4% abaucin 相比,P= 0.0039)。
p.s. 一般一个星号代表 p 值小于 0.05,两个星号代表 p 值小于 0.01,三颗星代表 p 值小于 0.001。p 值越小,显著性越强。
b. 感染前 (t = 0)、用 DMSO 处理 24 小时后以及用 abaucin 处理 24 小时后的小鼠背表面图像。在 DMSO 中观察到的炎症 (箭头) 在用 abaucin 处理的小鼠中不存在。
图 3 显示,使用 abaucin 进行治疗后的伤口细菌负荷明显低于 DMSO 组,且在用 DMSO 处理 24 小时后仍存在的伤口炎症在 abaucin 中消失了,abaucin 能够有效地控制鲍曼不动杆菌的感染。
综上,研究人员利用深度学习发现 abaucin 能够有效控制鲍曼不动杆菌感染,这项研究强调了机器学习在发现新抗生素方面的作用,并引导了具有挑战性的革兰阴性病原体研究。
AI 改变传统新药发现方式
值得注意的是,本研究为麦克马斯特大学助理教授 Jon Stokes 与 MIT 医学工程和科学教授 James J. Collins、麦克马斯特大学研究生 Gary Liu 及 Denise Catacutan 等人一起开展的。Jon Stokes、Gary Liu 与 Denise Catacutan 皆为 Stokes Laboratory 的成员。
Stokes Laboratory 地址:
Projects — We are the Stokes Lab
该实验室通过利用最先进的高通量生物学和当代人工智能方法来发现新型抗生素。具体而言,研究人员通过训练深度学习模型从庞大的计算机模拟数据库中预测新型抗生素,还建立模型来预测新抗生素的作用机制和体内特性。他们利用机器学习来加速新型抗生素的发现进程,同时降低研发成本。
Stokes 教授曾在一次采访中说到「人工智能将从根本上改变新药发现方式」。研究结果表明,人工智能对发现针对多种具有挑战性病原体的新型抗生素具有重大意义,这种方法在寻找其他抗菌疗法方面也具有潜力。人工智能虽然不能解决所有问题,但作为一种强大的工具,它可以帮助我们寻找新药,为人们带来希望。
参考文章:
[1]http://ccm.dxy.cn/article/805579
[2]https://zh.wikipedia.org/wiki/%E9%AE%91%E6%B0%8F%E4%B8%8D%E5%8B%95%E6%A1%BF%E8%8F%8C
[3]https://www.ctvnews.ca/health/scientists-use-ai-to-discover-antibiotic-for-very-difficult-to-treat-bacteria-1.6411927
本文首发自 HyperAI 超神经微信公众平台~