临床数据 5. 如何构建微卫星不稳定性结直肠癌预后评分系统?
临床数据分析方案
桓峰基因公众号推出临床数据分析方案教程,整理如下:
临床数据 1. 临床基因突变数据如何发高分?
临床数据 2. 基于NGS的胃癌诊疗全过程的临床应用方案
临床数据 3. 肿瘤微小残留病灶(MRD)如何发文章?
临床数据 4. 肿瘤克隆进化分析结果解读?
克隆进化生信分析教程
我们在桓峰基因公众号也推出克隆进化分析教程,从组织到单细胞的克隆进化分析都有介绍,供大家参考:
Clone 1. 肿瘤克隆进化之前世今生
Clone 2. 肿瘤克隆进化之不同进化模
Topic 1.克隆进化之sciClone
Topic 2.克隆进化之ClonEvol
Topic 3. 克隆进化之 fishplot
Topic 4. 克隆进化之 Pyclone
Topic 5. 克隆进化之 CITUP
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
Topic 9. 克隆进化之 TimeScape
如何构建微卫星不稳定性结直肠癌预后评分系统?
这个还是蛮有意思的,比如我们选取了一些微卫星不稳定的结直肠癌患者(>50)例癌和癌旁配对样本,进行一系列的分析,之后得到的基因标记再通过数据库TCGA里面的数据集进行验证,最后按照临床预测模型的方法,找到预后独立因子,构建微卫星不稳定性结直肠癌预后评分系统。
那么我们通过下面完整的课题设计思路看一个数据的分析策略,其实有很多分析我们都可以去尝试,幸运的话会出现意想不到的结果!!!
中国结直肠癌概况-新增死亡病例28万,死亡率高达30%
2020年WTO最新数据显示:全球结直肠癌新增死亡人数达93万,成为全球第二大死亡率高的癌症,而中国结直肠癌新增死亡人数高达28万,成为全球的死亡率最高的国家。
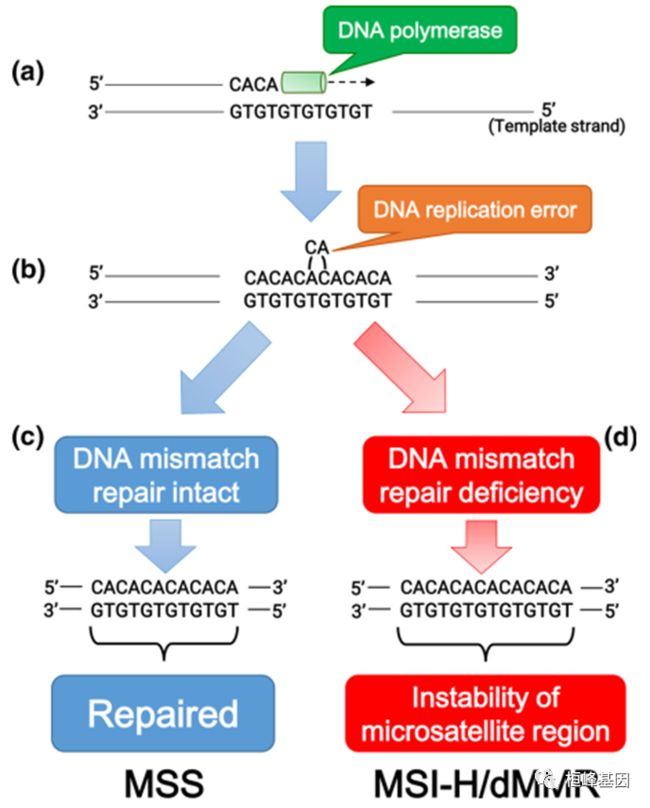
1.微卫星:贯穿人类基因组的1-6个短串联重复序列核苷酸
2.DNA错配修复(DNA mismatch repair, MMR):10个蛋白(MSH2、MSH6或MSH3,MLH1、PMS2、PMS1,MLH3及其形成的复合体
3.MMR缺陷(Deficient MMR,dMMR) :种系和/或体细胞突变发生在10个基因
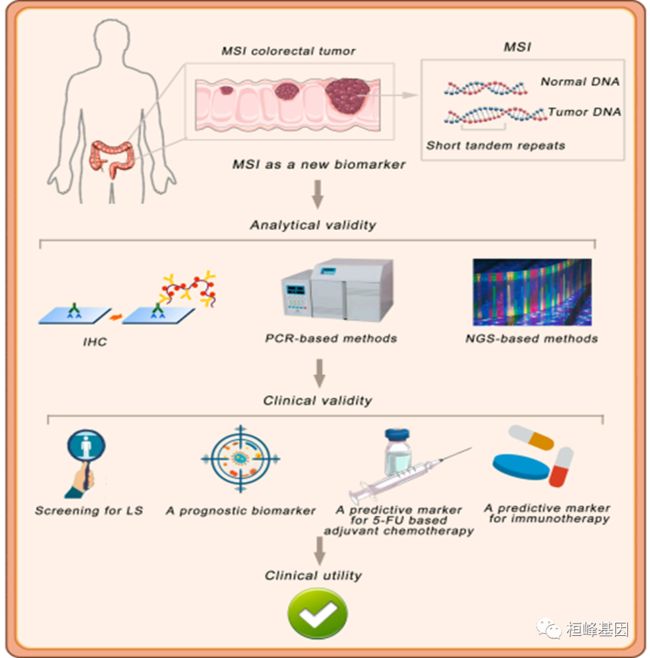
微卫星不稳定在结直肠癌中的应用-临床三大检测方法四大方向
三大检测方法
1.IHC:MMR蛋白表达包括10个蛋白基因
2.PCR:200左右微卫星位点
3.NGS:个性化定制微卫星检测位点
临床四大方向
1.筛选工具:MSI分析作为鉴别LS的筛选工具
2.预后因素:MSI作为结直肠癌预后生物标志物
3.预测指标:MSI作为结直肠癌5-氟尿嘧啶辅助化疗预测指标
4.预测标记:MSI作为结肠直肠癌免疫治疗预测标记物
MSIsensor检测技术全球领先
MSIsensor 是通过 MS 位点两端各 5bp 的侧翼序列来定位的,原理为
1.对于在肿瘤和正常样本中测序深度都 ≥20 的微卫星位点, 统计其每种重复长度的 reads 数目分布情况
2.使用卡方检验对微卫星位点上的分布进行统计检验, 若存在显著差异,则认为该位点是不稳定的
3.最后统计不稳定位点的比例, 如果该比例超过指定的阈值, 则认为该样本为 MSI-H
n应用于超过27个国家的 670个基因检测企业产品、药企的研发和科研团队的项目中
n被实际应用于超过233,000例癌症病人的MSI检测
n被顶级癌症研究机构及美国FDA推荐为NGS-MSI检测标准,并被用于FDA批准的首个基于NGS的基因检测大Panel(MSKCC-IMPACT)
MSIsensorII: MSIsensor 的新进化,更准、更快、更通用
MSIsensor-ct:MSI血检速达
MSIsensor |
n2014年发表在Bioinformatics n肿瘤组织+正常组织对照 n纪念斯隆凯特琳癌症研究中心于2017年获得FDA批准的MSK-IMPACT采用该算法 |
MSIsensor II |
n基于深度机器学习,在MSIsensor的基础上提出了一套标准位点集(2,828 位点),可仅需肿瘤组织,无需对照 n更高的灵敏度和特异性,更快的运行速度 n经第三方独立实验室(the Jackson Laboratory)验证,性能远优于MSINGS,MANTIS和MSIseq |
MSIsensor-ct |
n2021年发表在生信领域顶级期刊Briefings in Bioinformatics上(影响因子:11.622) n采用机器学习,MSIsensor-ct专门为基于外周血中cfDNA进行MSI检测进行了一系列优化 n3000X以上测序深度可探测到0.5%以下的cfDNA,ctDNA含量在0.05%以上时的准确率达到100% |
1.结直肠癌预后分层并不明确
2.探索MSIsensor2 3000余位点中哪些位点与Ⅱ、Ⅲ期结肠癌患者的复发及预后有着显著关联
3.建立结直肠癌预后评分系统,利于临床上的个性化精准诊疗和治疗方案决策
基于NGS的Ⅱ/Ⅲ期肠癌复发预后评分系统流程图
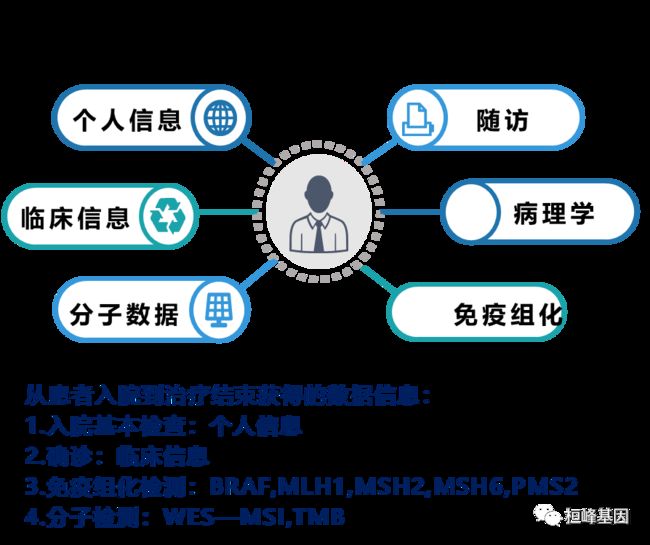
1,IHC确定dMMR型癌和癌旁配对病例XXX例
2,全外显子测序:癌200X,癌旁100X
3,收集患者相关数据
4,突变基因即差异基因分析统计
5,MSIsensor计算每个患者的MSIsensor score
6,计算每个患者的TMB
7,突变信号检测
8,KM生存分析
9,单因素,多因素分析确定预后独立因子
基于WES数据检测MSI——XX例配对样本临床信息
XX例癌和癌旁配对样本选取标准:
1.Ⅱ、Ⅲ期结肠癌(单纯接受手术治疗,未接受新辅助放±化疗)
2.患者类型均为dMMR
原发与复发差异基因——XX个基因与复发相关
利用突变位点计算差异突变基因(DMG),找到XX个基因与复发相关(p-value<0.01)
差异突变基因进行GO功能注释
1.ATPaseactivity紧密调节RecA核丝促进同源重组
2.damaged DNA blinding
XX例患者与TCGA-CRC的生存分析一致
差异基因Pathway分析——差异基因与肿瘤复发转移高度相关
1,对DMG进行Pathway分析,得到10个通路
2,NOTCH通路促进结直肠癌转移
3,NRF2是CRC进展的关键通路
探索预后MSI位点——差异位点
1,利用Fisher-test检验每个MSI位点,得到XX个差异位点
2,对差异MSI位点进行基因注释,得到XX个基因
3,对基因进行GO功能注释,得到XX个显著功能及对应基因如下:
PMS1:错配修复基因(Mismatch Repair, MMR)基因家族
RPA2,TTN,TPM1,SMYD3等基因与结直肠癌相关
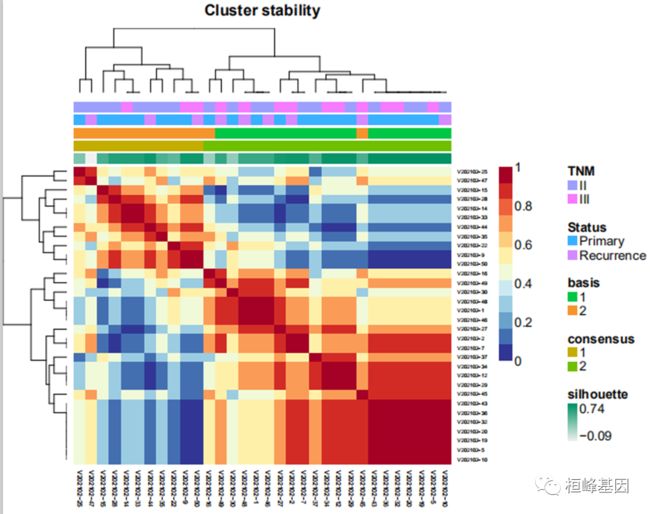
MSI Signature 与生存关系的显著性分析
1.利用NMF模型计算最优适合个数:2
2.利用2个cluster进行分析
dMMR结直肠癌患者TMB及MSI分布—复发的TMB在dMMR中明显升高
通过T检验,发现TMB在原发-VS-复发样本差异显著(p-value=0.013),而MSI有上升趋势但是不显著(p-value=0.22),但是TMB与MSI正相关(r2=0.357)。基于上述结果,进一步分析TMB对dMMR预后的影响。
TMB是结直肠癌dMMR人群中预后预测因子
将TMB以及MSI分成高低两组,做生存分析并计算AUC
1. TMB与生存显著相关即p-value=0.0056,
AUC 2年生存期准确率0.755,4年期0.758
2. MSI表型不明显即p-value=0.74,
AUC 2年生存期准确率0.596,4年期0.630
Mutational Signatrue构建结直肠癌预测模型
突变信号:细胞在成长过程中,基因组不断受到内源性和外源性DNA损伤的威胁,正是由于这些威胁,使得细胞基因组不断发生变化,并最终发生一些突变的积累。每一个突变过程都会留下一个不同的基因组标记,也就称为突变信号。
突变信号(Mutational Signatures)计算方法:
1.利用NMF模型计算最优适合个数;
2.与COSMIC数据库中的已知签名进行比较,并计算相似度以确定最佳匹配;
3.结果解读:5个Signatures均与DNA缺失及修复相关
TCGA数据库173个患者验证Signatures Model
突变信号(Mutational Signatures)计算方法:
1.利用NMF模型计算最优适合个数;
2.与COSMIC数据库中的已知签名进行比较,并计算相似度以确定最佳匹配;
3.结果解读:5个Signatures均与DNA缺失及修复相关
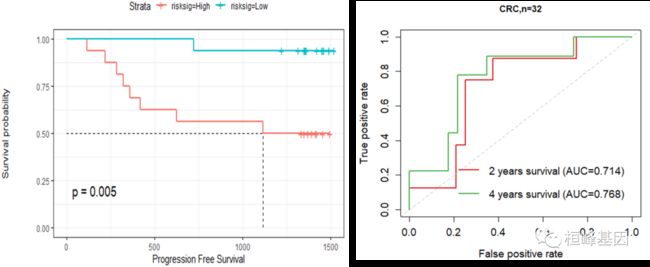
Signatures Model生存和AUC分析——RiskScore与预后相关
利用5-Signtures Model构建预后风险模型,进行生存相关性分析及准确率分析:
1.风险评分高度与生存相关(p-value=0.005)
AUC分析结果显示:2年预测准确率0.714,4年期预测准确率0.768
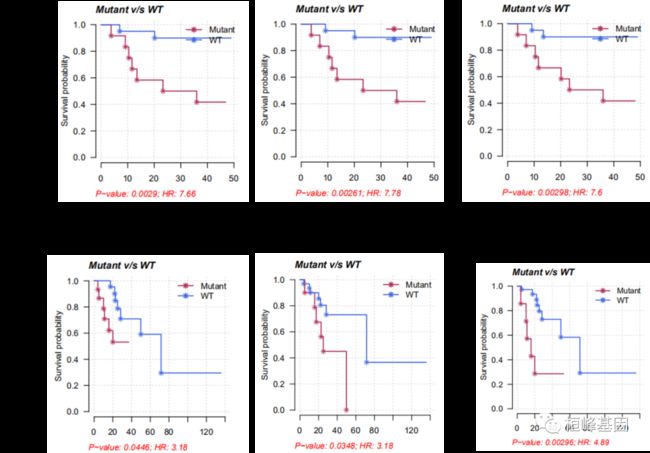
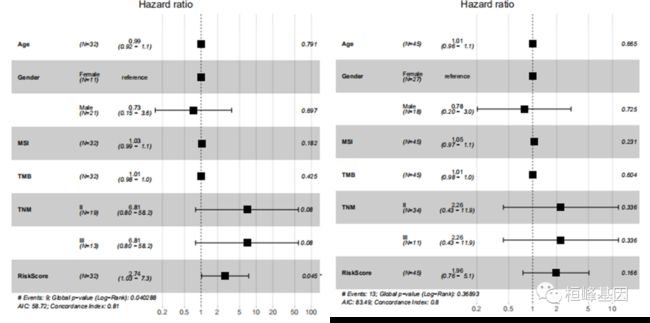
独立预后因子——TMB、TNM和RiskScore
多因素COX回归分析,确定预后独立因子:
1.TNM:多因素分析结果显著(p-vlaue=0.037)
2.5-Signatures Model的RiskScore:多因素分析结果显著(p-value=0.025)
3.TMB:单因素分析结果显著(p-value<0.023)
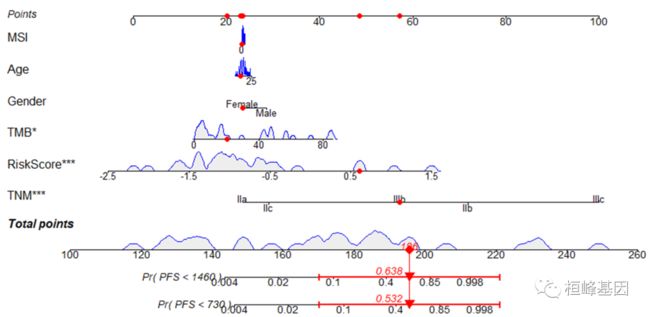
dMMR型结直肠癌预后评分系统——个性化精准评估预后复发风险
dMMR类型的直肠癌II,III期患者中,TMB,TNM, Signatures Model的RiskScore成为预后预测标志物
选取结直肠癌患者1例,计算预后复发风险及生存期预测,得到2年期概率0.532,四年期概率0.638
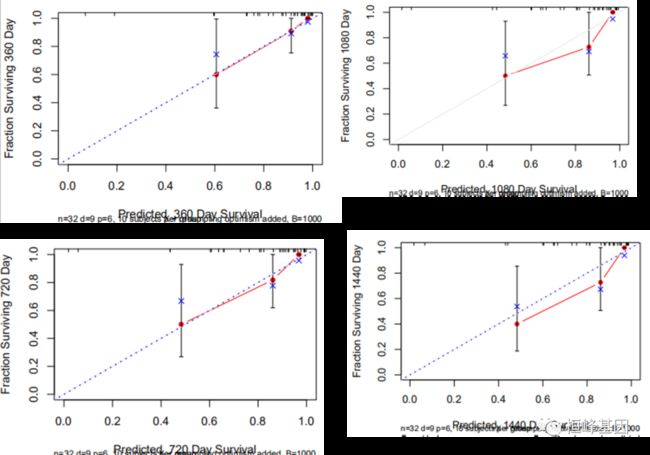
内部验证C-index及外部验证
一致性指数 C-index : 1-0.189 =0.811
独立验证
Reference:
Data Source: GLOBOCAN 2020
Z. Diao et al. Oncology / Hematology 157 (2021) 103171 2021
Cell Discovery volume 3, Article number: 16053 (2017)
Cancer cell, Volume 36, Issue 3, 16 September 2019, Pages 319-336.e7.
Cancer Res. 2021 Jun 23;canres.0453.2021
BMC Cancer. 2009 Aug 13;9:282. doi: 10.1186/1471-2407-9-282.
A. B. Schrock1, et al. Annals of Oncology 30: 1096–1103, 2019
Nature volume 500, pages415–421 (2013)
以上分析内容利用桓峰基因公众号上的教程完全可以实现这些图表的绘制,过程需要自己细致的探究,同时也需要有一定的生信分析和结果解读的能力,有需要此类分析的老师可以联系桓峰基因!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出临床数据分析系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!
http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
有想进生信交流群的老师可以扫最后一个二维码加微信
备注“单位+姓名+目的”!