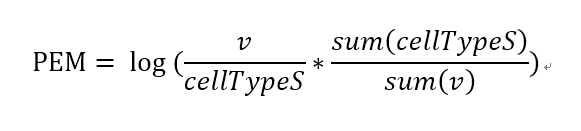生物信息学 | 富集分析
主要目标:理解这个代码的主要的思路。想分析一下老师的这个富集分析的主要的思路是什么?
一行一行的理解这个代码。
# Get cell type mean of each gene
cellTypeMean <- t(apply(dat, 1, function(v) {
tapply(v, droplevels(factor(cellSubtypes, levels=subtypeOrder)), mean)
}))
}
cellTypeMean2 <- cellTypeMean + 0.01
(1) droplevels()是什么意思
> x <- c(1, 3, 4, 8, 1, 5, 4, 4, 5, 6)
> f<-factor(x)
> f
[1] 1 3 4 8 1 5 4 4 5 6
Levels: 1 3 4 5 6 8
> f<-f[-2]
> f
[1] 1 4 8 1 5 4 4 5 6
Levels: 1 3 4 5 6 8
> new_f <-droplevels(f)
> new_f
[1] 1 4 8 1 5 4 4 5 6
Levels: 1 4 5 6 8
这个的意思就是说,当我们剔除factor中的某一个数值的时候,其levels仍然会被保留下来。(这个问题,我之前也有遇到过)所以droplevels的意思就是把那些冗余的levels也去冗余掉。这就是这个函数的功能。
(2) factor()
factor(cellSubtypes,levels=subtypeOrder)
这里我不理解,factors()函数中的levels=subtypeOrder是什么意思?
a<-c(1,3,6,8,5,4)
factor(f,levels = a)
[1] 1 4 8 1 5 4 4 5 6
Levels: 1 3 6 8 5 4
它的意思是对level的顺序进行规定,按照我们level的是顺序。
所以经过上述探索,我们明白上面那一行代码的意思是计算属于每一种细胞类型的细胞表达值得均值。
# get specificity scores
cellTypeS <- colSums(cellTypeMean2)
cellTypePEM <- t(apply(cellTypeMean2, 1, function(v) {
log10(v/cellTypeS*sum(cellTypeS, na.rm=TRUE)/sum(v, na.rm=TRUE))
}))
这里是计算数据集中的每一个基因相对于每一个细胞类型的特异性的值。用数学公式可以表示为:
至于这个PEM值为什么能够表示这个基因在这种细胞类型中的富集程度我是不太明白的。
那么通过这种方法,可以分别计算出每一个基因在上述给定的细胞类型中的富集的值。
我突然有一点比较朦胧的认知:觉得数学就是表征关系。将我们遇到的情况,表征为数与数之间的关系。
下面这一部分就是,这里最升华的一部分。
# KS test (m is the PEM score matrix)
getKS.p <- function(genes, m) {
selDat <- m[rownames(m) %in% genes, ]
bgDat <- m[!(rownames(m) %in% genes), ]
p <- sapply(1:ncol(selDat), function(i) {
ks.test(selDat[,i], bgDat[,i], alternative="less")$p.value
})
names(p) <- colnames(m)
return(p)
}
这行代码的意思,就是使用KS-test去检验,我们感兴趣的一群基因在特定细胞类型中的富集程度与相应的背景基因集去比较,去比较这两者的分布是否一致。
对于统计学的还是真糟糕,这里的零假设是啥都没有反应过来。
通过最后得到的P值,我们确定我们感兴趣的这群基因在特定细胞类型中的富集程度。
这是一种判断的思路(关于富集分析还有其他方法,立志等我学会了,我就再写一篇博客。)