基于cat12和SPM12进行VBM数据分析
一、基于cat12和SPM12进行VBM数据分析
VBM 能定量计算局部灰质体素的大小和信号强度,从而能够检测出局部脑
区的特征和脑组织成分的差异。
1.前期软件安装准备:
1.1 spm12和cat12软件安装
参考这篇文章操作:
https://blog.csdn.net/qq_44354520/article/details/103951458?ops_request_misc=%257B%2522request%255Fid%2522%253A%2522169131449516800188580354%2522%252C%2522scm%2522%253A%252220140713.130102334..%2522%257D&request_id=169131449516800188580354&biz_id=0&utm_medium=distribute.pc_search_result.none-task-blog-2~all~sobaiduend~default-4-103951458-null-null.142^v92^chatgptT0_1&utm_term=spm12%E5%AE%89%E8%A3%85%E6%95%99%E7%A8%8B&spm=1018.2226.3001.4187
1.2 dpabi软件安装
根据这篇文章操作:
https://blog.csdn.net/qq_42356925/article/details/123928073
2.VBM图像预处理一般步骤
2.1图像格式转换
将原始的磁共振文件.dcm文件转换成spm以及cat能够处理的格式.nii文件。这里格式转换的方式有很多,比如使用dcm2niigui转换工具,或者直接使用dpabi软件进行转换,这里演示如何用dpabi进行图像格式的转换。
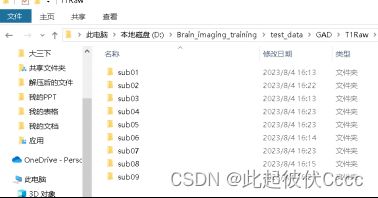
(1) 将工作路径切换至图像数据存在的地方,并在该路径下按如下图格式创建文件夹,其中T1Img文件夹中存放未经过格式转换的被试.dcm文件。
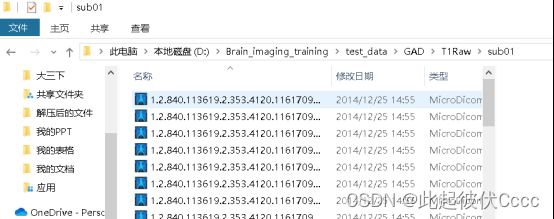
每个subxx文件存放被试的.dcm图像文件:
(2)安装好dpabi后在Matlab中键入dpabi进入dpabi操作界面,并点击如下图所示的dpabi按键进入图像处理界面。
(3)按照下图所示进行格式转换,选择好后点击Run即可。
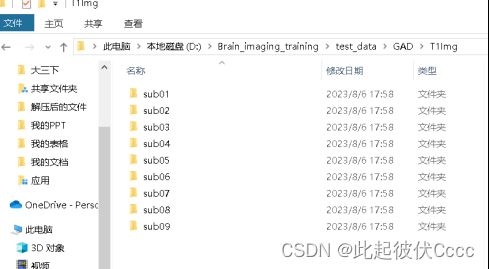
在与T1Raw相同文件夹的T1Img文件中生成了图像格式转换后的.nii文件,如下图所示:
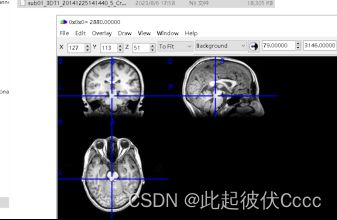
其中sub01_3DT1_20141225141440_5_Crop_1.nii文件便是我们后期需要处理的.nii文件,用mricron软件查看图像如下图所示:
2.2 检查图像,调整原点
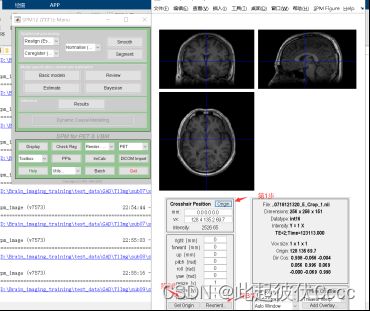
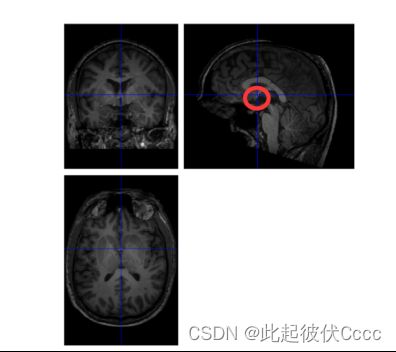
首先就是需要我们自己手动调整一下结构像,打开spm,点击spm首页的PET&VBM,然后点Display,摆正每个被试的 T1 数据,并使 AC-PC 的连线水平。
手动将蓝色十字线的交叉点放到大概下面这个位置,即前联合的位置。
2.3 原始脑结构图分割
基于cat12对被试者的原始脑结构图进行分割,分割成灰质、白质和脑脊液三个部分。
(1)运行Matlab2018a,在命令框中输入:spm敲回车,调用spm工具包。
(2)在SPM for functional MRI部分找到Toolbox,下拉找到cat12,点击打开cat12工具包,之后便会出现工具包cat12的操作界面,如下图:
(3)点击segment,进入如下界面:
选择需要处理的T1图像,参数配置好后点击左上角的小三角运行即可:
点击后出现如下运行界面:
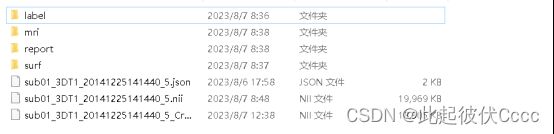
(4)最终会在每个被试文件夹中生成三个文件夹——mri(分割之后的图像)、report(数据分割过程及结果存)、label。
其中在mri文件夹内有分割后的文件。mwp1对应灰质, mwp2对应白质,report中有分割质量报告。
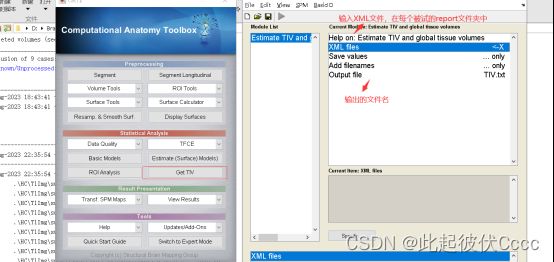
2.4 体积数据文件获取
回到cat12操作主界面,在statistical analysis部分,点击Get TIV,如图所示:
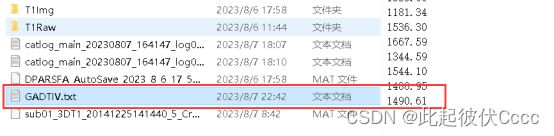
输入文件后点击运行,在相应的默认文件夹下生成输出的.txt文件:
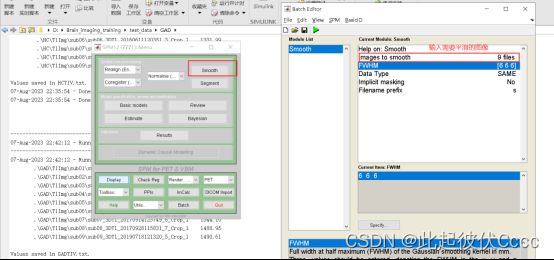
2.5 对图像进行平滑
运行spm12,点击Smooth,按下图操作,平滑核大小参考文献选择。
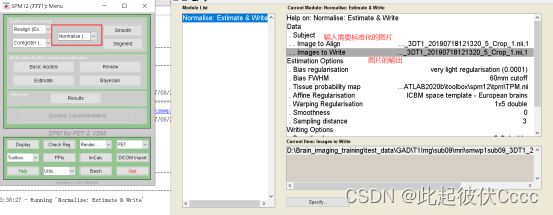
2.6 对图像进行标准化
使用spm进行标准化,这里选择上一步以及平滑后的图片
3.VBM分析过程
3.1 生成设计矩阵
(1)点击上图中的圈1(Specify 2nd-level)按钮,进入以下界面:
(2)双击Directory,这个是需要你选择你的输出结果放置的文件夹
(3)点击Design,选择two-sample t-test,接下来给Group 1 scans和Group 2 scans导入被试的结构特征数据。
以TIV(之前在2.4步保存的文件)、年龄、性别作为协变量回归。(注意顺序)
4.点击RUN运行后在之前设置的输出路径下生成.mat文件
3.2 组间分析
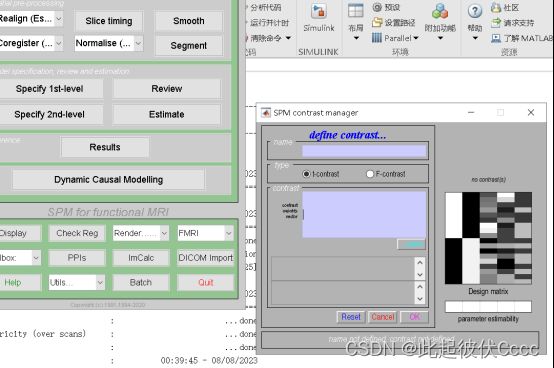
1.点击Estimate,选择上一步生成的SPM.mat文件,点击run运行
2.点击Result,点击t-contrasts按钮,然后点击Define new contrast按钮
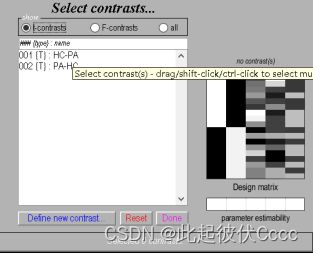
3.输入对比矩阵名称如HC-PA,输入对比矩阵,1 -1表示第一组减去第二组,点击OK,点击Done,即添加了一组,这里添加两组,分别为HC-PA与PA-HC,如下图所示:
4.点击Done,之后按下图进行选择
5.出现结果
6.将结果保存,点击Save->threshold SPM,之后再给文件取名字回车即可