热点综述 | 跨模态单细胞分析的最佳实践
跨模态的单细胞数据的可用性越来越高,推动了新的计算方法的发展,以帮助科研人员获得生物学见解。近日《Nature Reviews Genetics 》发表了一篇综述文章,总结了单模态和多模态单细胞数据分析的独立基准研究,为最常见分析步骤提供全面的最佳实践工作方案。
跨模态单细胞分析
转录组
scRNA-seq测量每个细胞的mRNA 分子丰度。提取的生物组织样本构成了单细胞实验的输入。组织在单细胞解离过程中被消化,然后进行单细胞分离以分别分析每个细胞的 mRNA。
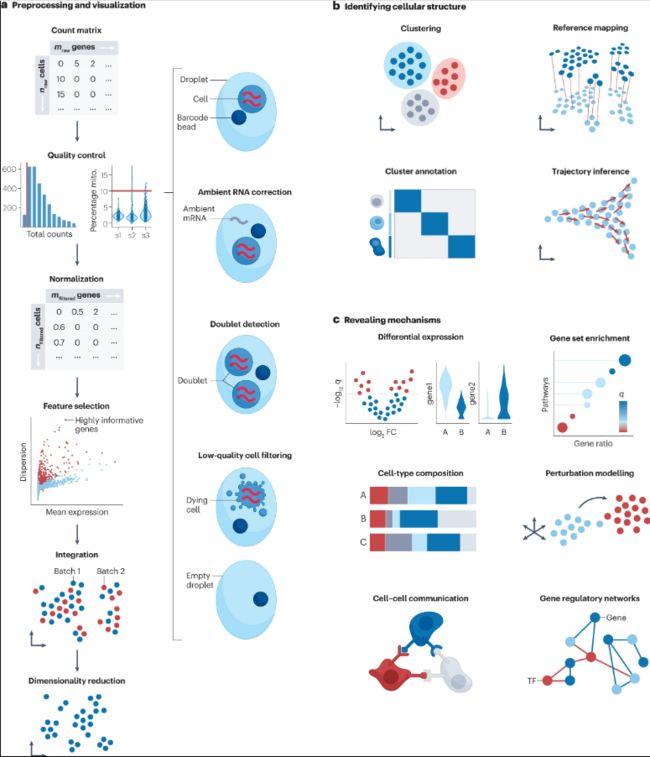
scRNA-seq单模态分析步骤概述
从原始计数矩阵到高质量的细胞数据
过滤低质量细胞和噪声校正:SoupX 等流行方法根据数据集中其他“空”液滴和细胞簇的表达谱估计细胞特异性污染分数;CellBender 将环境 RNA 的去除制定为无监督贝叶斯模型;scDblFinder在二重检测精度和计算效率方面优于其他方法。当低质量细胞和双细胞聚集在一起时,通常需要在下游分析期间重新评估所选的质量控制策略。因此,我们建议最初设置允许阈值,并可能在(重新)分析期间根据需要移除更多细胞。
归一化和方差稳定:最近的一个基准测试比较了基于KNN图与ground truth重叠的单细胞数据的22个转换,具有大小因子s的移位对数变换log(y/s+1)表现良好,但不应将每百万计数用作输入;表现类似良好的一种方法是皮尔逊残差的分析近似,它拟合以排序深度为协变量的广义线性模型,以获得变换的计数矩阵。归一化方法应该在后续分析任务的基础上仔细选择。移位对数被证明能更好地稳定方差,用于后续的降维,Scran在批量校正任务中表现良好,分析Pearson残差更适合选择生物可变基因和鉴定稀有细胞身份。
消除混杂的变异来源:一项基准比较了14个指标的16种集成方法,这些方法基于批量校正和生物方差守恒。线性嵌入模型,如正则相关分析和Harmony,在具有不同批处理结构的更简单集成任务上表现良好。根据集成任务的复杂性,如图谱集成,深度学习方法(如scANVI、scVI和scGen)以及线性嵌入模型(如Scanorama)表现最好,而对于不太复杂的集成任务,Harmony是首选方法。scIB包可用于使用上述基准的评估指标来评估集成。除了计数采样效应外,scRNA-seq数据可能包含生物混杂因素(如细胞周期效应),Tricycle被证明对具有高细胞类型异质性的数据集表现良好。
选择信息特征和降维:在不影响小亚群可识别性的情况下,特征选择方法应理想地选择解释数据集中生物变异的基因,方法是优先考虑亚群之间而不是一个亚群内的变异基因。Deviance在识别亚群中具有高变异性的基因方面表现良好,从而在选择信息基因方面也表现良好。在特征选择之后,可以通过主成分分析(PCA)等降维算法进一步降低数据集的维数。
从聚类到细胞识别
单细胞聚类:识别细胞群体的第一步是将细胞聚类成具有相似表达谱的组,以解释数据中的异质性。独立的基准测试表明,通过Louvain算法基于图模块化优化的聚类检测最适合于聚类识别。Louvain的继任者Leiden通过产生有保证的连接细胞群来规避连接不良的问题,并且在计算上更高效。
细胞类型注释:建议采用三步方法,利用自动注释,然后是专家手动注释和最后一步验证,以获得理想的注释结果。第一步,自动细胞类型注释,可以分为基于分类器的方法(例如CellTypist和Clustifyr)和参考映射(例如 scArches、Symphony或Azimuth),第二步,手动注释,利用每个簇的基因特征来注释细胞簇。这些基因特征通常被称为标记基因,可以使用简单的差异表达测试方法(例如 t 检验或 Wilcoxon 秩和检验)来识别,作为最后一步,注释应由专家验证,特别是对于具有高复杂性的数据集或涉及可能无法获得参考的稀有细胞亚群的研究。
从离散状态到连续过程:轨迹推理方法的性能取决于数据集中存在的轨迹类型,Slingshot在简单拓扑上表现更好,PAGA和RaceID/StemID在复杂轨迹上得分更高。因此,我们建议使用 dynguidelines来选择适用的方法。为了推断动态、定向信息,velocyto和scVelo模型使用未剪接和剪接reads来推断RNA 速度。谱系追踪数据的分析可以使用Cassiopeia 进行。
揭示机制
差异基因表达(DGE)分析:DGE分析目前从两个角度进行。样本级视图聚合每个样本-标签组合的计数,以创建pseudobulks,使用最初设计用于批量表达分析的包进行分析,如edgeR、DEseq2或limma(推荐这些方法允许进行复杂的实验设计)。或者,细胞级视图使用广义混合效应模型(例如MAST)单独地对细胞进行建模。目前DGE分析方法仍然显示出真实阳性率(TPR)和精确度之间的权衡,在DGE分析之前,应通过聚集个体内的细胞类型特异性计数来说明样本内的相关性。
基因集富集分析:常见的数据库包括 MSigDB、Gene Ontology、KEGG 或 Reactome。这一概念的扩展是加权基因集,包括用于信号通路的PROGENy和用于转录因子 (TF) 的DoRothEA。常用的富集方法包括超几何分布检验、GSEA或GSVA,可在 DGE分析后或在单个细胞水平上应用。基因集富集分析对基因集的选择比统计方法更敏感;因此,我们建议仔细选择数据库,以确保潜在的基因集被覆盖。decoupleR 等丰富框架在单个工具中提供了对不同数据库和方法的访问;为批量转录组学开发的富集方法可以应用于scRNA-seq,但一些基于单细胞的方法,即Pagoda2可能优于它们。
破译细胞组成的变化:专门为使用细胞类型计数的单细胞数据设计的测试包括scDC、scCODA和tascCODA,它们可以包含分层细胞类型信息。DA-seq和MILO使用KNN图来定义在实验条件之间测试差异丰度的亚群。
推断扰动效应:扰动建模的一个领域试图在未知任务的实验装置中成功区分未成功的靶向细胞,并评估扰动效应。Mixscape和MUSIC首先去除了混淆的变异源,然后成功地从未成功扰动的细胞中进行解剖,最终对扰动效应进行可视化和评分。Augur和MELD仅涵盖第三步,并根据扰动响应的程度对细胞类型进行排序,以确定受扰动影响最大的细胞群体。扰动建模的第二个领域涉及未经实验测量的扰动。scGen、CPA和CellBox等潜在空间学习模型旨在预测对不可见扰动、组合或药物剂量的反应。此类模型通常适用于高表达基因,但由于缺乏方差,可能难以处理低表达基因。
跨细胞的通信事件:方法和相互作用数据库的选择对预测的相互作用有很大影响。CellChate和CellPhoneDB也考虑了异构相互作用复合物,以及SingleCellSignalR被发现对数据和资源噪声都很稳健。我们建议使用LIANA,它为方法和数据库的几种组合提供了总体排名。此外,可使用Nichenet或Cytotalk等工具提供细胞内活动的补充估计,例如诱导的基因表达变化或空间信息,可用于增加预测相互作用的置信度。
染色质可及性
分析调控元件对于解读细胞多样性和理解细胞决策至关重要。基因表达受调控机制的复杂相互作用控制,包括表观遗传学和染色质可及性。为了深入了解单细胞水平上染色质状态的动力学,scATAC-seq测量单个细胞中全基因组染色质的可及性。
scATAC-seq分析步骤概述
特征定义和质量控制
scATAC-seq数据由于数据的全基因组性质而缺乏标准化的特征集。大多数工作流程使用逐个峰或逐个细胞的矩阵作为分析的基础,其性能优于基因矩阵或TF motif特征。scATAC-seq质量控制最常见的入口点是片段文件,其中包含由两个相邻的Tn5转座事件生成的所有已测序 DNA 片段。这些用于计算一组scATAC-seq特异性质量度量,以确定低质量细胞。与scRNA-seq数据中的测序深度相比较,检查了每个细胞的测序片段总数、片段对数总数和转录起始位点(TSS)富集分数。低质量的细胞通常会形成一个集群,将低计数和低TSS富集分数结合在一起,这些分数应该被去除。此外,核小体信号用于评估片段长度分布。进一步建议验证映射到与伪影信号相关的基因组区域的读数的比率。为了对双因子进行评分,我们建议遵循Germain等人的建议。使用两种专门为scATAC-seq数据设计的正交方法,并在下游分析中考虑这两种评分。第一种方法是调整scDblFinder;第二种是AMULET。
数据降维表示
在scATAC-seq数据中,最常见的归一化策略是峰的二值化。然而,这也可能会删除生物信息,因此建议直接对scATAC计数进行建模。基于潜在语义索引(ArchR 和 Signac)、潜在狄利克雷分配(cisTopic)和光谱嵌入(snapATAC)的降维方法被证明对下游聚类和细胞注释表现最佳。关于批量校正,LIGER 被证明对scATAC-seq数据表现最佳。最近,针对scATAC-seq数据提出了 PeakVI或MultiVI等深度学习模型作为降维和批量校正相结合的方法。在获得校正后的低维表示后,我们推荐Leiden聚类,因为它在scRNA-seq衍生表示中具有良好的性能。
基于可访问区域细胞类型注释
可以根据差异可及区域 (DAR) 和基因活性评分对细胞簇进行注释。DAR可以通过类似于scRNA-seq的差异测试方法获得,现有的批量ATAC-seq数据基准建议在样本量有限时使用edgeR来确定DAR,在大样本量的情况下使用DESeq2。DAR可能包含信息序列模式,例如已知的顺式调控元件 (CRE),或者可以链接到近端基因,这在功能丰富分析工具(例如GREAT、LOLA或GIGGLE)中得到利用。与基因相关的CRE的染色质可及性可以概括为基因表达的估计(基因活性评分)。为了指导细胞类型注释,简单的模型通常就足够了,并且可以通过平滑相邻细胞之间的基因活性评分来增强可视化,这通常使用MAGIC进行。
TF-motif分析
为了获得每个细胞的富集分数,chromVAR 可用于计算每个细胞所有包含基序的峰的可及性偏差,同时校正Tn5转座酶的插入偏差,这是由转座酶的序列结合偏好引起的。TF标记有助于簇注释并代表决定细胞状态的调节蛋白的最佳候选者。一旦确定了感兴趣的TF,scATAC-seq数据就可以通过足迹进一步验证 TF 的影响。当前的足迹工具通常使用k-mer模型来纠正此偏差,该模型通过每个k-mer内的切割位点数量相对于全基因组出现次数来估计偏差。
将单细胞染色质可及性与转录组联系起来
专有的10x Multiome、sci-CAR 或 scCAT-seq 等分析允许对基因表达和染色质可及性进行联合分析。当前的工作流程使用已建立的方法进行单模态质量控制,并采用所有模态的高质量细胞的交集进行综合分析。由于尚未确定此集成的最佳方法,我们建议首先执行单模态分析,包括细胞类型注释。这可以通过将更新的聚类结果与单模态分析的细胞类型标签进行比较来评估联合表示。然后,高质量的多模态表示可作为大多数单模态分析方法的输入,包括细胞类型注释、差异测试和轨迹分析。
配对的scRNA-seq和scATAC-seq数据还支持使用新的联合方法来识别基因表达和细胞状态的调节因子。为了识别潜在的CRE,使用基于相关性的方法将峰与细胞簇内的基因联系起来。可以通过使用SCENIC推断活性TF,然后将相应的基序与峰值区域匹配以增加额外的可解释性来扩展这种方法。为了深入了解局部或全局染色质景观是否影响特定细胞状态下基因的表达,可以比较基于局部邻域和全基因组染色质状态表达的可预测性。目前正在开发利用两种模态(例如FigR或Pando)推断基因调控网络的方法。
表面蛋白表达
用于结合scRNA-seq和表面蛋白分析的最广泛使用的方案是CITE-seq和REAP-seq,主要区别是用于量化表面蛋白表达水平的抗体衍生标签(ADT)。
CITE-seq数据处理概述
校正ADT计数
应在ADT模式中对单个质量控制指标进行仔细评估,RNA和ADT的联合测量应分别进行质量控制。由于抗体功效是可变的,跨多项研究的 ADT 数据整合可能导致强烈的批次效应,需校正。
计算ADT成分偏差
细胞特性会导致异质捕获效率,从而导致细胞组成偏差。只有表达目标蛋白的细胞会导致标签计数增加,这可能只是特定的细胞类型。这可以通过使用中心对数比 (CLR) 转换或按背景去噪和缩放 (DSB) 进行归一化来解决。
联合分析转录组学和ADT数据
在各自的预处理之后,可以使用通常适用的多模态集成工具或CITE-seq特定的、基于深度学习的totalVI来获得联合嵌入,该工具学习成对测量的联合概率表示,该联合概率表示也考虑了噪声和技术偏差,包括每个模态的批处理效应。另一种方法是使用CiteFuse,它使用CLR对ADT进行归一化,并将两种模态矩阵与相似性网络融合算法相结合。然后,可以使用Leiden对联合嵌入进行聚类,并通过将聚类与所有其他聚类进行比较,使用Wilcoxon秩和检验基于差异表达的RNA和ADT进行注释。这两种模态都可用于下游任务,如研究细胞-细胞通信,其中考虑配体簇的RNA表达和受体簇的蛋白质表达,或使用CiteFuse进行RNA和ADT相关性分析。
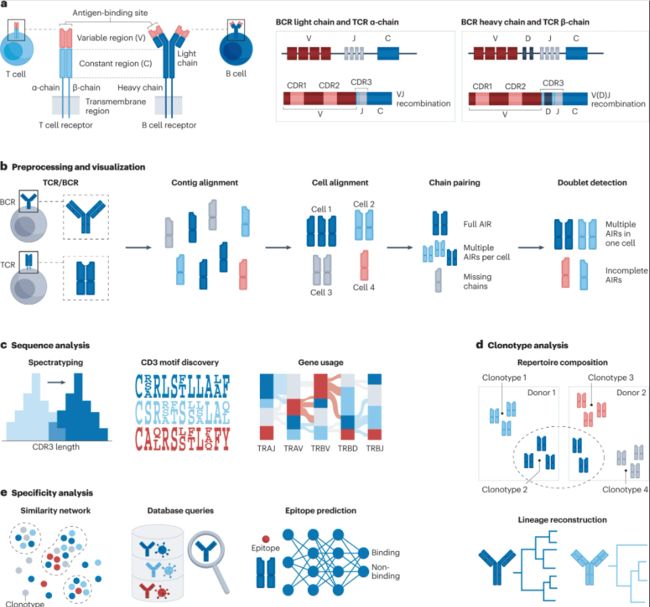
单细胞免疫组库
商用10x Chromium单细胞免疫分析和BD Rhapsody TCR/BCR 多组学分析能够生成配对的转录组学和AIRR数据。可以使用 scirpy、Dandelion或scRepertoire等流程进行免疫受体分析,包括解码AIRR序列特征、筛选功能性适应性免疫受体、克隆型的鉴定和分类、确定细胞特异性、将适应性免疫受体与转录组学测量相结合等。
适应性免疫受体分析概述
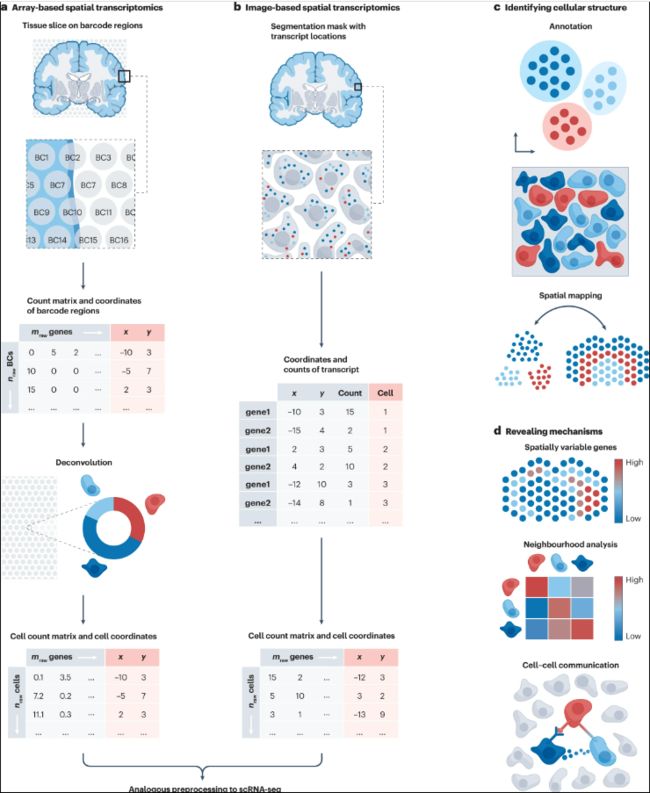
在空间中解析单细胞数据
分析空间数据集需要专门定制的分析工具,可以使用Squidpy、Giotto、Seurat或SpatialExperiment等流程进行分析。
空间转录组学预处理和下游分析步骤概述
获得细胞计数矩阵和空间坐标
已经提出了多种方法来分解基于芯片的基因表达谱中的基因表达谱。Cell2location、SpatialDWLS和RCTD根据单细胞解析参考中细胞群的基因表达谱估计每个点的细胞类型组成。对于模拟数据集,Cell2location优于其他细胞类型去卷积方法,但需要更多的计算资源,而对于真实数据集,SpatialDWLS和 RCTD在基于四种不同精度指标的总体精度得分方面表现最佳。
对基于图像的技术,Giotto和squidpy等处理流程允许在其中添加定制的分割方法,这简化了对所选方法的比较、选择和评估。此外,转录物的定位可以用于无分割的方法,如SSAM或Baysor,这些方法直接将细胞标签分配给空间邻近的像素。Baysor还结合了通过组织学图像获得的细胞形状信息,以增强分割结果。这些工具可以作为基于分割的方法的有用替代方案。
细胞特性和细胞微环境的表征
对于单细胞分辨率的基于成像的空间转录组学数据,可以类似于scRNA-seq数据对细胞进行注释。标准空间原始scRNA-seq 数据和目标空间解析数据的对齐能够以空间解析的方式对整个转录组(以标准scRNA-seq测量)进行插补,Tangram通过优化空间和 scRNA-seq 数据之间的基因相似性来估算空间样本中未检测到的转录本。它被证明在各种准确性指标和可扩展性方面优于其他插补方法(例如 gimVI和SpaGE)。
除了仅根据基因表达谱来注释细胞外,还可以利用空间位置来识别细胞身份。BayesSpace、stLearn和spaGCN等工具通过考虑基因表达共性和空间邻域结构来识别空间域。获得的标签可用于识别组织中具有相似表达谱的区域,并且可能对应于数据集的整体形态。
不同样品的细胞微环境的识别可能会因图像取向的差异而受到阻碍,Tangram, GridNet和eggplant生成跨样本的通用坐标框架以缓解此问题。
识别与细胞组织和组织结构相关的空间模式
scRNA-seq在鉴定高度可变基因和DGE分析方面对基因表达差异的分析进行了广泛的探索。对于空间转录组学数据,这是通过空间可变基因(SVG)的鉴定来补充的。为此目的的方法在其假设和SVG的定义方面存在很大差异,并且在如何最好地识别SVG方面没有达成共识。例如,SPARK和SpatialDE利用空间相关性测试,BayesSpace使用马尔可夫随机场,spaGCN使用图神经网络来整合基因表达数据、空间信息和组织学图像,sepal使用基于扩散的建模来识别具有空间模式的基因。
细胞间依赖空间的通信事件
空间细胞间通讯的方法通常比较基于周围相邻细胞的基因表达模式。GCNG、Misty和 NCEM根据细胞空间图和图神经网络制定此任务,SpaOTsc使用最优传输,SVCA通过空间方差分量分析量化细胞间通讯事件对基因表达谱的影响。
参考文献
Heumos, L., Schaar, A.C., Lance, C. et al. Best practices for single-cell analysis across modalities. Nat Rev Genet (2023). https://doi.org/10.1038/s41576-023-00586-w