基于Python根据置信度区间计算植被覆盖度
“把别人的经验变成自己的,他的本事就大了”
1 简述
大概九天前,我发了篇记录,大致是讲用Python计算Landsat8遥感生态指数RSEI,也就是下篇
“基于Python计算Landsat8 OLI遥感生态指数RSEI”
其中在2.6部分,计算LST时,涉及到植被覆盖度的计算,当时用的是一个经验模型,但这其实是不完全合理的,因为随着时间和空间的变化,NDVI的值也会随之改变,甚至对于同一幅影像来说由于研究区的不同的原因,所取的值都可能是不一样的,这里要非常感谢同学“张大佬”的指正和建议,因此本次是对植被覆盖度的一次新的计算。
同时,因为我也是个初学者,以前接触过python基础,跟着网上断断续续的学了很久,毕业了又捡起来了,是个纯菜鸡,所以写的内容可能比较小白,也比较啰嗦,如有不足,请多指正!
2 主要内容
- 使用rasterio查看数据的详细信息
- 植被覆盖度简介
- 计算NDVI
- 3.1 去除NDVI的异常值
- 根据置信度区间获取对应的NDVImin、NDVImax
- 计算植被覆盖度
- ENVI验证(ps:如果我的ENVI还能用的话)
- 完整代码
3 环境
- Jupyter Notebook
- python 3.9
- 主要包 numpy rasterio matplotlib
4 详细过程
本次使用的影像数据来源于GEE,tif格式,数据已经预处理,包含波段为
B1 B2 B3 B4 B5 B6 B7 B10 B11
4.1 使用rasterio查看数据的详细信息
如果会的朋友可以直接跳过此部分哦!
# 首先导入所需的包
import numpy as np
import rasterio as rio
import matplotlib.pyplot as plt
# 影像存放的路径
path = r'G:\Project\geo\data\test_clip.tif'
rasterio使用rasterio.open()函数获取影像,其中传入的一般是数据的路径,返回一个数据集的对象,在获取到数据集后,我们就可以得到数据的一些基本信息。open()默认的模式是读,我们也可以改为写模式,也就是保存影像,需要将’w’即写传入参数中。
比如影像的波段数目、影像的行列、数据的类型、影像的地理转换信息和数据集的投影等等,请注意,我在导rasterio包时对其命名成了rio,所以代码中出现的rio也即是rasterio
# 获取影像数据集
data = rio.open(path)
# 影像的波段数目
count = data.count
# 影像的宽(列)
width = data.width
# 影像的高(行)
height = data.height
# 数据的类型
type = data.dtypes
# 影像的地理转换信息
transform = data.transform
# 影像的投影
crs = data.crs
count
9
width
977
height
755
type
('float64',
'float64',
'float64',
'float64',
'float64',
'float64',
'float64',
'float64',
'float64')
transform
Affine(30.0, 0.0, 269130.0,
0.0, -30.0, 4227510.0)
crs
CRS.from_epsg(32650)
可以看到,在读取数据后成功获取到数据的一些基本信息,影像包含9个波段,宽977列,高755行,9个波段的数据类型都是float64,tansform是Affine(30.0, 0.0, 269130.0, 0.0, -30.0, 4227510.0),坐标系是EPSG:32650(关于EPSG如有不了解的可以了解下,算是相当重要的一个基础点)
不知道看到这的朋友是否会有疑问,这些信息获取到后有什么用?就这样看看吗?反正我学的时候是很不理解,就感觉这有啥用,后来在把计算后的数据保存下来时才知道,比如保存计算后的NDVI到新的tif中,那这些是保存时所必须的条件,不然我们就只是把一堆数据塞进了一个文件中甚至有可塞不进去。在保存文件的时候我就需要给这个文件定义行列、投影等这些基本信息,那肯定是需要和原始影像保持一致,但一个个获取很繁琐,这些信息都属于元数据,所以,也可以直接获取影像是元数据信息
# 获取影像的元数据
meta = data.meta
meta
{'driver': 'GTiff',
'dtype': 'float64',
'nodata': None,
'width': 977,
'height': 755,
'count': 9,
'crs': CRS.from_epsg(32650),
'transform': Affine(30.0, 0.0, 269130.0,
0.0, -30.0, 4227510.0)}
计算数据肯定需要获取波段的值,rasterio使用read()函数获取影像的波段值
# 获取波段1的值
band1 = data.read(1)
band1
array([[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]])
band2 = data.read(2)
band2
array([[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]])
# 获取所有波段值
bands = data.read()
bands
array([[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]],
[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]],
[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]],
...,
[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]],
[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]],
[[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]]])
如上所示,当我们想获取单个波段的时候直接在read()函数中指定波段数就行,当想读取所有波段值时不传参即可,您可能发现以上读取的所有值都是nan,这是因为我的这个影像是个不规则的,经过裁剪,所以在没有影像的部分都是nan值,由于显示原因无法全部显示出。
另外,请注意,rasterio在读取波段时的索引是从1开始,而不是0,如果传0会报错,如下所示
band_test = data.read(0)
band_test
---------------------------------------------------------------------------
IndexError Traceback (most recent call last)
g:\Project\geo\code\基于python计算植被覆盖度.ipynb Cell 20 in | ()
----> 1 band_test = data.read(0)
2 band_test
File rasterio\_io.pyx:249, in rasterio._io.DatasetReaderBase.read()
IndexError: band index 0 out of range (not in (1, 2, 3, 4, 5, 6, 7, 8, 9))
| 可以从报错信息中看到,0不在索引范围内,其索引是从1开始。
以上就是本部分的关于rasterio查看获取影像数据信息的简单介绍和演示。
4.2 植被覆盖度简介
植被覆盖度可以根据像元二分模型计算,基本原理是假定植被覆盖部分地表和无植被覆盖部分地表构成的一个像元,如果全植被覆盖的像元信息为Sveg,土壤覆盖的像元信息是Ssoil,那么可以得到植被覆盖度的计算公式如下
F c = S − S s o i l S v e g − S s o i l Fc = \frac {S - Ssoil} {Sveg - Ssoil} Fc=Sveg−SsoilS−Ssoil
植被覆盖度和NDVI之间有着显著的线性相关关系,根据像元二分模型,一个像元的NDVI值由植被贡献的新NDVIveg和无植被覆盖部分贡献的信息NDVIsoil组成,所有可以将NDVI带入公式进行估算,可得
F c = N D V I − N D V I s o i l N D V I v e g − N D V I s o i l Fc = \frac {NDVI - NDVIsoil} {NDVIveg - NDVIsoil} Fc=NDVIveg−NDVIsoilNDVI−NDVIsoil
而NDVIveg和NDVIsoil应用中通常根据实际影像中的置信区间取NDVI的最大最小值
关于植被覆盖度的代表这个Fc,我看到的有的写FVC、FC、PV等,我也不知道哪个正确,如果有了解的可以告诉下哈,感谢!
关于更多有关植被覆盖度的专业知识可以参考论文
- 李苗苗、吴炳方、颜长珍等 密云水库上游植被覆盖度的遥感估算
- 穆少杰、李建龙、陈奕兆等 2001年-2010年内蒙古植被覆盖度时空变化特征
4.3 计算NDVI
从植被覆盖度公式中可以知道,我们首先需要计算影像的NDVI值,NDVI的计算公式为
N D V I = N I R − R E D N I R + R E D NDVI = \frac {NIR - RED} {NIR + RED} NDVI=NIR+REDNIR−RED
其中,NIR指的是近红外波段,RED指的是红外波段
在Landsat8中B4是红外波段,B5是近红外波段,所以Landsat8计算NDVI的公式为
N D V I = B 5 − B 4 B 5 + B 4 NDVI = \frac {B5 - B4} {B5 + B4} NDVI=B5+B4B5−B4
4.3.1 去除NDVI异常值
NDVI的值域范围一般是处于-1到1之间,将小于-1和大于1的值归为异常值,并将其赋值为nan值
此处用到了numpy的where()函数,关于具体的函数可以查询,这里用到的主要是通过条件判段计算后的ndvi数组中哪些值处于(-1,1)之间,如果处于则返回原来的NDVI值,如果不属于,则将返回np.nan即nan值,从而达到一种去除异常值的目标。当然,如果你想赋值别的值也可以更改。
4.3.2 代码
为了方便后面使用,将NDVI的计算代码写成函数
def ndvi(red, nir):
"""
计算植被指数 ndvi
:param red: landsat8的B4波段,红外波段red
:param red: landsat8的B5波段,近红外波段nir
:return: ndvi 计算结果
"""
ndvi = (nir - red) / (nir + red)
# 去除异常值
ndvi = np.where((ndvi > -1) & (ndvi < 1), ndvi, np.nan)
# 返回最后的NDVI的计算结果
return ndvi
# 获取红外波段
red = data.read(4)
# 获取近红外波段
nir = data.read(5)
ndvi = ndvi(red, nir)
ndvi
array([[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]])
从上面可以看到NDVI的计算结果,但由于显示的原因,看不出来具体的值,所以我们可以抽取其中的部分看下
# 索引从0开始,左闭右开,先行后列
ndvi[200:205, 200:205]
array([[0.54883609, 0.53111163, 0.57332026, 0.57702084, 0.62110553],
[0.33744574, 0.39278512, 0.59430278, 0.53371203, 0.55261845],
[0.21507656, 0.33413002, 0.67471661, 0.58942541, 0.44131185],
[0.27263374, 0.36076244, 0.61868687, 0.60346696, 0.48701299],
[0.27899687, 0.40038591, 0.60087188, 0.65544752, 0.47488706]])
这样看着就比较舒服了
4.4 根据置信度区间获取对应的NDVImin、NDVImax
由于图像中存在的各种噪声,所以NDVI的极值有可能并不是我们想要的NDVI最大最小值,所以在计算植被覆盖度时关于NDVI最大最小值的选择是在给定的置信度区间内选择其最大最小值。
关于置信度的取值,这个说实话我没有深入研究,根据多数论文上面写的通常是取5%和95%的置信度区间,但是我在李苗苗论文中提到的这两个值其实是用土地利用图和土壤图切割后定的,所以,具体怎么取此处如果有大佬了解或者研究可以告知一二,感谢!
我在此处提取置信度对应的NDVI的方法是将计算后的NDVI进行百分比提取,比如将累积百分比在5%的NDVI值或者离5%最近的NDVI值定位NDVImin,同理,将累积百分比在95%的NDVI值或者离95%最近的NDVI值定位NDVImax。
此处使用的主要是numpy的统计函数percentile()和nanpercentile()函数。
percentile(a, q, [options])函数可以沿某轴(axis)方向计算数组a中第q数值的百分比位数,百分比分位数就是位于q%位置处的值,它使得至少有q%的数据项小于或等于这个值,且至少有(100-q)%的数据项大于或等于这个值。具体参数介绍可以自行查找。
nanpercentile()函数格式与percentile()函数格式相同,只是在计算百分位数时会忽略数组中的nan值。显而易见,这里我们需要使用nanpercentile(),除非你确定你的数组里没有nan值。
# 获取最接近5%和95%对应的NDVI值
ndvimin, ndvimax = np.nanpercentile(ndvi, [5, 95], method='nearest')
ndvimin
0.1426209177346011
ndvimax
0.771513353115727
4.5 计算植被覆盖度Fc
F c = N D V I − N D V I s o i l N D V I v e g − N D V I s o i l Fc = \frac {NDVI - NDVIsoil} {NDVIveg - NDVIsoil} Fc=NDVIveg−NDVIsoilNDVI−NDVIsoil
而NDVIveg和NDVIsoil应用中通常根据实际影像中的置信区间取NDVI的最大最小值。
此时根据公式,我们获取到了计算植被覆盖度所需的所有值,包括一个研究区范围内的NDVI数组,置信度区间5%、95%范围内的NDVImin和NDVImax。
# 计算植被覆盖度
fc = (ndvi - ndvimin) / (ndvimax + ndvimin)
fc
array([[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
...,
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan],
[nan, nan, nan, ..., nan, nan, nan]])
# 设置色带
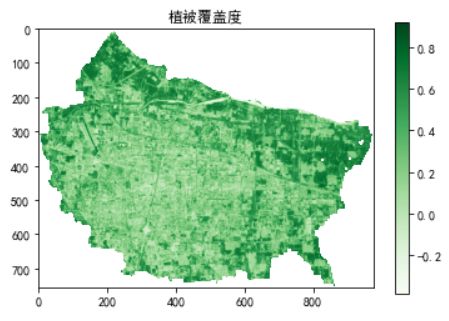
plt.imshow(fc, cmap=plt.cm.Greens)
# 添加色带
plt.colorbar()
# 用来正确显示中文
plt.rcParams['font.sans-serif'] = ['SimHei']
# 用来正确显示负号
plt.rcParams['axes.unicode_minus'] = False
# 添加标题
plt.title('植被覆盖度')
plt.savefig(r'G:\Project\geo\img\植被覆盖度.png', dpi=300)
4.5.1 保存植被覆盖度影像至本地
最后,我们将计算后的结果保存至本地,使用的是上篇文章中写的保存单波段影像的函数save()
def save(outPath, data, meta):
"""
保存单波段影像
:param outPath: 输出影像
:param data: 波段数据
:param meta: 原始影像的元数据
:return:
"""
meta.update({'count': 1,
'dtype': 'float64'
})
with rio.open(outPath, "w", **meta) as dest:
dest.write_band(1, data)
save(r'G:\Project\geo\data\fc.tif', fc, meta)
4.6 ENVI验证
我将计算的NDVI保存到本地,然后用ENVI统计了下直方图,ENVI统计结果如下,当保持默认参数时
累计百分比为5.27889485左右时是
0.14042771
累计百分比为95.58883223左右时是
0.77095871
可以看到,和我统计到的数据0.1426209177346011、0.771513353115727只能保证小数点后两位相同
至于原因,我不知道,哈哈哈哈,如果有知道的朋友可以告知下,感谢!
4.7 完整代码
# 首先导入所需的包
import numpy as np
import rasterio as rio
import matplotlib.pyplot as plt
def ndvi(red, nir):
"""
计算植被指数 ndvi
:param red: landsat8的B4波段,红外波段red
:param red: landsat8的B5波段,近红外波段nir
:return: ndvi 计算结果
"""
ndvi = (nir - red) / (nir + red)
# 去除异常值
ndvi = np.where((ndvi > -1) & (ndvi < 1), ndvi, np.nan)
# 返回最后的NDVI的计算结果
return ndvi
def save(outPath, data, meta):
"""
保存单波段影像
:param outPath: 输出影像
:param data: 波段数据
:param meta: 原始影像的元数据
:return:
"""
meta.update({'count': 1,
'dtype': 'float64'
})
with rio.open(outPath, "w", **meta) as dest:
dest.write_band(1, data)
path = r'G:\Project\geo\data\test_clip.tif'
# 获取影像数据集
data = rio.open(path)
# 影像的元数据
meta = data.meta
# 获取红外波段
red = data.read(4)
# 获取近红外波段
nir = data.read(5)
ndvi = ndvi(red, nir)
# 获取最接近5%和95%对应的NDVI值
ndvimin, ndvimax = np.nanpercentile(ndvi, [5, 95], method='nearest')
# 计算植被覆盖度
fc = (ndvi - ndvimin) / (ndvimax + ndvimin)
# 保存植被覆盖度影像
save(r'G:\Project\geo\data\fc.tif', fc, meta)
# matplotlib画图
# 设置色带
plt.imshow(fc, cmap=plt.cm.Greens)
# 添加色带
plt.colorbar()
# 用来正确显示中文
plt.rcParams['font.sans-serif'] = ['SimHei']
# 用来正确显示负号
plt.rcParams['axes.unicode_minus'] = False
# 添加标题
plt.title('植被覆盖度')
# 保存绘图☞本地
plt.savefig(r'G:\Project\geo\img\植被覆盖度.png', dpi=300)
5 总结
对于鸽王来说,emmmmmm,其实是不会更新那么勤的,但是这几天陆续新增了很多关注,所以,反向“cpu”来了,总想着倒逼自己去思考学习然后输出些什么,但我本身脑子里存储的东西真的是太"贫瘠"了,最近经常刷到董宇辉,真的很佩服他的知识储备量,每次采访都很思路清晰,就像整本书就在他的脑海中,语言如溪流,娓娓道来,延绵不绝!
周四周五的时候本来在看TVDI,结果突然想到了这个问题,想着快两周了,也该更新了,趁着摸鱼的时候思考了下,中间也请教了些研究生态遥感的朋友,非常感谢!今天早上九点多起来理了下思路开始写,中间边写边搜索,也学到了很多,比如百分比那个函数,本身我想的很麻烦,还得计算直方图什么的,后面无意间看到了numpy的统计函数,其中提到了percentile,也不用考虑直方图时掩膜Nan值的问题了,直接省时省事!
本来没想写那么多,也没那么多思路,结果水着水着字数就多了,中午饭也没做,但一点都不饿,写计算过程不难,因为只是些波段计算,难的是把自己想的梳理写出来,太花时间了,但好处是记忆加深一些,如果去年毕业的我,估计连第一部分都写不出来。快四点了,哈哈,果然是话痨。
不过,今天看到了两句话,第一句我放在了开头,第二句放在这吧
“趁着年轻,好好犯病”
不定时更新,如有帮助,可以点赞!
公众号:壹贰叁言
本文由mdnice多平台发布