CCPlotR | 轻松拿捏单细胞分析之细胞交互!~
1写在前面
周末了各位,昨天去看了奥本海默,不得不说,大神就是大神。
比起我们的电影,似乎诺兰更好地还原了奥本海默的真实。
言归正传,今天分享的是CCPlotR包,用于基于scRNAseq数据推断细胞间相互作用的可视化。
2用到的包
rm(list = ls())
#devtools::install_github("Sarah145/CCPlotR")
library(tidyverse)
library(CCPlotR)
3示例数据
我们先把示例数据导进来吧,一共有2个文件哦。
data(toy_data, toy_exp, package = 'CCPlotR')
3.1 数据一
先查看一下数据的结构呢。
DT::datatable(toy_data)
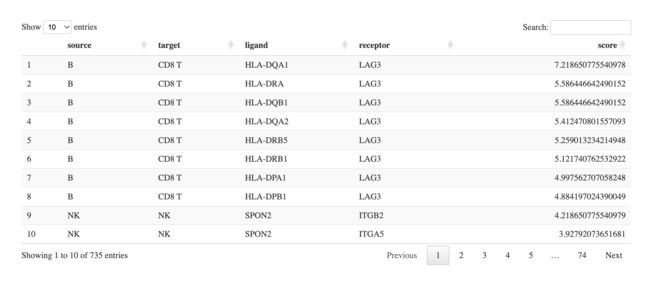
3.2 数据二
接着是另外一个data。
DT::datatable(toy_exp)
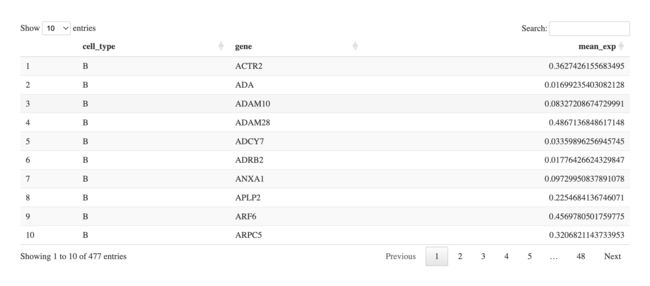
4heatmap可视化
4.1 可视化一
cc_heatmap(toy_data)
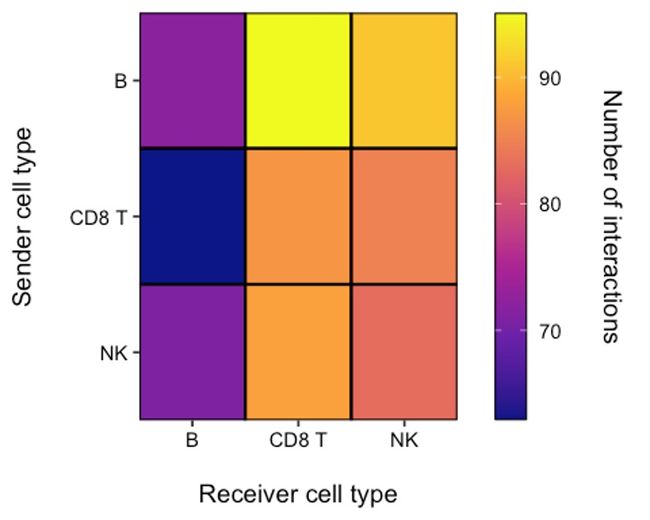
4.2 可视化二
展示top10。
cc_heatmap(toy_data, option = 'B', n_top_ints = 10)

4.3 可视化三
cc_heatmap(toy_data, option = 'CellPhoneDB')

4.4 可视化四
cc_heatmap(toy_data, option = 'Liana')
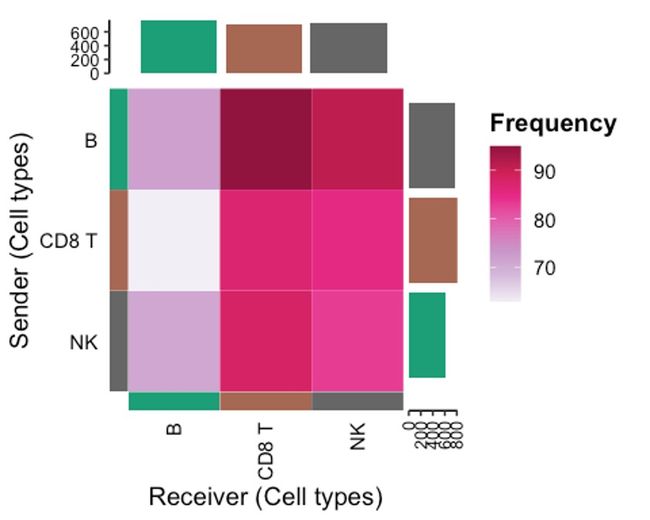
5doplot可视化
5.1 可视化一
cc_dotplot(toy_data)

5.2 可视化二
展示top15。
cc_dotplot(toy_data, option = 'B', n_top_ints = 15)

5.3 可视化三
cc_dotplot(toy_data, option = 'CellPhoneDB', n_top_ints = 15)
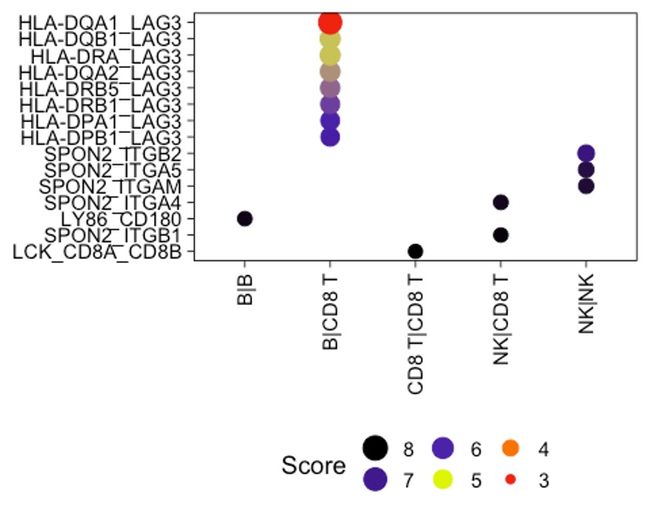
5.4 可视化四
cc_dotplot(toy_data, option = 'Liana', n_top_ints = 15)
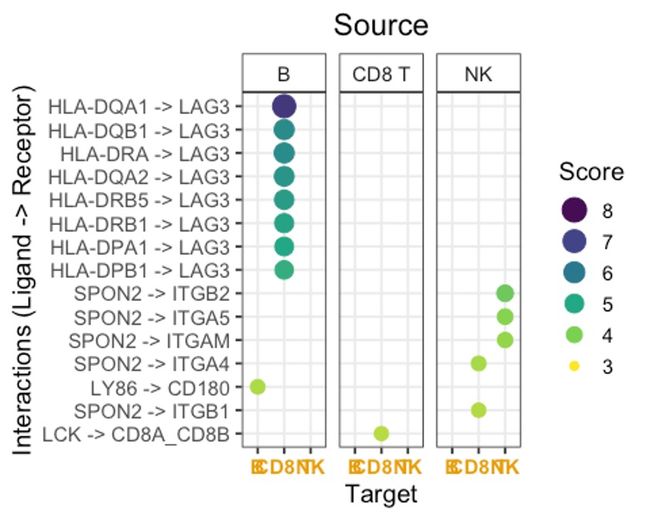
6network可视化
6.1 可视化一
cc_network(toy_data)

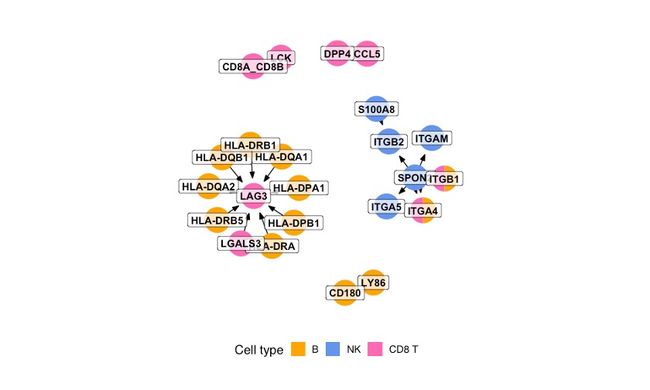
6.2 可视化二
cc_network(toy_data, colours = c('orange', 'cornflowerblue', 'hotpink'), option = 'B')
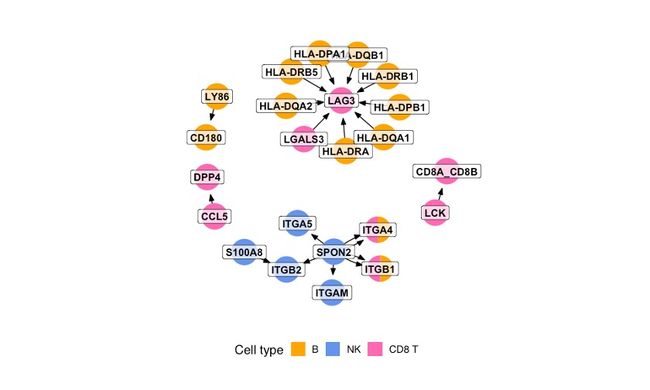
6.2 可视化三
cc_network(toy_data, colours = c('orange', 'cornflowerblue', 'hotpink'),
option = 'B',
layout = "nicely"
)
7circos可视化
7.1 可视化一
cc_circos(toy_data)

7.2 可视化二
cc_circos(toy_data, option = 'B', n_top_ints = 10)

7.3 可视化三
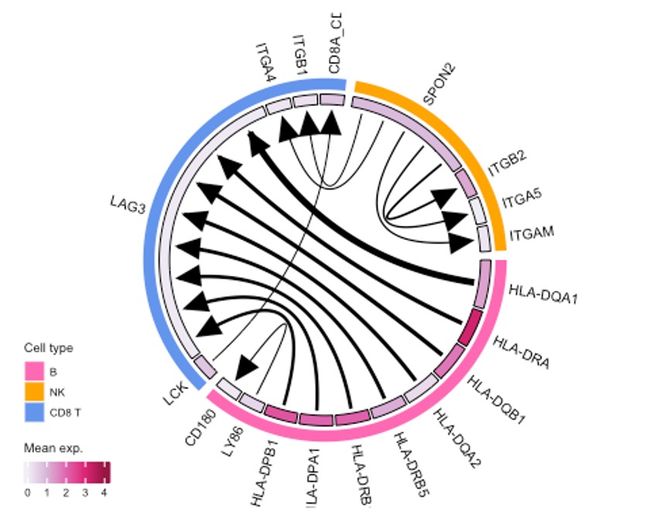
cc_circos(toy_data, option = 'C', n_top_ints = 15,
exp_df = toy_exp,
cell_cols = c(`B` = 'hotpink', `NK` = 'orange', `CD8 T` = 'cornflowerblue'),
palette = 'PuRd',
cex = 0.5)
8arrow可视化
8.1 可视化一
cc_arrow(toy_data, cell_types = c('B', 'CD8 T'), colours = c(`B` = 'hotpink', `CD8 T` = 'orange'))

8.2 可视化二
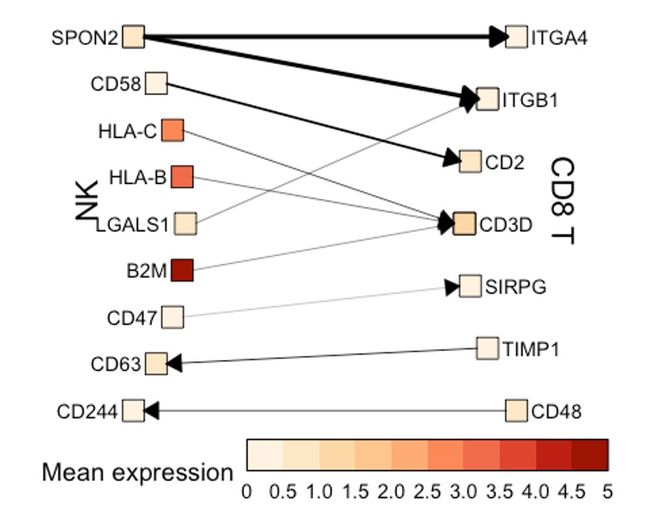
cc_arrow(toy_data, cell_types = c('NK', 'CD8 T'), option = 'B', exp_df = toy_exp, n_top_ints = 10, palette = 'OrRd')
9sigmoid可视化
cc_sigmoid(toy_data)

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
LASSO | 不来看看怎么美化你的LASSO结果吗!?
chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~
WGCNA | 值得你深入学习的生信分析方法!~
ComplexHeatmap | 颜狗写的高颜值热图代码!
ComplexHeatmap | 你的热图注释还挤在一起看不清吗!?
Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
scRNA-seq | 吐血整理的单细胞入门教程
NetworkD3 | 让我们一起画个动态的桑基图吧~
RColorBrewer | 再多的配色也能轻松搞定!~
rms | 批量完成你的线性回归
CMplot | 完美复刻Nature上的曼哈顿图
Network | 高颜值动态网络可视化工具
boxjitter | 完美复刻Nature上的高颜值统计图
linkET | 完美解决ggcor安装失败方案(附教程)
......



本文由 mdnice 多平台发布