遗传算法求解旅行商问题(含python源代码)
目录
前言
编码初始化种群
计算适应度
选择
交叉
变异
完整代码
总结
前言
这次的算法有一点不能确定是否正确,希望有大佬能够批评指正。
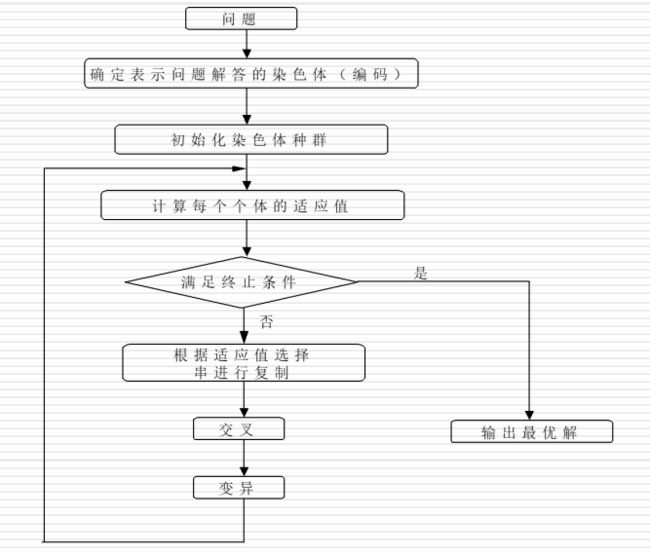
遗传算法的一般步骤
编码初始化种群
种群(population)指同一时间生活在一定自然区域内,同种生物的所有个体。
所以种群是由个体组成的,所以先需要得到个体,然后再随机产生一定数目的个体。
在本算法中个体采用的是实数编码(表示城市的编号,对应列表的下标,按照顺序就是在城市之间的移动路线)。
先对城市的位置进行初始化,采用的是用列表来表示城市的坐标,可以单个定义,也可以随机生成。
先生成一串有序的数字用来表示城市的编号,再每次随机进行打乱后储存到pop列表中,pop表示种群,里面装着的是列表用来表示个体。(个体表示城市的编号,种群表示编号打乱后所有个体的集合)
群体规模太小,不能提供足够的采样点,以致算法性能很差,易陷入局部最优解。
群体规模太大,尽管可以增加优化信息,阻止早熟收敛的发生,但无疑会增加计算量,造成收敛时间太长,表现为收敛速度缓慢。
City_Map = 100 * np.random.rand(20, 2) # 随机产生20个城市(20行2列,数值乘以100)
DNA_SIZE = len(City_Map) # 编码长度(返回行的个数)
POP_SIZE = 100 # 种群大小
# 生成初代种群pop
pop = []
list = list(range(DNA_SIZE)) # 生成[0,DNA_SIZE)的列表
for i in range(POP_SIZE): # POP_SIZE是指种群大小,在程序中是一个固定的值(打乱POP_SIZE次之后把结果储存到pop列表中
random.shuffle(list) # 随机打乱list,进行初始化操作
l = list.copy() # 把list中的数据拷贝到l中
pop.append(l) # 将l添加到pop列表中计算适应度
适应度函数值只能是正值,越大越好。
DNA表示个体,根据个体的值(表示城市的编号)来计算距离。
旅行商问题要求距离越短越好,所以距离越大越不满足要求,故而可以通过对距离求倒数来表示适应度。
在最后减去适应度最小的值,可以保证适应度都为正值。(如果有负数,减去一个更小的负数,会变成正值)
适应度表示的是个体对种群的适应程度,distance函数返回的是按照pop[i]路线计算出来的路径距离大小,故而getfitness函数返回的列表表示的是每一个个体pop[i]通过distance函数计算出来距离的倒数归一化(减去最小的距离的倒数)之后的结果。
distance求出来的距离不是两个城市之间的距离,而是某条路线经过城市的距离之和(是所有的距离)。
def distance(DNA): # 根据DNA的路线计算距离
dis = 0
temp = City_Map[DNA[0]]
for i in DNA[1:]:
# sqrt(pow(x-x0,2)+pow(y-y0,2))
dis = dis + ((City_Map[i][0] - temp[0]) ** 2 + (City_Map[i][1] - temp[1]) ** 2) ** 0.5
temp = City_Map[i]
return dis + ((temp[0] - City_Map[DNA[0]][0]) ** 2 + (temp[1] - City_Map[DNA[0]][1]) ** 2) ** 0.5
def getfitness(pop): # 计算种群适应度,这里适应度用距离的倒数表示
temp = []
for i in range(len(pop)):
temp.append(1 / (distance(pop[i])))
# 减去最小值是为了防止适应度出现负值
return temp - np.min(temp)选择
选择操作也称为复制( reproduction) 操作:从当前群体中按照一定概率选出优良的个体, 使它们有机会作为父代繁殖下一代子孙。
判断个体优良与否的准则是各个个体的适应度值:个体适应度越高, 其被选择的机会就越多。
在程序中个体的选择方法采用的是轮盘赌的方法:
按个体的选择概率产生一个轮盘,轮盘每个区的角度与个体的选择概率成比例。
产生一个随机数, 它落入转盘的哪个区域就选择相应的个体交叉。
适应度的小的个体也有可能被选中。
def select(pop, fitness): # 根据适应度选择,以赌轮盘的形式,适应度越大的个体被选中的概率越大
# print(fitness)
s = fitness.sum()
# np.random.choice(a,size,replace,p=None)随机抽取样本a,表示范围,replace=True被抽中后仍有机会被再次抽中,p没抽中的概率
temp = np.random.choice(np.arange(len(pop)), size=POP_SIZE, replace=True, p=(fitness / s))
p = []
for i in temp:
p.append(pop[i])
return p交叉
程序中交叉采用部分匹配交叉,如果直接采用两点交叉会导致一个个体中出现两个重复的城市,显然是和题目要求是不符合的。
部分匹配交叉保证了每个染色体中的基因仅出现一次,通过该交叉策略在一个染色体中不会出现重复的基因,所以部分匹配交叉经常用于旅行商(TSP)或其他排序问题编码。部分匹配交叉类似于两点交叉,通过随机选择两个交叉点确定交叉区域。执行交叉后一般会得到两个无效的染色体,个别基因会出现重复的情况,为了修复染色体,可以在交叉区域内建立每个染色体的匹配关系,然后在交叉区域外对重复基因应用此匹配关系就可以消除冲突。
交叉概率太大时,种群中个体更新很快,会造成高适应度值的个体很快被破坏掉;
概率太小时,交叉操作很少进行,从而会使搜索停滞不前,造成算法的不收敛。
def crossmuta(pop, CROSS_RATE): # 交叉变异
new_pop = []
for i in range(len(pop)): # 遍历种群中的每一个个体,将该个体作为父代
n = np.random.rand()
if n >= CROSS_RATE: # 大于交叉概率时不发生变异,该子代直接进入下一代
temp = pop[i].copy()
new_pop.append(temp) # 直接进行拷贝
if n < CROSS_RATE: # 小于交叉概率时发生变异
list1 = pop[i].copy()
list2 = pop[np.random.randint(POP_SIZE)].copy() # 选取种群中另一个个体进行交叉(随机选择)
status = True
while status: # 产生2个不相等的节点,中间部分作为交叉段,采用部分匹配交叉(直到k1变异
互换变异:随机选取染色体的两个基因进行简单互换。
采用随机的形式,在[0,DNA_SIZE)的范围内生成两个下标(确保两个位置不一致),在将两个位置上面的值进行交换。
变异概率太小则很难产生新模式,变异概率太大则会使遗传算法成为随机搜索算法。
def mutation(DNA, MUTA_RATE): # 进行变异
# 两点变异
if np.random.rand() < MUTA_RATE: # 以MUTA_RATE的概率进行变异
mutate_point1 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置
mutate_point2 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置
while (mutate_point1 == mutate_point2): # 保证2个所选位置不相等
mutate_point2 = np.random.randint(0, DNA_SIZE) #如果相等将mutate_point2重新进行随机生成位置
DNA[mutate_point1], DNA[mutate_point2] = DNA[mutate_point2], DNA[mutate_point1] # 2个所选位置进行互换
逆转变异:在个体码串中随机选择两点( 逆转点) ,然后将两点之间的基因值以逆向排序插入到原位置中。
随机生成两个下标,如x1,x2(确保x1 插入变异:在个体码串中随机选择一个码, 然后将此码插入随机选择的插入点中间。 遗传算法入门详解 - 知乎 (zhihu.com) 这个最好适应度,最差适应度以及平均适应度的概念没完全掌握,不知道是不是这一个意思,所以在程序中注释了,大家可以根据自己的理解来添加。 我理解到的: 最好适应度就是每一次的迭代都会有一个适应度最高的个体,把每一代适应度最高的个体进行比较选出最大的那个,得到的就是整体适应度最好的。 最差适应度就是0,因为每一次都进行了归一化操作。 平均适应度就是保存每一代的适应度以及个体的数量,最后进行求平均值。 运行时间就是在算法运行前记录当前系统的时间,算法运行后记录当前系统的时间,所形成的差值(运行后-运行前)就是程序运行的时间。 适应度因为采用的是距离的倒数,加上还进行了归一化的处理,随机生成城市的的坐标*100,导致两点之间的距离较大,如果想得到较大一点的适应度,可以缩小城市之间的距离。 City_Map = 100 * np.random.rand(20, 2) 改 City_Map = np.random.rand(20, 2) 遗传算法的思路是“适者生存,优胜劣汰”,模拟生物的进化,以一个初始生物群体为起点,经过竞争后,一部分个体被淘汰而无法再进入这个循环圈,而另一部分则胜出成为种群。对于算法的选择的个体,适应度高的并不一定进入种群,只是进入种群的可能性比较大;而适应度低的个体并不一定被淘汰,只是进入种群的可能性比较小,模拟生物进化的过程。def mutation(DNA, MUTA_RATE): # 进行变异
# 逆转变异
if np.random.rand() < MUTA_RATE: # 以MUTA_RATE的概率进行变异
status = True
while status: # 产生2个不相等的节点,中间部分作为交叉段,采用部分匹配交叉(直到mutate_point1
随机生成两个实数,这次不是交换,是插入的方式,也就是插入点之后的元素的位置都会发生改变。def mutation(DNA, MUTA_RATE): # 进行变异
#插入变异
if np.random.rand() < MUTA_RATE: # 以MUTA_RATE的概率进行变异
mutate_point1 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置(选中一个基因)
mutate_point2 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置(插入点)
while (mutate_point1 == mutate_point2): # 保证2个所选位置不相等
mutate_point2 = np.random.randint(0, DNA_SIZE)
temp=DNA[mutate_point1]#先保存mutate_point1对应的值
del DNA[mutate_point1]#删除mutate_point1对应的值
DNA.insert(mutate_point2,temp)#重新插入到列表中完整代码
import time
import numpy as np
import random
import matplotlib.pyplot as plt
# 各个城市的坐标
City_Map = 100 * np.random.rand(10, 2) # 随机产生10个城市(10行2列,数值乘以100)
DNA_SIZE = len(City_Map) # 编码长度(返回行的个数)
POP_SIZE = 100 # 种群大小
CROSS_RATE = 0.85 # 交叉率
MUTA_RATE = 0.15 # 变异率
Iterations = 500 # 迭代次数
def distance(DNA): # 根据DNA的路线计算距离
dis = 0
temp = City_Map[DNA[0]]
for i in DNA[1:]:
# sqrt(pow(x-x0,2)+pow(y-y0,2))
dis = dis + ((City_Map[i][0] - temp[0]) ** 2 + (City_Map[i][1] - temp[1]) ** 2) ** 0.5
temp = City_Map[i]
return dis + ((temp[0] - City_Map[DNA[0]][0]) ** 2 + (temp[1] - City_Map[DNA[0]][1]) ** 2) ** 0.5
def getfitness(pop): # 计算种群适应度,这里适应度用距离的倒数表示
temp = []
for i in range(len(pop)):
temp.append(1 / (distance(pop[i])))
# 减去最小值是为了防止适应度出现负值
return temp - np.min(temp)
def select(pop, fitness): # 根据适应度选择,以赌轮盘的形式,适应度越大的个体被选中的概率越大
# print(fitness)
s = fitness.sum()
# np.random.choice(a,size,replace,p=None)随机抽取样本a,表示范围,replace=True被抽中后仍有机会被再次抽中,p没抽中的概率
temp = np.random.choice(np.arange(len(pop)), size=POP_SIZE, replace=True, p=(fitness / s))
p = []
for i in temp:
p.append(pop[i])
return p
def mutation(DNA, MUTA_RATE): # 进行变异
# 两点变异
if np.random.rand() < MUTA_RATE: # 以MUTA_RATE的概率进行变异
mutate_point1 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置
mutate_point2 = np.random.randint(0, DNA_SIZE) # 随机产生一个实数,代表要变异基因的位置
while (mutate_point1 == mutate_point2): # 保证2个所选位置不相等
mutate_point2 = np.random.randint(0, DNA_SIZE) #如果相等将mutate_point2重新进行随机生成位置
DNA[mutate_point1], DNA[mutate_point2] = DNA[mutate_point2], DNA[mutate_point1] # 2个所选位置进行互换
def crossmuta(pop, CROSS_RATE): # 交叉变异
new_pop = []
for i in range(len(pop)): # 遍历种群中的每一个个体,将该个体作为父代
n = np.random.rand()
if n >= CROSS_RATE: # 大于交叉概率时不发生变异,该子代直接进入下一代
temp = pop[i].copy()
new_pop.append(temp) # 直接进行拷贝
if n < CROSS_RATE: # 小于交叉概率时发生变异
list1 = pop[i].copy()
list2 = pop[np.random.randint(POP_SIZE)].copy() # 选取种群中另一个个体进行交叉(随机选择)
status = True
while status: # 产生2个不相等的节点,中间部分作为交叉段,采用部分匹配交叉(直到k1参考信息
![]() https://zhuanlan.zhihu.com/p/100337680遗传算法python进阶理解+论文复现(纯干货,附前人总结引路)_python神经网络遗传算法_不想秃头的夜猫子的博客-CSDN博客
https://zhuanlan.zhihu.com/p/100337680遗传算法python进阶理解+论文复现(纯干货,附前人总结引路)_python神经网络遗传算法_不想秃头的夜猫子的博客-CSDN博客![]() https://blog.csdn.net/golden_knife/article/details/128510731通俗易懂地解释遗传算法 - 知乎 (zhihu.com)
https://blog.csdn.net/golden_knife/article/details/128510731通俗易懂地解释遗传算法 - 知乎 (zhihu.com)![]() https://zhuanlan.zhihu.com/p/136393730遗传算法解决旅行商问题(详细解释+代码分享) - 知乎 (zhihu.com)
https://zhuanlan.zhihu.com/p/136393730遗传算法解决旅行商问题(详细解释+代码分享) - 知乎 (zhihu.com)![]() https://zhuanlan.zhihu.com/p/344588977用遗传算法求解旅行商问题_中国旅行商问题,34个省会-CSDN博客
https://zhuanlan.zhihu.com/p/344588977用遗传算法求解旅行商问题_中国旅行商问题,34个省会-CSDN博客![]() https://blog.csdn.net/breeze_blows/article/details/102992997遗传算法(三)——适应度与选择_遗传算法适应度函数-CSDN博客
https://blog.csdn.net/breeze_blows/article/details/102992997遗传算法(三)——适应度与选择_遗传算法适应度函数-CSDN博客![]() https://blog.csdn.net/weixin_30239361/article/details/101540896
https://blog.csdn.net/weixin_30239361/article/details/101540896总结