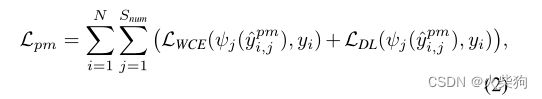- 194、Django Channels实战:构建实时WebSocket应用
多多的编程笔记
djangowebsocketsqlite
DjangoChannels:实现WebSocket与实时通信本文将向您介绍Python开发框架Django中的一个重要组件——DjangoChannels,它使得在Django中实现WebSocket通信变得轻而易举。通过阅读本文,您将了解WebSocket的概念、DjangoChannels的工作原理以及如何在实际项目中使用它来实现实时通信。1.WebSocket:实现快速双向通信在介绍Dja
- apache-dolphinscheduler-3.2.0调度器简介和集群部署详细安装文档
1、为什么选用apache-dolphinscheduler轻松管理复杂的任务工程支持跨项目和跨工作流程的任务依赖支持Kill、暂停和恢复操作任务支持以租户、Worker分组组和环境中隔离运行每个任务都可以修改输出参数,并将其传递给后续任务在一分钟内创建你的工作流程通过拖拉拽的工作流创建方式提高效率支持Python、Yaml和OpenApi的方式生成工作流支持将一个工作流作为另一个工作流的子流程执
- 《卷积神经网络到Vision Transformer:计算机视觉的十年架构革命》
HeartException
人工智能学习
前言前些天发现了一个巨牛的人工智能免费学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家。点击跳转到网站题目《卷积神经网络到VisionTransformer:计算机视觉的十年架构革命》展开深度解析,全文采用技术演进史+架构对比+产业影响的三段式结构,附关键数据与趋势预测:卷积神经网络到VisionTransformer:计算机视觉的十年架构革命副标题:从局部感知到全局建模,一场改变AI视觉基石的
- 基于python版本secsgem源码开发gem,该gem作为一个中间平台,既要连接EAP,又要连接探针台,应该如何设置devicetype、connectmode
SunkingYang
#SECS协议pythongemeap中间平台三方连接设备类型
文章目录一、角色定义与连接模式设计1.中间平台的双向角色2.核心参数设置二、代码实现步骤1.创建两个独立连接实例2.数据转发逻辑实现三、高级配置与注意事项1.状态机管理2.多线程与异步处理3.协议兼容性4.调试与错误排查四、典型应用场景1.配方管理2.事件与报警处理五、扩展方案(多设备协同)总结基于Python的secsgem库开发中间平台需同时连接EAP(作为Host端)和探针台(作为Equip
- Python 爬虫入门(九):Scrapy安装及使用「详细介绍」
blues_C
Python爬虫实战python爬虫scrapy
Python爬虫入门(九):Scrapy安装及使用「详细介绍」前言1.Scrapy简介2.Scrapy的安装2.1环境准备2.2安装Scrapy3.创建Scrapy项目3.1创建项目3.2项目结构简介4.编写爬虫4.1创建爬虫4.2解析数据4.3运行爬虫5.存储数据5.1存储为JSON文件5.2存储到数据库5.2.1MongoDB6.处理请求和响应6.1请求头设置6.2处理响应7.高级功能7.1使
- python 会议室预约系统解决方案_会议预约管理系统解决方案
weixin_39963853
python会议室预约系统解决方案
随着时代快速发展,各大会议中心都有密集的会议安排,同时企业也会有大大小小的会议安排。会议室没有管理,会造成会议室被争抢、重要会议被非重要会议挤占、会议室信息不能及时发布、会议室设备开会时不满足会议要求、会议室设备没有提前调试以及设备因没有及时检修造成故障、会中设备出现故障且没有应急预案等等问题。这些问题往往频繁发生,使人们平时习以为常,用时抱怨连连。如果任由问题发生、日积月累,则会产生严重影响,轻
- python 会议室预约系统解决方案_智能会议预约系统解决方案
weixin_39914868
python会议室预约系统解决方案
随着时代的发展,科技的进步,效率在工作中成为了评价工作能力的一项标准。人的工作效率,机器的工作效率等等,我们都是逐步在提高。达到高效的工作状态离不开智能的解决方案,在此我将分享上海铭港公司做的智能办公空间—会议预约系统的案例。上海铭港做的此项会议预约系统案例的解决方案围绕的主题中心即是:效率提到会议室,可能令大家的头疼的事情随之而来。特别是公司的行政部门人员。哪间会议室是在空闲状态?哪间会议室有人
- python 会议室预约系统解决方案_会议室预约管理系统方案书.pdf
会议预约管理系统系统方案目录一、系统概述31.1什么是会议预约管理31.2会议管理趋势4二、系统功能52.1功能特点52.2系统优势62.3系统界面7三、系统部署103.1安装示意图103.2安装实景图11四、会议显示12五、应用行业135.1教育行业135.2法律行业135.3医疗保健135.4企业13一、系统概述为符合现代信息化的考虑,在办公楼的底楼进出大厅设置一套多媒体信息显示公告系统,用于
- python 会议室预约系统解决方案_会议预约系统_智能会议预约管理系统_轻松实现会议管理解决方案...
weixin_39779032
python会议室预约系统解决方案
随着社会的发展,会议预约管理系统在近年来呈现高速发展,但是各种等级层次不齐。现代办公会议室是组织的公共资源,会议室及其附属的设备构成召开会议的基础环境。广州朗歌公司以提高会议效率为焦点,以会议全过程管理为理念,开发了会议预定管理系统产品。会议室预约管理系统的目标是为会议的准备提供便捷的服务,实时动态的将会议室预定信息,传递到每个会议室门口、大厅及楼梯口等公共通道的显示屏上,实现会议信息发布引导功能
- GitHub每周最火火火项目(6.30-7.6)
FutureUniant
Github周推githubmicrosoft人工智能ai计算机视觉
1.NanniCoder/MediaCrawler项目名称:MediaCrawler项目介绍:该项目采用Python语言开发,专注于多平台媒体内容的爬取工作。从用途来看,它能够精准抓取小红书笔记、抖音视频、快手视频、B站视频、微博帖子、百度贴吧帖子及评论、知乎问答文章等各类媒体平台的内容。在使用场景方面,对于新媒体运营者而言,可借助它批量采集行业内多平台的热门内容、竞品动态,为自身内容创作、运营策
- python项目使用poetry管理依赖项
zQIANYUN
python开发python开发语言
1.poetry管理依赖项Poetry是Python项目中用于依赖管理和项目打包的工具。相比传统的pip和requirements.txt,Poetry提供了更加现代和集成的解决方案。优点:Poetry能够自动处理依赖关系冲突,帮助开发者避免版本不兼容问题。在安装新的依赖时,它会检查现有依赖,并更新pyproject.toml文件和生成锁定的poetry.lock文件,以确保项目使用的依赖版本在团
- Seaborn 教程
froginwe11
开发语言
Seaborn教程引言Seaborn是一个强大的Python数据可视化库,它建立在Matplotlib的基础上,专门用于统计图形的绘制。Seaborn提供了一系列的绘图功能,使得统计数据的可视化变得更加简单和直观。本文将为您提供一个全面的Seaborn教程,帮助您快速掌握其基本用法和高级技巧。安装与导入在开始之前,请确保您的Python环境中已经安装了Seaborn和Matplotlib。您可以使
- python中的logger包的详细使用教程
SunkingYang
#python入门之日志使用python日志logger使用方法说明
文章目录功能说明一、Logger的创建与基础配置二、Handler的配置与使用三、Formatter自定义日志格式四、记录不同级别的日志五、高级配置与最佳实践六、常见问题与调试使用方法一、基础配置与快速使用二、自定义Logger对象三、高级用法四、最佳实践与注意事项五、实际应用场景示例Python的logging模块是标准库中用于记录日志的核心工具,通过灵活配置可实现多级别、多目标、多格式的日志管
- 10分钟掌握Python缓存
项目背景代码检查项目,需要存储每一步检查的中间结果,最终把结果汇总并写入文件中在中间结果的存储中可以使用context进行上下文的传递,但是整体对代码改动比较大,违背了开闭原则也可以利用缓存存储,处理完成之后再统一读缓存并写入文件在权衡了不同方案后,我决定采用缓存来存储中间结果。接下来,我将探讨Python中可用缓存组件。python缓存分类决定选择缓存,那么python中都有哪些类型的缓存呢?1
- Go与Python爬虫对比及模板实现
go语言和Python语言都可选作用来爬虫项目,因为python经过十几年的累积,各种库是应有尽有,学习也相对比较简单,相比GO起步较晚还是有很大优势的,么有对比就没有伤害,所以我利用一个下午,写个Go爬虫,虽说运行起来没啥问题,但是之间出错的概率太高了,没有完备的模版套用得走很多弯路,这就是为啥go没有python受欢迎的原因。为何Go爬虫远没有Python爬虫流行?1、历史生态差距Python
- pip设置国内源:阿里云、腾讯云、清华大学源
[email protected]
Pythonpippython
更换Python的pip源(尤其是默认源访问速度较慢时)是一个常见需求,可以显著提升安装Python包的速度。以下是如何为pip设置国内源的步骤,以阿里云、腾讯云、清华大学源为例:1.备份原有源配置(可选但推荐)在进行任何更改之前,备份现有的pip配置文件是一个好习惯。这样,如果遇到问题,你可以轻松恢复到初始状态。Bash1mkdir~/.pip_backup2cp~/.pip/pip.conf~
- 前端如何借助 Postman 进行接口性能调优
前端视界
前端艺匠馆前端postmanluaai
前端如何借助Postman进行接口性能调优关键词:前端开发、Postman、接口性能调优、API测试、性能分析摘要:本文围绕前端开发中借助Postman进行接口性能调优展开。首先介绍了相关背景知识,包括目的、预期读者、文档结构和术语表。接着阐述了核心概念,如接口性能的相关概念及其联系,并给出了对应的文本示意图和Mermaid流程图。详细讲解了核心算法原理和具体操作步骤,结合Python代码示例进行
- 数据分析全流程:从收集到可视化的高效实战
晨曦543210
python
1.数据收集来源:数据库、API、传感器、日志文件、社交媒体、问卷调查等。工具:Python(requests、Scrapy)、SQL、Excel、Kafka(实时流数据)。2.数据清洗处理缺失、重复、错误或不一致的数据:缺失值:删除、填充(均值/中位数/众数)、插值或预测。异常值:使用箱线图、Z-score或IQR方法检测并处理。格式标准化:统一日期、单位、文本格式(如大小写、去除空格)。去重:
- R语言初学者爬虫简单模板
q56731523
r语言爬虫开发语言iphone
习惯使用python做爬虫的,反过来使用R语言可能有点不太习惯,正常来说R语言好不好学完全取决于你的学习背景以及任务复杂情况。对于入门学者来说,R语言使用rvest+httr组合,几行代码就能完成简单爬取(比Python的Scrapy简单得多),R语言数据处理优势明显,爬取后可直接用dplyr/tidyr清洗,小打小闹用R语言完全没问题,如果是企业级大型项目还是有限考虑python,综合成本还是p
- 目标检测:从基础原理到前沿技术全面解析
随机森林404
计算机视觉目标检测人工智能计算机视觉
引言在计算机视觉领域,目标检测是一项核心且极具挑战性的任务,它不仅要识别图像中有什么物体,还要确定这些物体在图像中的具体位置。随着人工智能技术的快速发展,目标检测已成为智能监控、自动驾驶、医疗影像分析等众多应用的基础技术。本文将全面介绍目标检测的基础概念、发展历程、关键技术、实践应用以及未来趋势,为读者提供系统性的知识框架。第一章目标检测概述1.1目标检测的定义与重要性目标检测(ObjectDet
- 邻近巷道爆破振动模拟与可视化:计算力学的工程应用
碳酸的唐
动态规划数学建模
引言隧道爆破施工是现代工程建设中常用的方法,但爆破产生的振动会对周围结构和地质环境产生影响。本文介绍一个基于Python的邻近巷道爆破振动模拟系统,该系统通过数值计算模拟爆破引起的应力波传播过程,并提供多种可视化方式展示振动效应。本研究对于理解爆破振动机理、评估爆破安全距离以及优化爆破参数具有重要意义。理论基础爆破应力波传播模型爆破引起的应力波在岩体中的传播可通过弹性波动理论描述。在均匀介质中,应
- 手把手教你编写Python抢购脚本_抢单脚本的制作教程
网络安全小宇哥
python开发语言测试工具学习web安全网络安全安全架构
想买苹果手机,但总是抢不到,所以想试着能不能写个脚本代码。第一步:把想要抢购的商品加进购物车,注意:脚本是对购物车内全部商品进行下单操作,所以不够买的商品最好先从购物车内删除。第二步:写好Python脚本,在抢购之前运行,并设置好抢购时间。Python脚本实现安装Python。我安装的是anaconda安装webdriver扩展。它是Selenium模块的一部分。Selenium是一个用于Web应
- 【Python】类(class)的创建
Herbert_JL
pythonpythonlinux
1类简介1.1什么是类在面向对象编程(OOP)中,类(Class)是一种封装了数据和操作这些数据的函数的编程结构。它是一种抽象的概念,用于定义具有相同属性(变量)和方法(函数)的对象的模板。类可以看作是一个“蓝图”,用于创建具有相同特征和行为的对象实例。1.2类的作用1.2.1封装(Encapsulation)类将数据(属性)和操作数据的方法封装在一起,形成一个独立的单元。这样可以隐藏内部实现细节
- 【Python】类的继承、重载与多态
类的继承(Inheritance)类的继承是面向对象编程(OOP)中的一个重要概念,它允许一个类(称为子类或派生类)继承另一个类(称为父类或基类)的属性和方法。继承可以提高代码的复用性,减少重复代码,并且能够构建出层次化的类结构。继承的基本概念父类(基类):被继承的类,提供了可以被继承的属性和方法。子类(派生类):继承父类的类,可以使用父类的属性和方法,并且还可以添加新的属性和方法,或者覆盖父类的
- 【Python】For
Herbert_JL
pythonpython开发语言
For基本语法forelementiniterable:statement(s)element:是循环变量,用于存储可迭代对象中当前遍历到的元素。iterable:是需要遍历的可迭代对象,如列表、元组、字典等。for遍历列表fruits=["apple","banana","cherry"]forfruitinfruits:print(fruit)applebananacherryfor遍历字符串
- 【python 进阶】argparse模块
Herbert_JL
pythonpythonjavalinux
argparse模块Python的argparse模块用于解析命令行参数,使得脚本能够灵活地接受用户从命令行传入的各种参数,从而根据不同的参数配置来执行不同的操作。ArgumentParser类argparse.ArgumentParser是Python中argparse模块的核心类,用于创建一个解析器对象,该对象能够读取和解析命令行参数和选项,将它们转换为相应的数据类型,并提供给程序使用。功能常
- 在python中function啥类型_Python中function和method
这两个概念已经有很多人解释过了,从本文的『参考』中就可以看出来。之所以还要写一篇这个主题,主要是为了用自己的语言表述一下,并且尽可能的讲的清楚一点。泛泛地说,function是一般意义上的函数,即对一段代码的封装,并由一个地址(函数名)来调用。method通常是面向对象的概念,即method是属于一个类或类的对象的。method是与类或类的对象相关的函数。下面讲一下我对这两个概念的更具体的理解。如
- 【pytorch】——Could not export Python function call ‘Scatter‘
pytorch用pytorch的trace导出模型的时候,报错errorRuntimeError:CouldnotexportPythonfunctioncall'Scatter'.RemovecallstoPythonfunctionsbeforeexport.Didyouforgettoadd@scriptor@script_methodannotation?Ifthisisann.Modul
- Python中如何将非内置数据类型导出成Json格式
前言:在Python开发中往往使用自定义的数据类型来封装属性特定对象。但是这些自定义的数据类型在导出成Json文件时往往会报出不支持的错误。本篇文章将介绍如何解决这个问题。操作步骤:1、创建自定义数据类型:fromdataclassesimportdataclassimportjson@dataclassclassOwnObject():prop1:strprop2:int2、将自定义数据类型导出
- 探索AI人工智能领域多智能体系统的技术原理
AI大模型应用之禅
人工智能网络ai
探索AI人工智能领域多智能体系统的技术原理关键词:AI人工智能、多智能体系统、技术原理、智能体交互、分布式计算摘要:本文深入探索了AI人工智能领域多智能体系统的技术原理。首先介绍了多智能体系统的背景,包括其目的、预期读者、文档结构和相关术语。接着阐述了多智能体系统的核心概念与联系,通过文本示意图和Mermaid流程图进行清晰展示。详细讲解了核心算法原理,结合Python源代码进行说明,并给出了相关
- xml解析
小猪猪08
xml
1、DOM解析的步奏
准备工作:
1.创建DocumentBuilderFactory的对象
2.创建DocumentBuilder对象
3.通过DocumentBuilder对象的parse(String fileName)方法解析xml文件
4.通过Document的getElem
- 每个开发人员都需要了解的一个SQL技巧
brotherlamp
linuxlinux视频linux教程linux自学linux资料
对于数据过滤而言CHECK约束已经算是相当不错了。然而它仍存在一些缺陷,比如说它们是应用到表上面的,但有的时候你可能希望指定一条约束,而它只在特定条件下才生效。
使用SQL标准的WITH CHECK OPTION子句就能完成这点,至少Oracle和SQL Server都实现了这个功能。下面是实现方式:
CREATE TABLE books (
id &
- Quartz——CronTrigger触发器
eksliang
quartzCronTrigger
转载请出自出处:http://eksliang.iteye.com/blog/2208295 一.概述
CronTrigger 能够提供比 SimpleTrigger 更有具体实际意义的调度方案,调度规则基于 Cron 表达式,CronTrigger 支持日历相关的重复时间间隔(比如每月第一个周一执行),而不是简单的周期时间间隔。 二.Cron表达式介绍 1)Cron表达式规则表
Quartz
- Informatica基础
18289753290
InformaticaMonitormanagerworkflowDesigner
1.
1)PowerCenter Designer:设计开发环境,定义源及目标数据结构;设计转换规则,生成ETL映射。
2)Workflow Manager:合理地实现复杂的ETL工作流,基于时间,事件的作业调度
3)Workflow Monitor:监控Workflow和Session运行情况,生成日志和报告
4)Repository Manager:
- linux下为程序创建启动和关闭的的sh文件,scrapyd为例
酷的飞上天空
scrapy
对于一些未提供service管理的程序 每次启动和关闭都要加上全部路径,想到可以做一个简单的启动和关闭控制的文件
下面以scrapy启动server为例,文件名为run.sh:
#端口号,根据此端口号确定PID
PORT=6800
#启动命令所在目录
HOME='/home/jmscra/scrapy/'
#查询出监听了PORT端口
- 人--自私与无私
永夜-极光
今天上毛概课,老师提出一个问题--人是自私的还是无私的,根源是什么?
从客观的角度来看,人有自私的行为,也有无私的
- Ubuntu安装NS-3 环境脚本
随便小屋
ubuntu
将附件下载下来之后解压,将解压后的文件ns3environment.sh复制到下载目录下(其实放在哪里都可以,就是为了和我下面的命令相统一)。输入命令:
sudo ./ns3environment.sh >>result
这样系统就自动安装ns3的环境,运行的结果在result文件中,如果提示
com
- 创业的简单感受
aijuans
创业的简单感受
2009年11月9日我进入a公司实习,2012年4月26日,我离开a公司,开始自己的创业之旅。
今天是2012年5月30日,我忽然很想谈谈自己创业一个月的感受。
当初离开边锋时,我就对自己说:“自己选择的路,就是跪着也要把他走完”,我也做好了心理准备,准备迎接一次次的困难。我这次走出来,不管成败
- 如何经营自己的独立人脉
aoyouzi
如何经营自己的独立人脉
独立人脉不是父母、亲戚的人脉,而是自己主动投入构造的人脉圈。“放长线,钓大鱼”,先行投入才能产生后续产出。 现在几乎做所有的事情都需要人脉。以银行柜员为例,需要拉储户,而其本质就是社会人脉,就是社交!很多人都说,人脉我不行,因为我爸不行、我妈不行、我姨不行、我舅不行……我谁谁谁都不行,怎么能建立人脉?我这里说的人脉,是你的独立人脉。 以一个普通的银行柜员
- JSP基础
百合不是茶
jsp注释隐式对象
1,JSP语句的声明
<%! 声明 %> 声明:这个就是提供java代码声明变量、方法等的场所。
表达式 <%= 表达式 %> 这个相当于赋值,可以在页面上显示表达式的结果,
程序代码段/小型指令 <% 程序代码片段 %>
2,JSP的注释
<!-- -->
- web.xml之session-config、mime-mapping
bijian1013
javaweb.xmlservletsession-configmime-mapping
session-config
1.定义:
<session-config>
<session-timeout>20</session-timeout>
</session-config>
2.作用:用于定义整个WEB站点session的有效期限,单位是分钟。
mime-mapping
1.定义:
<mime-m
- 互联网开放平台(1)
Bill_chen
互联网qq新浪微博百度腾讯
现在各互联网公司都推出了自己的开放平台供用户创造自己的应用,互联网的开放技术欣欣向荣,自己总结如下:
1.淘宝开放平台(TOP)
网址:http://open.taobao.com/
依赖淘宝强大的电子商务数据,将淘宝内部业务数据作为API开放出去,同时将外部ISV的应用引入进来。
目前TOP的三条主线:
TOP访问网站:open.taobao.com
ISV后台:my.open.ta
- 【MongoDB学习笔记九】MongoDB索引
bit1129
mongodb
索引
可以在任意列上建立索引
索引的构造和使用与传统关系型数据库几乎一样,适用于Oracle的索引优化技巧也适用于Mongodb
使用索引可以加快查询,但同时会降低修改,插入等的性能
内嵌文档照样可以建立使用索引
测试数据
var p1 = {
"name":"Jack",
"age&q
- JDBC常用API之外的总结
白糖_
jdbc
做JAVA的人玩JDBC肯定已经很熟练了,像DriverManager、Connection、ResultSet、Statement这些基本类大家肯定很常用啦,我不赘述那些诸如注册JDBC驱动、创建连接、获取数据集的API了,在这我介绍一些写框架时常用的API,大家共同学习吧。
ResultSetMetaData获取ResultSet对象的元数据信息
- apache VelocityEngine使用记录
bozch
VelocityEngine
VelocityEngine是一个模板引擎,能够基于模板生成指定的文件代码。
使用方法如下:
VelocityEngine engine = new VelocityEngine();// 定义模板引擎
Properties properties = new Properties();// 模板引擎属
- 编程之美-快速找出故障机器
bylijinnan
编程之美
package beautyOfCoding;
import java.util.Arrays;
public class TheLostID {
/*编程之美
假设一个机器仅存储一个标号为ID的记录,假设机器总量在10亿以下且ID是小于10亿的整数,假设每份数据保存两个备份,这样就有两个机器存储了同样的数据。
1.假设在某个时间得到一个数据文件ID的列表,是
- 关于Java中redirect与forward的区别
chenbowen00
javaservlet
在Servlet中两种实现:
forward方式:request.getRequestDispatcher(“/somePage.jsp”).forward(request, response);
redirect方式:response.sendRedirect(“/somePage.jsp”);
forward是服务器内部重定向,程序收到请求后重新定向到另一个程序,客户机并不知
- [信号与系统]人体最关键的两个信号节点
comsci
系统
如果把人体看做是一个带生物磁场的导体,那么这个导体有两个很重要的节点,第一个在头部,中医的名称叫做 百汇穴, 另外一个节点在腰部,中医的名称叫做 命门
如果要保护自己的脑部磁场不受到外界有害信号的攻击,最简单的
- oracle 存储过程执行权限
daizj
oracle存储过程权限执行者调用者
在数据库系统中存储过程是必不可少的利器,存储过程是预先编译好的为实现一个复杂功能的一段Sql语句集合。它的优点我就不多说了,说一下我碰到的问题吧。我在项目开发的过程中需要用存储过程来实现一个功能,其中涉及到判断一张表是否已经建立,没有建立就由存储过程来建立这张表。
CREATE OR REPLACE PROCEDURE TestProc
IS
fla
- 为mysql数据库建立索引
dengkane
mysql性能索引
前些时候,一位颇高级的程序员居然问我什么叫做索引,令我感到十分的惊奇,我想这绝不会是沧海一粟,因为有成千上万的开发者(可能大部分是使用MySQL的)都没有受过有关数据库的正规培训,尽管他们都为客户做过一些开发,但却对如何为数据库建立适当的索引所知较少,因此我起了写一篇相关文章的念头。 最普通的情况,是为出现在where子句的字段建一个索引。为方便讲述,我们先建立一个如下的表。
- 学习C语言常见误区 如何看懂一个程序 如何掌握一个程序以及几个小题目示例
dcj3sjt126com
c算法
如果看懂一个程序,分三步
1、流程
2、每个语句的功能
3、试数
如何学习一些小算法的程序
尝试自己去编程解决它,大部分人都自己无法解决
如果解决不了就看答案
关键是把答案看懂,这个是要花很大的精力,也是我们学习的重点
看懂之后尝试自己去修改程序,并且知道修改之后程序的不同输出结果的含义
照着答案去敲
调试错误
- centos6.3安装php5.4报错
dcj3sjt126com
centos6
报错内容如下:
Resolving Dependencies
--> Running transaction check
---> Package php54w.x86_64 0:5.4.38-1.w6 will be installed
--> Processing Dependency: php54w-common(x86-64) = 5.4.38-1.w6 for
- JSONP请求
flyer0126
jsonp
使用jsonp不能发起POST请求。
It is not possible to make a JSONP POST request.
JSONP works by creating a <script> tag that executes Javascript from a different domain; it is not pos
- Spring Security(03)——核心类简介
234390216
Authentication
核心类简介
目录
1.1 Authentication
1.2 SecurityContextHolder
1.3 AuthenticationManager和AuthenticationProvider
1.3.1 &nb
- 在CentOS上部署JAVA服务
java--hhf
javajdkcentosJava服务
本文将介绍如何在CentOS上运行Java Web服务,其中将包括如何搭建JAVA运行环境、如何开启端口号、如何使得服务在命令执行窗口关闭后依旧运行
第一步:卸载旧Linux自带的JDK
①查看本机JDK版本
java -version
结果如下
java version "1.6.0"
- oracle、sqlserver、mysql常用函数对比[to_char、to_number、to_date]
ldzyz007
oraclemysqlSQL Server
oracle &n
- 记Protocol Oriented Programming in Swift of WWDC 2015
ningandjin
protocolWWDC 2015Swift2.0
其实最先朋友让我就这个题目写篇文章的时候,我是拒绝的,因为觉得苹果就是在炒冷饭, 把已经流行了数十年的OOP中的“面向接口编程”还拿来讲,看完整个Session之后呢,虽然还是觉得在炒冷饭,但是毕竟还是加了蛋的,有些东西还是值得说说的。
通常谈到面向接口编程,其主要作用是把系统��设计和具体实现分离开,让系统的每个部分都可以在不影响别的部分的情况下,改变自身的具体实现。接口的设计就反映了系统
- 搭建 CentOS 6 服务器(15) - Keepalived、HAProxy、LVS
rensanning
keepalived
(一)Keepalived
(1)安装
# cd /usr/local/src
# wget http://www.keepalived.org/software/keepalived-1.2.15.tar.gz
# tar zxvf keepalived-1.2.15.tar.gz
# cd keepalived-1.2.15
# ./configure
# make &a
- ORACLE数据库SCN和时间的互相转换
tomcat_oracle
oraclesql
SCN(System Change Number 简称 SCN)是当Oracle数据库更新后,由DBMS自动维护去累积递增的一个数字,可以理解成ORACLE数据库的时间戳,从ORACLE 10G开始,提供了函数可以实现SCN和时间进行相互转换;
用途:在进行数据库的还原和利用数据库的闪回功能时,进行SCN和时间的转换就变的非常必要了;
操作方法: 1、通过dbms_f
- Spring MVC 方法注解拦截器
xp9802
spring mvc
应用场景,在方法级别对本次调用进行鉴权,如api接口中有个用户唯一标示accessToken,对于有accessToken的每次请求可以在方法加一个拦截器,获得本次请求的用户,存放到request或者session域。
python中,之前在python flask中可以使用装饰器来对方法进行预处理,进行权限处理
先看一个实例,使用@access_required拦截:
?