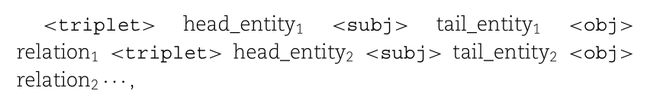【论文笔记】BioGPT: generative pre-trained transformer for biomedical text generation and mining
BioGPT: generative pre-trained transformer for biomedical text generation and mining
论文题目:BioGPT: generative pre-trained transformer for biomedical text generation and mining
论文地址:BioGPT: generative pre-trained transformer for biomedical text generation and mining | Briefings in Bioinformatics | Oxford Academic (oup.com)
代码:microsoft/BioGPT (github.com)
期刊:Briefings in Bioinformatics
Received: June 16, 2022. Revised: August 5, 2022. Accepted: August 23, 2022
摘要
预训练的语言模型在生物医学领域引起了越来越多的关注,这是受其在一般自然语言领域的巨大成功的启发。在自然语言领域的预训练语言模型的两个主要分支,即BERT(及其变体)和GPT(及其变体)中,第一个分支已经在生物医学领域得到了广泛的研究,如BioBERT和PubMedBERT。虽然它们在各种鉴别性的下游生物医学任务上取得了巨大的成功,但由于缺乏生成能力,限制了它们的应用范围。文章提出了BioGPT,一个针对特定领域的生成性Transformer语言模型,在大规模生物医学文献上进行了预训练。文章在六个生物医学自然语言处理任务上评估了BioGPT,并证明文章的模型在大多数任务上都优于以前的模型。在BC5CDR、KD-DTI和DDI端到端关系提取任务上分别得到44.98%、38.42%和40.76%的F1得分,在PubMedQA上得到78.2%的准确率,创造了一个新的记录。关于文本生成的案例研究进一步证明了BioGPT在生物医学文献上的优势。
一、简介
主要有两种预训练语言模型:类似BERT的模型和类似GPT的模型
-
类似BERT的模型:主要用于序列分类和序列标注
-
类似GPT的模型:主要用于生成式任务,如摘要生成、知识三元组生成
直接将通用领域nlp模型应用到生物领域会导致模型性能下降,因此常常训练一个针对于生物领域的预训练模型,如BioBERT和PubMedBERT。然而,以前的工作主要集中在BERT模型上,它更适合于理解任务,而不是生成任务。相比之下,GPT模型在生成任务上显示了它们的能力,但在直接应用于生物医学领域时,表现出较差的性能。
BioGPT遵循Transformer语言模型主干,并在15M PubMed摘要上从头开始预训练。
作者将BioGPT应用于六个生物医学NLP任务:BC5CDR、KD-DTI和DDI的端到端关系抽取,在PubMedQA上的问答,在HoC上的文档分类,以及文本生成。
为了适应下游任务,作者精心设计并分析了目标序列格式和提示,以更好地建模任务。
二、相关工作
在生物领域的预训练模型
-
BioBERT
-
BLUE benchmark【Transfer learning in biomedical natural language processing: An evaluation of BERT and ELMo on ten benchmarking datasets】论文中模型,在BERT的基础上再对PubMed文本和MIMIC-III的临床笔记进行预训练。
-
SciBERT【SciBERT: A pretrained language model for scientific text】没有采用在BERT的基础上进一步预训练,而是在大型科学文献(主要是生物和计算机文献)上从头训练。
-
PubMedBERT【Domain-specific language model pretraining for biomedical natural language processing】直接在14M PubMed 摘要上预训练,证明了在单词表更适合生物医学领域的特定领域数据上从头开始进行预训练是一个更好的策略。
所有这些工作都表明,与原始的BERT相比,在大量生物医学文献语言处理任务上有了改进,而没有一个是用于生物医学生成任务。
下游任务
6个下游任务都可以被表述为文本生成/挖掘任务。
Relation extraction 关系抽取
经典的基于管道的方法[23,33,34]将任务分解为几个独立的子任务,这些子任务需要额外的中间注释和信息,这些信息可能会遭受缺乏中间注释数据和错误积累的影响。
联合提取方法的目的是从文本中联合提取实体和它们之间的关系
序列标注方法通过对文本中的单词标注不同的标签来解决这一任务,以标注出所有提到的实体,然后通过分类器进行它们之间的关系分类[35-38]
表格填充方法将任务制定为由自身的笛卡尔积构成的表格,并预测标注对之间的关系[39-41]
这些方法可能会受到以前的标签过程和费力的中间标注(即命名实体识别)造成的错误积累的影响。
但是,许多联合抽取方法仍然需要额外的实体信息,本工作中,作者专注于端到端的关系抽取,将任务制定为文本生成任务,只将文本作为输入,并以端到端的方式生成关系三元组,而没有额外的中间标注。
Question answering 问答
典型方法是预测原文的一个片段作为答案,或者为较简单的任务预测一个标签(例如,是或不是),并预先定义分类答案。
Document classification 文档分类
文件分类是将文件归入预定的类别(单标签或多标签)
三、预训练方法
从数据集、词表和模型三个角度描述BioGPT
Dataset
论文【Domain-specific language model pretraining for biomedical natural language processing】中指出对于特定领域,在领域数据上从零训练至关重要。作者只考虑了领域内数据,具体而言是PubMed网站上的2021年前的数据。
Vocabulary
论文【Domain-specific language model pretraining for biomedical natural language processing】也指出,域内单词表是至关重要的,作者在收集的域内语料库中学习单词表。
Model
GPT-2作为backbone
实际上,作者采用 G P T - 2 m e d i u m GPT\text{-}2_{medium} GPT-2medium作为主干网络,它有24层,1024个隐藏层大小和16个注意力头,总共有355M个参数,而BioGPT有347M个参数(差异只来自于不同的单词表量所导致的不同的嵌入大小和输出投影大小
Training criteria
让 D = { x i } i D=\{x_{i}\}_{i} D={xi}i代表序列的集合,序列 x i x_{i} xi是由 n i n_{i} ni个tokens构成的,即 x i = ( s 1 , s 2 , . . . s n i ) x_{i}=(s_1,s_2,...s_{n_{i}}) xi=(s1,s2,...sni),训练目标就是使得负样本对数似然函数最小。
m i n − 1 ∣ D ∣ ∑ i = 1 ∣ D ∣ ∑ j = 1 n i log P ( s j ∣ s j − 1 , s j − 2 , ⋯ , s 1 ) . \mathrm{min~}-\frac1{|\mathcal{D}|}\sum_{i=1}^{|\mathcal{D}|}\sum_{j=1}^{n_i}\log P(s_j|s_{j-1},s_{j-2},\cdots,s_1). min −∣D∣1i=1∑∣D∣j=1∑nilogP(sj∣sj−1,sj−2,⋯,s1).
四、微调方法
本节内容介绍如何将预训练的BioGPT适应于下游任务:端到端关系抽取,问答以及文档分类。这些任务的输入都是序列,但是它们有着不同的输出格式。为了将BioGPT用于这些任务,需要将标签转换为序列。
作者将标签转换为自然语言的序列,而不是使用其他工作中探讨的特殊标注的结构化格式
端到端关系抽取
找到文本中的所有三元组<头实体,尾实体,关系>,例子包括提取药物-靶点相互作用、化学-疾病-关系和药物-药物相互作用。
作者为三元组设计了三种简单自然语言序列样式:
- ‘subject verb object’ (svo) ,分别对应头实体,关系,尾实体
- ‘subject is the rel.noun of object’ (is-of) ,rel.noun 是关系的名词形式
- ‘the relation between subject and object is rel.noun’(rel-is)
如果一个输入文件有多个关系三元组,根据它们在文件中出现的顺序进行排序,并使用分号将它们串联起来。
自然语言处理形式的句子可以用正则表达式转换回三元组,用户还可以根据任务自定义格式。
问答
任务描述:给出一个问题,一个参考背景和一个答案,目标是确定是否可以从参考背景中推理出问题的答案。标签属于是、不是或可能的范畴。
具体格式:
- source: question: question text. context: context text. answer: answer text.
- target: the answer to the question given the context is yes.
文档分类
给定一个文档文本,目标是对该文档的类型进行分类。
目标序列使用‘the type of this document is label’格式。
基于提示的微调
GPT-3使用**硬提示(手动设计的离散语言短语)**来为不同的任务生成。虽然硬提示可以达到令人满意的性能,但设计特定任务的提示是很费力的,而且人们发现,不同的提示会导致不同的性能。
作者采用软提示:利用连续嵌入(虚拟token)来引导预训练语言模型,直接在文本前附加几个虚拟token作为提示。这种连续嵌入是随机初始化的,并在下游任务上进行端到端的学习,以达到特定任务的效果。软提示没有直接加在source input的前面,而是在source和target之间。
问题:图片左部training部分为什么输入的序列尾部也有target?
五、实验部分
在四个任务上的六个数据集上进行了评估:
-
end-to-end relation extraction:BC5CDR、KD-DTI、DDI
-
question answering: PubMedQA
-
document classificaiton: HOC
-
text generation: self-created dataset
G P T - 2 m e d i u m GPT\text{-}2_{medium} GPT-2medium作为模型骨干backbone
使用BPE来学习token并构建词表,而不是使用 G P T - 2 GPT\text{-}2 GPT-2的词表
8个NVIDIA V100 GPU上预训练
在单个NVIDIA V100 GPU上进行微调实验
在推理过程中,对于文本生成任务采用==beam search== 策略,beam=5,而对于其他任务采用==greedy search==策略。
端到端关系抽取
模型将文本作为输入并直接生成三元组,主要与REBEL(一种基于seq2seq模型的端到端三元组提取方法)作比较。
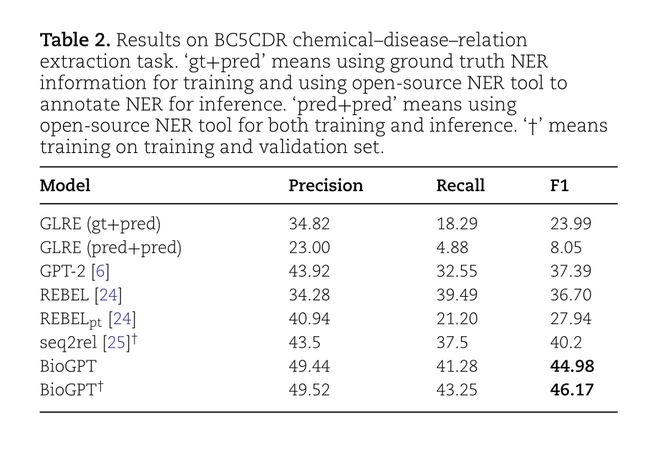
BC5CDR
BC5CDR:chemical-disease-relation extraction
R E B E L p t REBEL_{pt} REBELpt是对REBEL模型的增强,在额外的从维基百科创建的大型关系三元组数据集上进行了预训练。
seq2rel也是一种端到端关系抽取方法,并且是在训练和验证机上训练的,而在训练和验证集上训练的BioGPT记为 B i o G P T p t BioGPT_{pt} BioGPTpt
GLRE是一种采用pipeline方式的关系提取模型,该方法需要NER信息作为中间标注。(gt+pred)代表训练时需要的实体信息是真实的,而在推理时使用开源的NER工具生成;而(pred+pred)代表训练和推理时都采用开源的NER工具生成。
有两个发现
- 基于pipeline的方法GLRE在使用开源工具标记的NER而不是ground truth的NER时明显下降,这很符合实际情况
- 与REBEL相比,BioGPT有很大的提高,提高了8.28%
KD-DTI
KD-DTI: drug-target-interaction
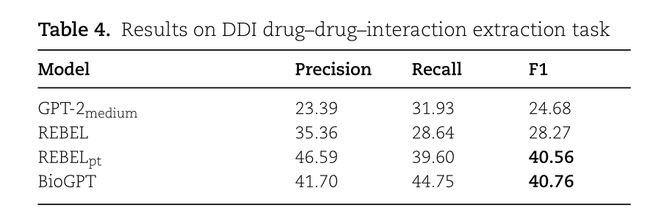
DDI
DDI:drug-drug-interaction
问答
PubMedQA,任务目标是给出yes/no/maybe
只评估了分类的准确性
文档分类
HoC(Hallmarks of Cancers语料库)
文本生成
作者还对预训练的BioGPT在生物医学领域的文本生成能力,以及一般领域的GPT-2在生物医学领域的表现如何感到好奇。
作者从KD-DTI测试集中提取三元组中的所有实体,然后对于每个药物/目标名称,将其作为前缀提供给语言模型,让模型以其为条件生成文本。然后,调查所生成的文本是否有意义和流畅。
- 给予相对常见的名称作为输入,GPT-2可以生成与该词和生物医学有关的有意义的和流畅的文本,而BioGPT则生成更具体和专业的描述。
- 当给出一些不常见的名字时,GPT2不能生成有意义的描述,而BioGPT仍然能生成具体的描述。
- 给定非常不常见或者特定领域名称,GPT-2不能产生任何信息性的文本,BioGPT仍能生成相关度高的描述
另外也输入了一些感兴趣的关键词来生成文本,并对GPT-2和BioGPT做对比,BioGPT也是表现更好。
总的来说,在各种生物医学NLP任务中,对域内生物医学文献从头开始进行预训练的BioGPT比一般域的GPT-2表现更好,并且在各自的任务中比以前的大多数方法表现更好,在六个任务中的四个任务中达到最先进水平。
六、消融实验
对==标签的提示设计和目标序列格式==进行了消融研究。
Target sequence format目标序列格式
之前的采用结构化表示
而在BioGPT中使用了统一的模块来编码上下文并生成答案。从直觉上讲,保持输入和答案之间的格式一致性会更好。
通过比较,自然语言的格式比结构化格式更好。所有格式中,rel-is格式在F1方面表现最好,提供了一个语义上更流畅和清晰的描述。
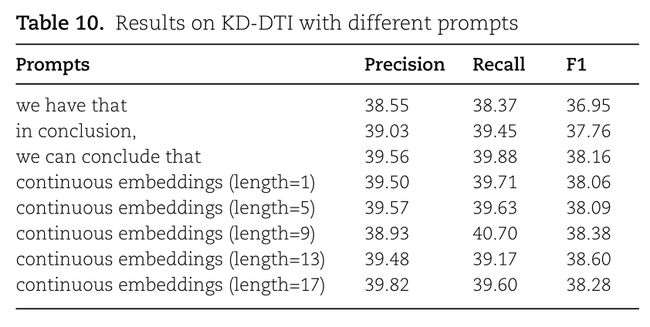
Prompt design提示设计
在KD-DTI提取任务上进行了人工设计的硬提示和连续嵌入的软提示的对比实验。表现最好的是长度为13个虚拟token的连续嵌入。
- 不同的人工设计的硬提示导致不同的性能,更有指导意义和信息丰富的提示会获得更好的性能
- 连续嵌入的软提示比硬提示更好
- 软提示的性能与长度大致无关
总结
作者提出了BioGPT,采用GPT-2作为backbone,在15M PubMed语料库上进行了从头开始的预训练。
作者仔细设计并对比了用于下游任务的提示和目标序列格式。
作者将BioGPT用于端到端关系抽取任务、问答任务、文档分类任务和文本生成任务,总体而言,BioGPT性能更优。
相关论文
-
[1906.05474] Transfer Learning in Biomedical Natural Language Processing: An Evaluation of BERT and ELMo on Ten Benchmarking Datasets (arxiv.org)
-
Domain-Specific Language Model Pretraining for Biomedical Natural Language Processing | ACM Transactions on Computing for Healthcare
-
[1903.10676] SciBERT: A Pretrained Language Model for Scientific Text (arxiv.org)
-
[2010.13415v1] TPLinker: Single-stage Joint Extraction of Entities and Relations Through Token Pair Linking (arxiv.org)
-
GraphRel: Modeling Text as Relational Graphs for Joint Entity and Relation Extraction - ACL Anthology
ngle-stage Joint Extraction of Entities and Relations Through Token Pair Linking (arxiv.org)](https://arxiv.org/abs/2010.13415v1) -
GraphRel: Modeling Text as Relational Graphs for Joint Entity and Relation Extraction - ACL Anthology