基于html 和javascript搭建基因组浏览器 ---Jbrowse(附代码,并说清楚踩坑之处)
由于自己的项目缺少一部分可以展示的基因组浏览器,看到很多网站都用了jbrowse这个浏览器,所以我也跟着学习了一下,并放在了自己的项目里。
下面说下学习方法:
!!!禁止直接搬运,违者必究
我是根据官方文档学习的:
https://jbrowse.org/jb2/docs/tutorials/embed_linear_genome_view/01_introduction/
1、安装jbrowse 我用的npm安装的
#在终端安装
npm install -g @jbrowse/cli
#安装之后,在终端输入下面这行命令,看看是否会出现帮助信息,如果出现,则代表安装成功
jbrowse --help
2、创建index.html
<html>
<head>
<script
src="//unpkg.com/react@16/umd/react.development.js"
crossorigin
>script>
<script
src="//unpkg.com/react-dom@16/umd/react-dom.development.js"
crossorigin
>script>
<script
src="//unpkg.com/@jbrowse/react-linear-genome-view/dist/react-linear-genome-view.umd.development.js"
crossorigin
>script>
head>
<body>
<h1>We're using JBrowse Linear Genome View!h1>
<div id="jbrowse_linear_genome_view">div>
body>
html>
3、创建js,并在index.html页面中引入
可以通过jbrowse add-assembly,jbrowse add-track 加入数据,再将生成的由这两个命令生成的config.json文件,复制出assembly和tracks,也可以自己直接写js。
assembly.js
export default {
name: "GRCm39",
sequence: {
type: "ReferenceSequenceTrack",
trackId: "GRCm39-ReferenceSequenceTrack",
adapter: {
type: "BgzipFastaAdapter",
fastaLocation: {
//下面所需要的数据,我是在ensembl网站下载到本地的,这部分是参考基因组的数据,不用处理分析,直接放入项目即可(下面附数据下载链接)
uri: "./Mus_musculus.GRCm38.dna.toplevel.fa.gz",
locationType: "UriLocation"
},
faiLocation: {
uri: "./Mus_musculus.GRCm38.dna.toplevel.fa.gz.fai",
locationType: "UriLocation"
},
gziLocation: {
uri: "./Mus_musculus.GRCm38.dna.toplevel.fa.gz.gzi",
locationType: "UriLocation"
}
}
},
aliases: ["mm10"],
refNameAliases: {
adapter: {
type: 'RefNameAliasAdapter',
location: {
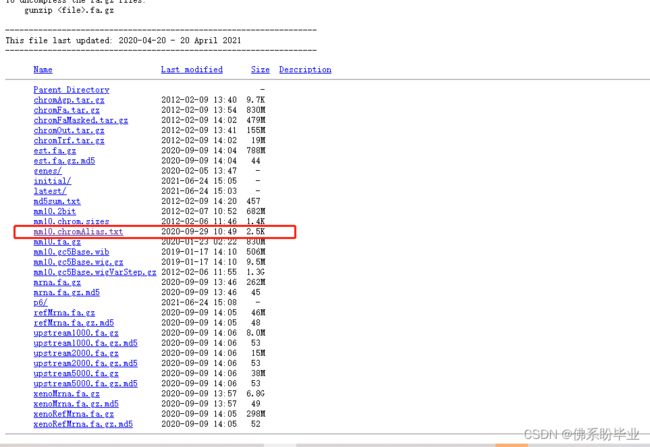
uri: './mm10.chromAlias.txt',
locationType: 'UriLocation',
},
},
},
}
assembly.js中所需要的参考基因组与参考基因组索引等信息下载地址:
refNameAliases 里面的mm10.chromAlias.txt 别名文件,下载地址为:
https://hgdownload.soe.ucsc.edu/downloads.html#mouse
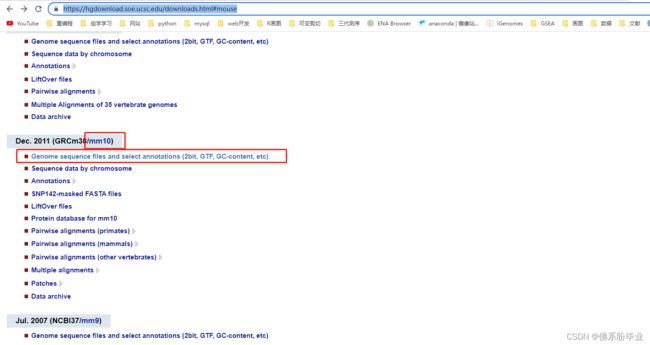
(我这里测试用的GRCm38的数据)
我是在ensembl网站下载到本地的,这部分是参考基因组的数据,不用处理分析,直接放入项目即可(下面附下载链接)
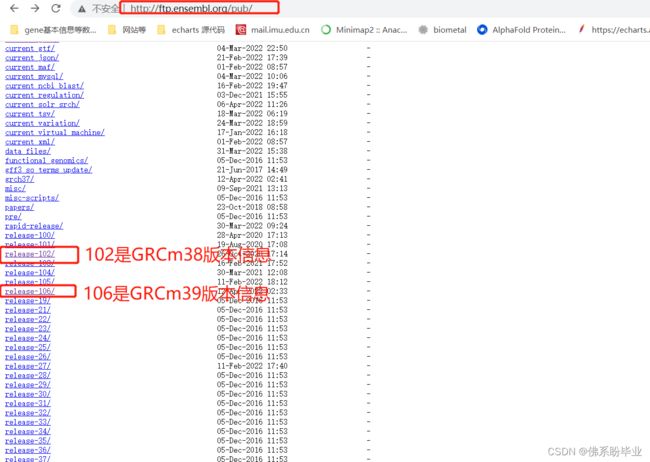
http://ftp.ensembl.org/pub/release-102/fasta/mus_musculus/dna_index/
tracks.js
export default [
{
type: "FeatureTrack",
// trackId: "volvox4.sorted.gff3",
trackId: "volvox1.sorted.gff3",
name: "NCBI RefSeq Genes",
adapter: {
type: "Gff3TabixAdapter",
gffGzLocation: {
//下面的uri里面的数据是我下载的Mus_musculus.GRCm38.102.gff3.gz并分析后的数据,这里面的数据要用bgzip排序,并给排序之后的gff文件创建tbi索引,才可以顺利展示,不然不处理会直接报错(最好把数据放在本地项目中)
uri: "./volvox1.sorted.gff3.gz",
locationType: "UriLocation"
},
index: {
location: {
uri: "./volvox1.sorted.gff3.gz.tbi",
locationType: "UriLocation"
},
indexType: "TBI"
},
},
category: [ "Genes"],
assemblyNames: ["GRCm39"]
}
]
gff数据下载链接:
http://ftp.ensembl.org/pub/release-102/gff3/mus_musculus/
gff数据处理流程(在linux处理):
1)将下载下来的Mus_musculus.GRCm38.102.gff3.gz解压
gunzip Mus_musculus.GRCm38.102.gff3.gz
2)解压之后,对其进行GUN排序:
(grep ^"#" Mus_musculus.GRCm38.102.gff3; grep -v ^"#" data/volvox.gff3 | grep -v "^$" | grep "\t" | sort -k1,1 -k4,4n) > volvox1.sorted.gff3
3)对排序之后的sorted.gff3文件,用bgzip压缩,得到volvox1.sorted.gff3.gz 数据:
bgzip volvox1.sorted.gff3 (volvox1.sorted.gff3.gz,这个数据,即为tracks里面我们所需要的gff3.gz文件,不要放错,如果放成Mus_musculus.GRCm38.102.gff3这个数据,会报错)
4)tabix给gff3文件,建索引,得到TBI文件
#安装
conda install -c bioconda tabix
#创建索引:
tabix -p gff volvox1.sorted.gff3.gz
处理之后,将得到的volvox1.sorted.gff3.gz 和 volvox1.sorted.gff3.gz.tbi数据下载到本地,并放入自己的项目中,并在tracks.js中放入(路径要对)
4、将配置好的assembly.js 和 tracks.js在index.html页面中引入,下面为完整html页面:
<html>
<head>
<title>jbrowsetitle>
<script src="//unpkg.com/react@16/umd/react.development.js" crossorigin>script>
<script src="//unpkg.com/react-dom@16/umd/react-dom.development.js" crossorigin >script>
<script src="//unpkg.com/@jbrowse/react-linear-genome-view/dist/react-linear-genome-view.umd.development.js" crossorigin>script>
head>
<body>
<div id="jbrowse_linear_genome_view">div>
<script type="module">
import assembly from './assembly.js' #在这里引入
import tracks from './tracks.js' #在这里引入
const { createViewState, JBrowseLinearGenomeView } = JBrowseReactLinearGenomeView
console.log('JBrowseReactLinearGenomeView',JBrowseReactLinearGenomeView)
const { createElement } = React
const { render } = ReactDOM
const state = new createViewState({
assembly,
tracks,
location: 'X:161,202,097..161,209,195', //这个染色体的位置信息,不能写错,还有逗号,是从后往前数三位,标一个逗号,中间的点为两个
})
render(
createElement(JBrowseLinearGenomeView, { viewState: state }),
document.getElementById('jbrowse_linear_genome_view'),
)
script>
body>
html>
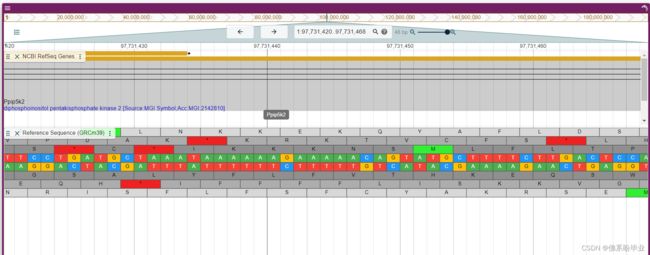
5、之后进入html页面,就可以看到基因组浏览器展示信息的页面了